Поиск особенностей структуры хроматина
Александра Галицына
Цикл лекций о структуре хроматина
10 декабря 2020
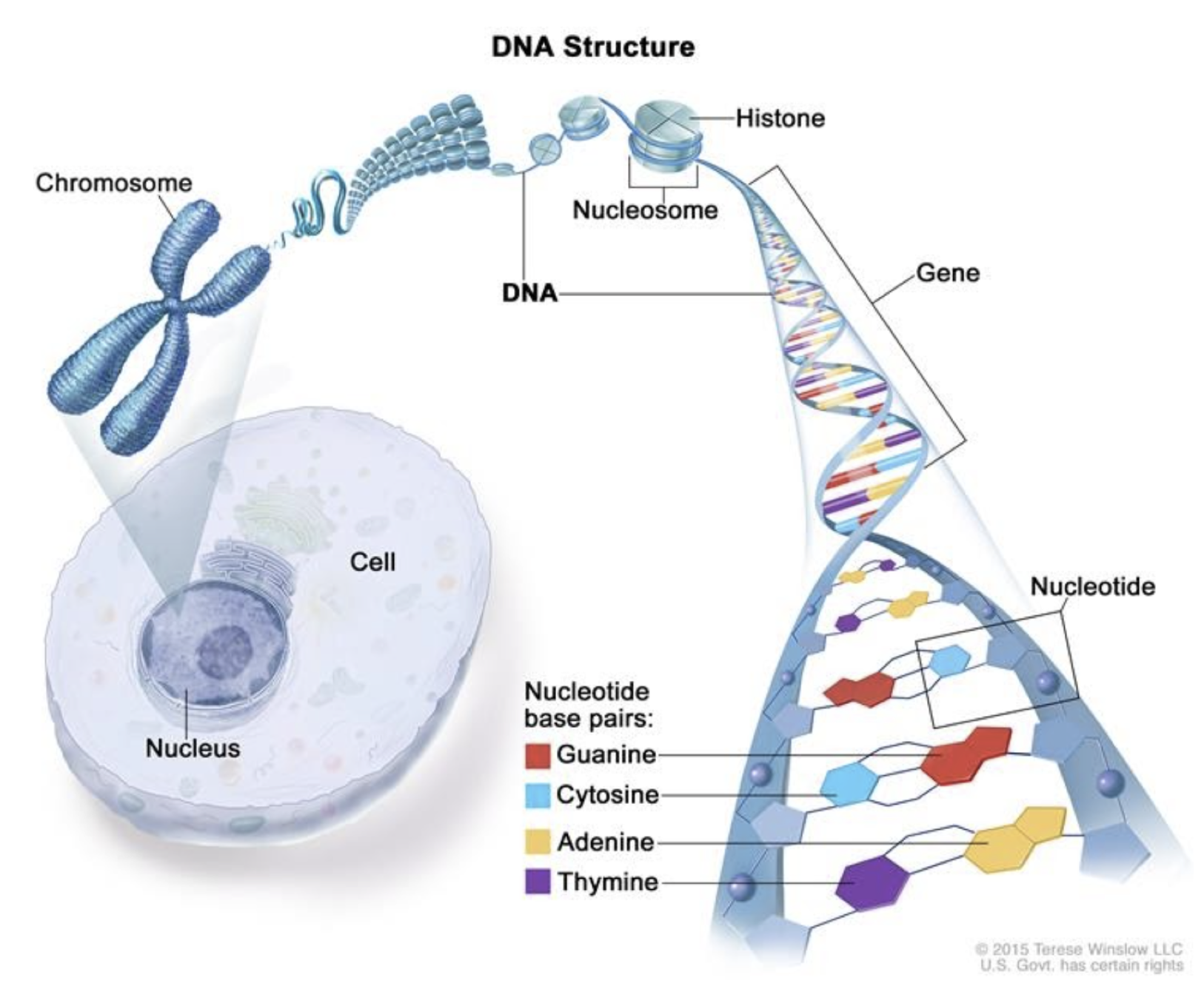
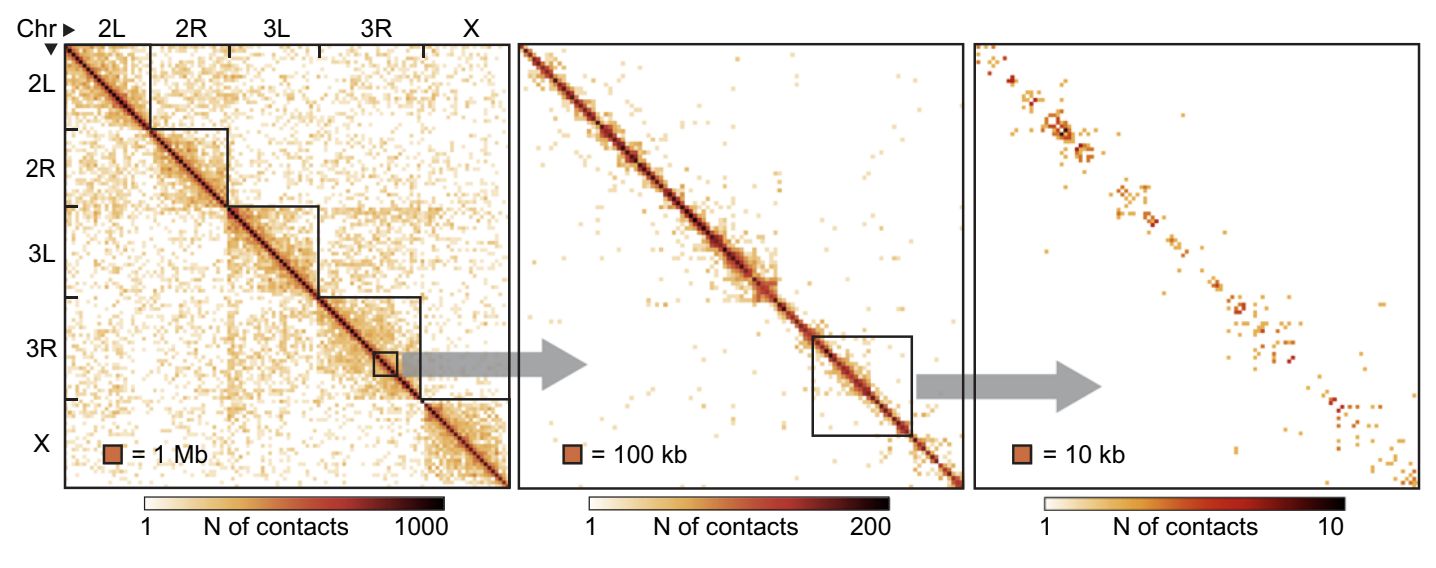
Hi-C - это МНОГО данных
Немного простой математики
длина ДНК клетки человека:
~2 м
National Human Genome Research Institute www.genome.gov
Hi-C - это МНОГО данных
Немного простой математики
длина ДНК клетки человека:
~2 м
длина генома человека:
National Human Genome Research Institute www.genome.gov
~1 м
Hi-C - это МНОГО данных
Немного простой математики
длина ДНК клетки человека:
размер генома человека:
количество всевозможных попарных контактов ДНК:
в 100Kb-бинах
в нуклеотидах
длина ДНК клетки человека:
~2 м
длина генома человека:
~1 м
размер одного нуклеотида = 0.34 нм = 0.00000000034 м
~3000000000 нт
3000000000·3000000000 =
9000000000000000000 нт
компьютерное представление:
~10 Эксабайт
(19 нулей)
Hi-C - это МНОГО данных
Немного простой математики
длина ДНК клетки человека:
длина ДНК клетки человека:
~2 м
длина генома человека:
~1 м
~ 1 Гигабайт
размер генома человека:
количество всевозможных попарных контактов ДНК:
в 100Kb-бинах
в нуклеотидах
~3000000000 нт
30000·30000 =
900000000 нт
компьютерное представление:
~10 Эксабайт
(19 нулей)
~30000 бинов
3000000000·3000000000 =
9000000000000000000 нт
Подходы для хранения Hi-C
Варианты хранения:
- плотная матрица
- разреженная матрица

игрушечный пример:
[[2,0,1], [0,2,0], [1,0,2]]
left right n_contacts 1 1 2 1 3 1 2 2 2 3 3 2
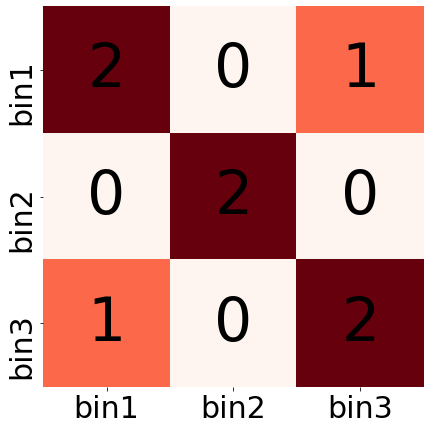
Подходы для хранения Hi-C
Варианты хранения:
- плотная матрица
- разреженная матрица .cool файл

игрушечный пример:
[[2,0,1], [0,2,0], [1,0,2]]
left right n_contacts 1 1 2 1 3 1 2 2 2 3 3 2


Подходы для обработки данных о взаимодействии ДНК
- Выбрать только заведомо важные контакты

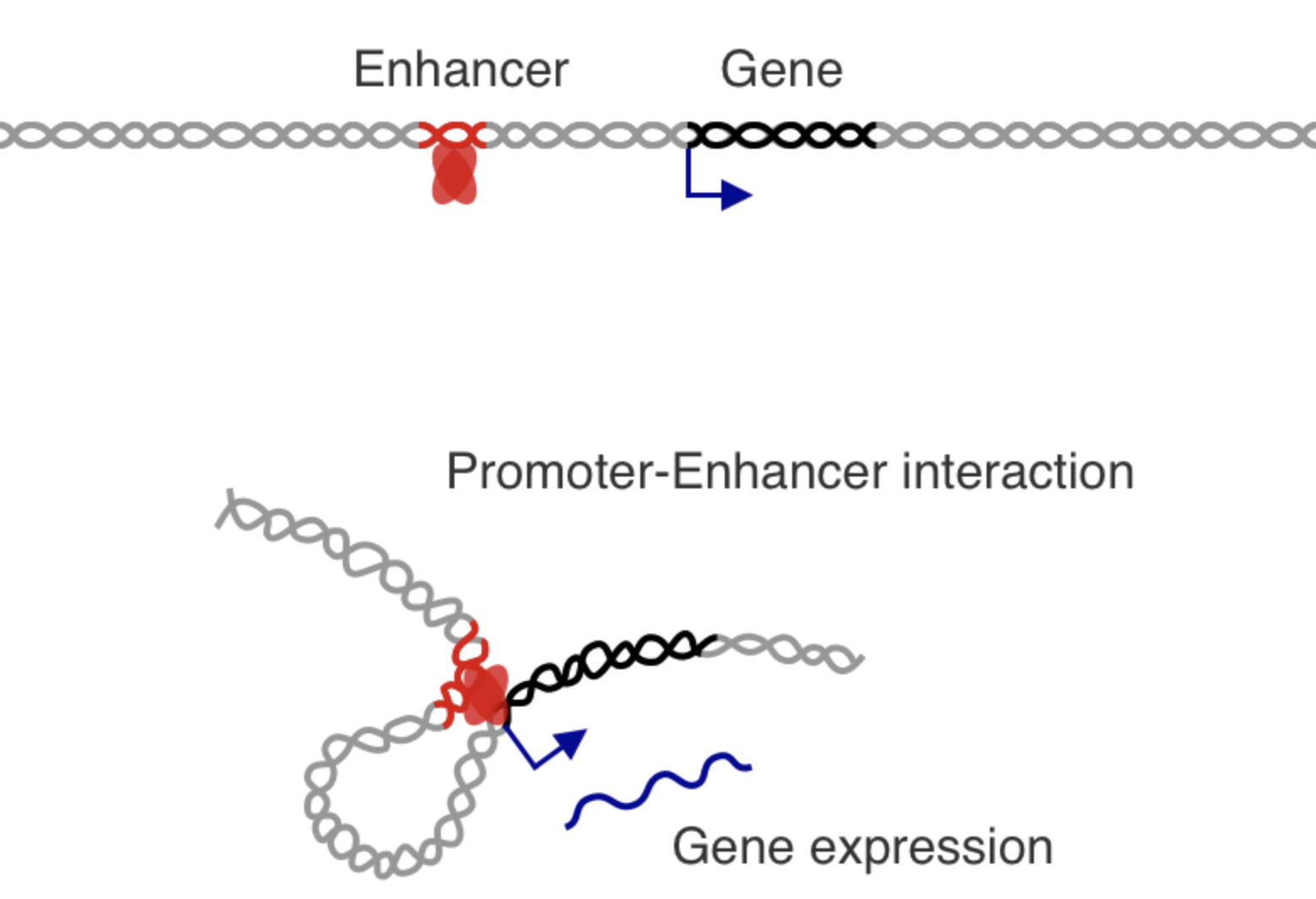
+
- точечные контакты промоторов и энхансеров
Подходы для обработки данных о взаимодействии ДНК
- Выбрать только заведомо важные контакты

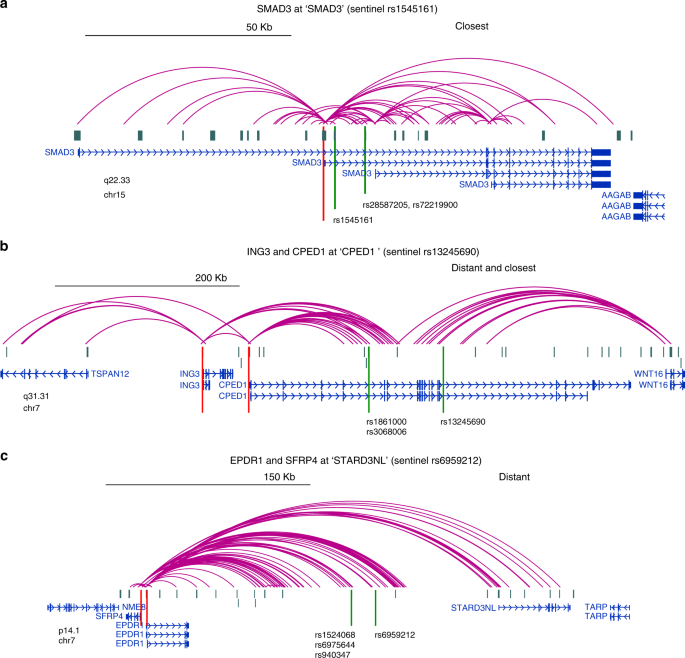
Chesi et al. 2019
- точечные контакты промоторов и энхансеров
- на уровне экспериментальной техники: Capture C
Подходы для обработки данных о взаимодействии ДНК
- Выбрать только заведомо важные контакты
2. Анализировать особенности (фичи, features) данных

- точечные контакты промоторов и энхансеров
- на уровне экспериментальной техники: Capture C



}
Особенности структуры хроматина
Fraser et al. Microbiology and Molecular Biology Reviews 2015
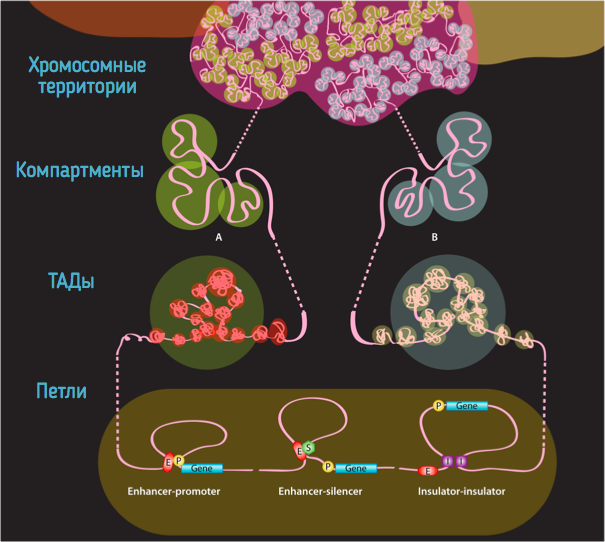
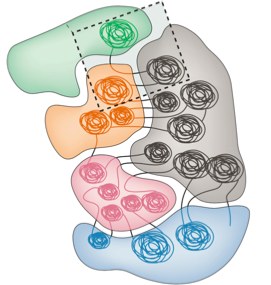
Хромосомные территории
Bonev et al. 2016 Nature Reviews
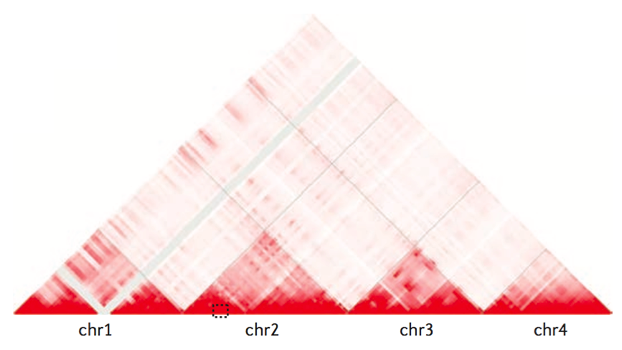
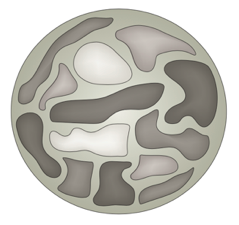
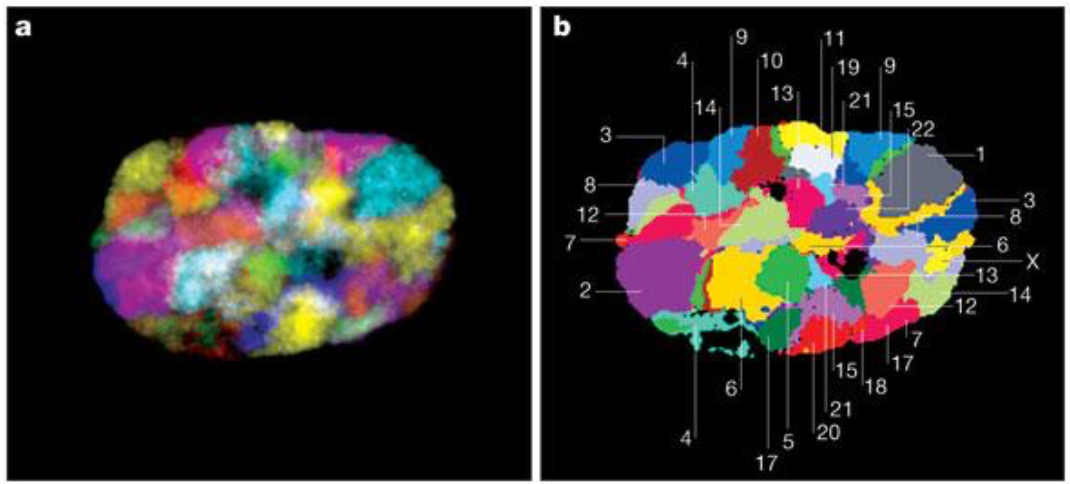
Хромосомные территории
Falk et al. Nature 2019
^^^ Сделали усреднение ^^^
получили средние контакты между парами хромосомам
Hi-C карты для трех хромосом:
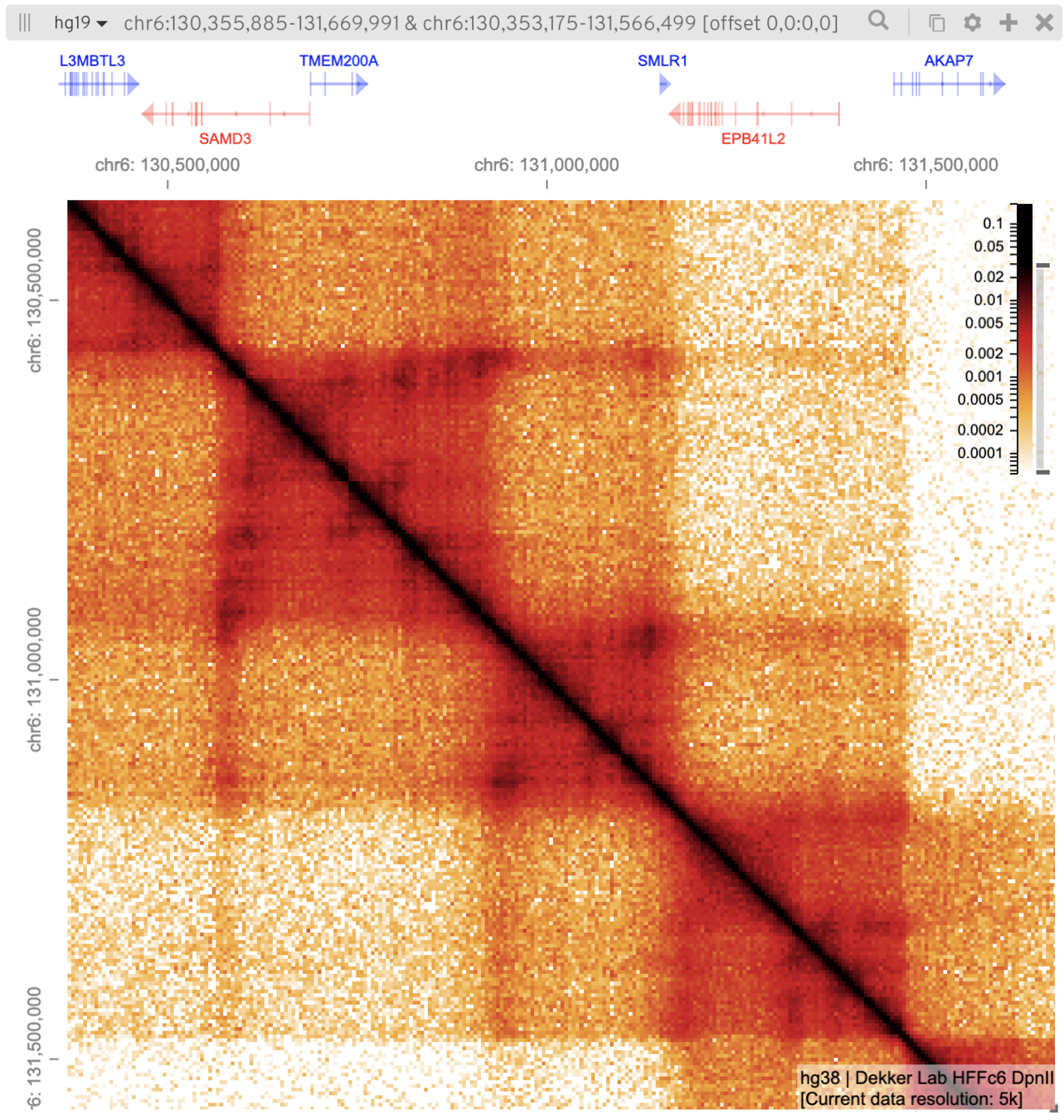
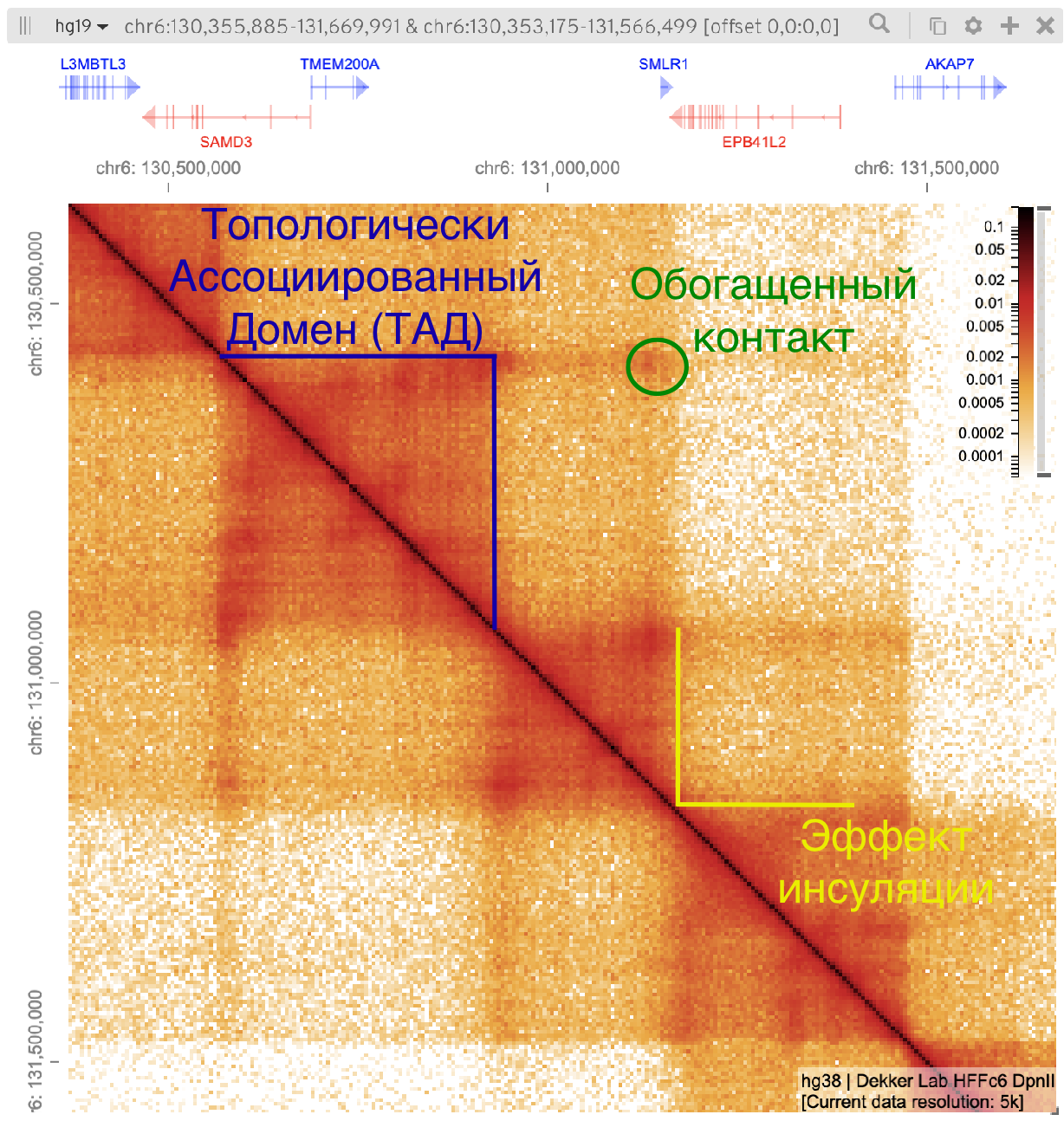
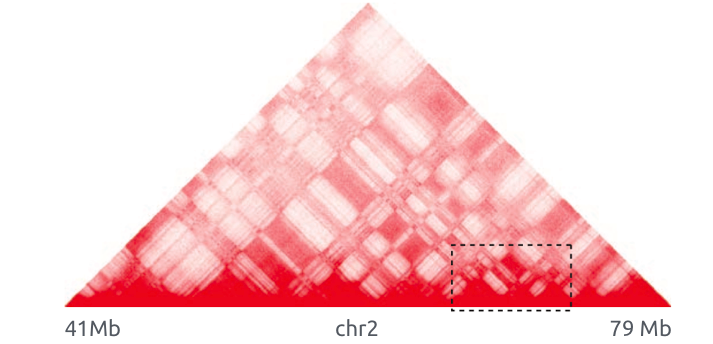
Топологически ассоциированные домены (ТАДы)

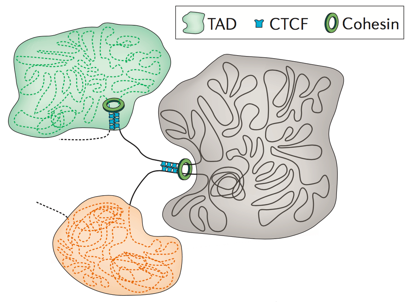
Bonev et al. 2016 Nature Reviews
одна из гипотетических моделей:
ТАДы сложнее
Filippova et al. Algorithms for Molecular Biology 2014
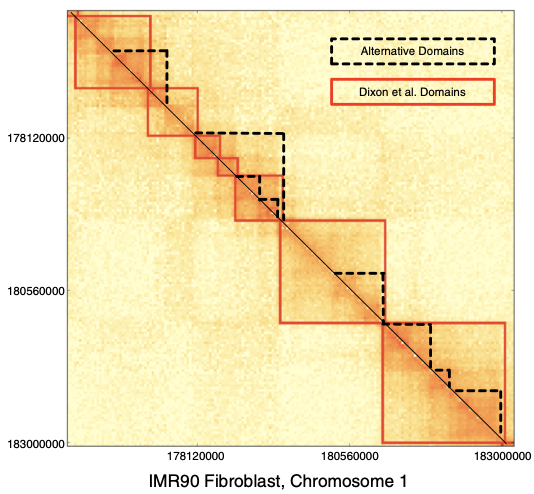
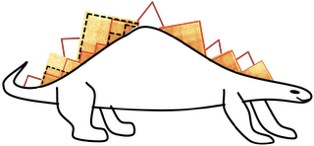
чем "квадраты" вдоль диагонали:
- Решается с помощью динамического программирования, например,
Armatus
Много способов искать ТАДы...
Forcato et al. Nature Methods 2017
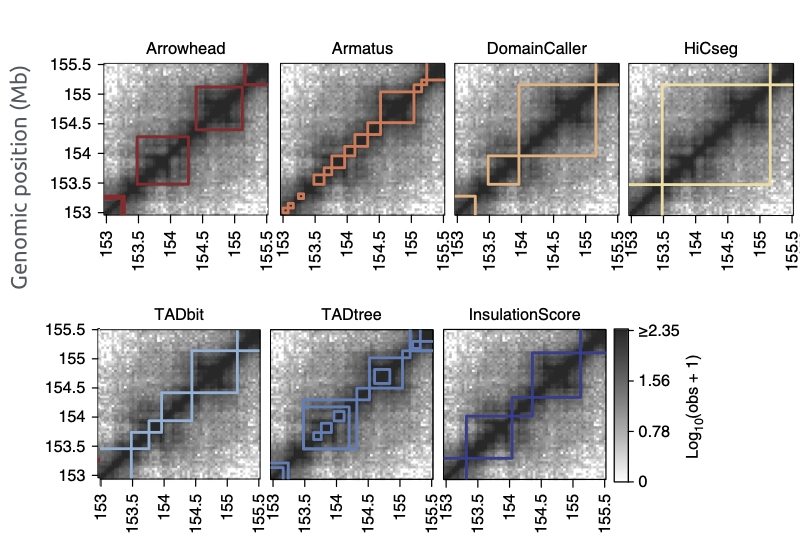
Разберем пример поиска ТАДов
based on Crane, 2015
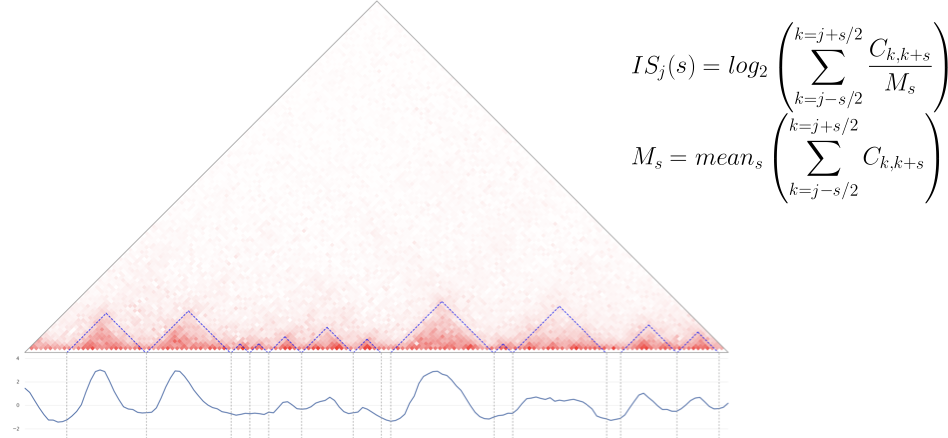
- Показатель инсуляции - один из простейших подходов:
- Подсчитать меру инсуляции для каждого бина,
- Найти локальные минимумы:
Может, ТАДы и не стоит искать?
- Использование меры Gamma transitional (близкая к IS)
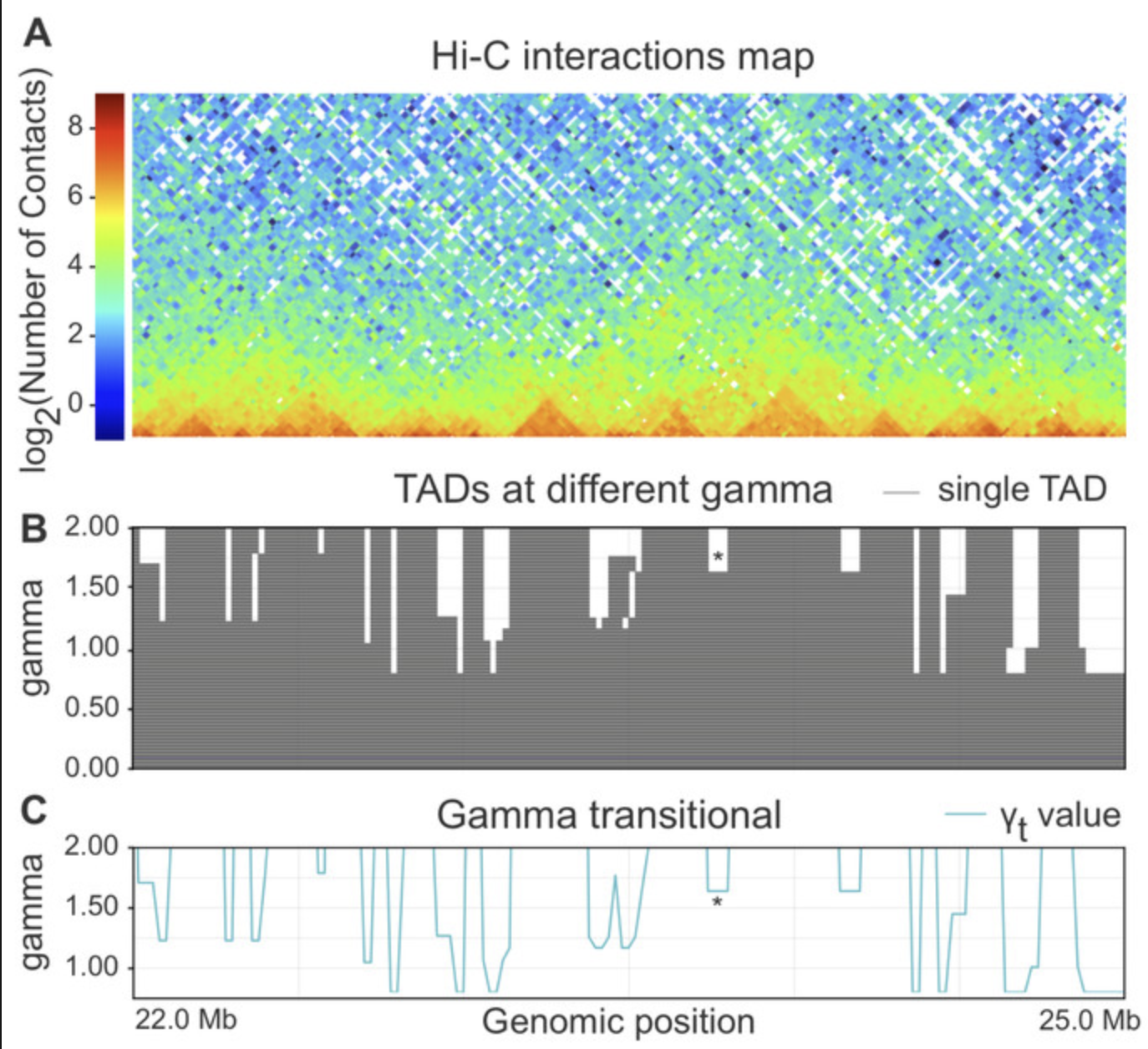
1. для каждого бина генома получаем
некоторую меру инсуляции
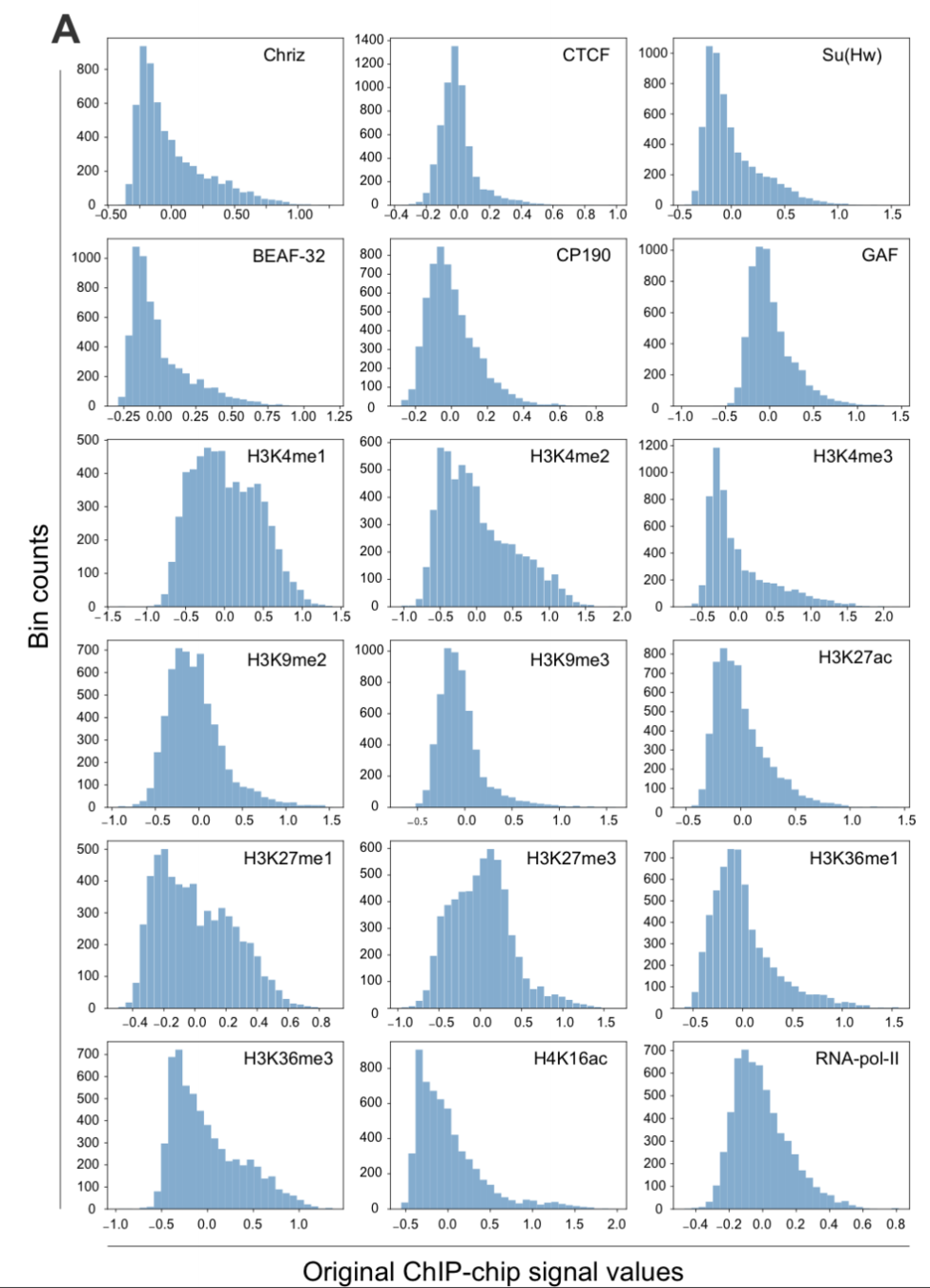
2. берем набор эпигенетических данных
Может, ТАДы и не стоит искать?
- Использование меры Gamma transitional (близкая к IS)
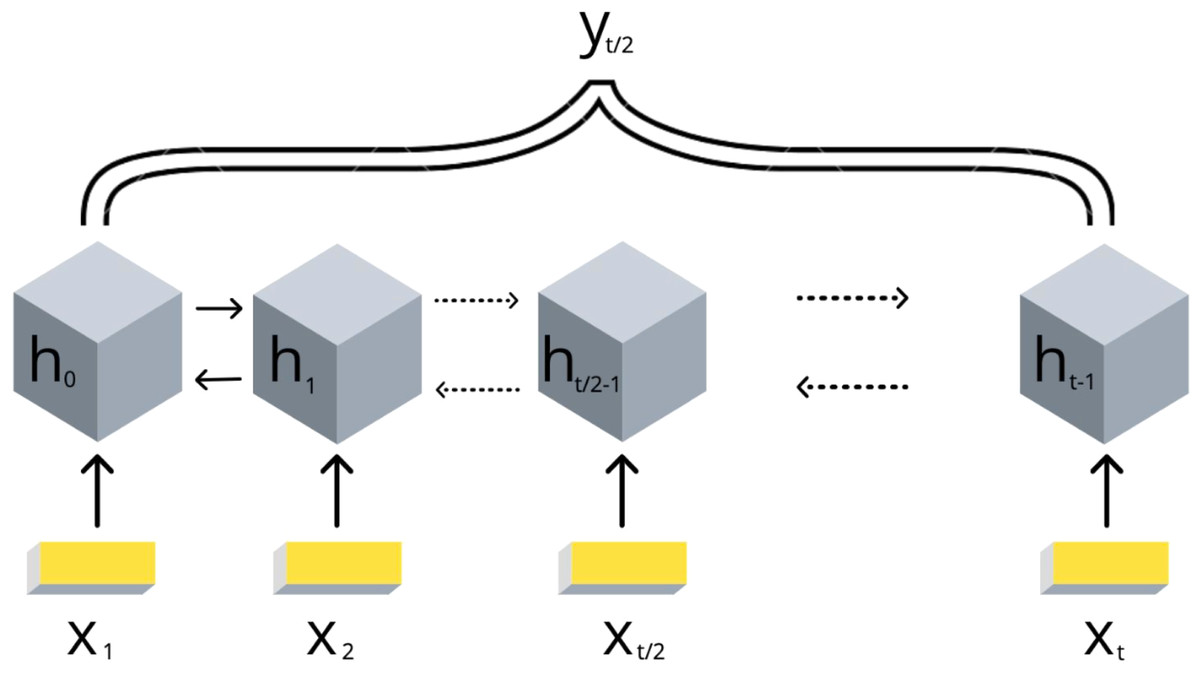
3. подбираем и обучаем модель машинного обучения
(в данном случае biLSTM):
4. получаем ранжирование эпигенетических факторов по важности для формирования структуры хроматина:
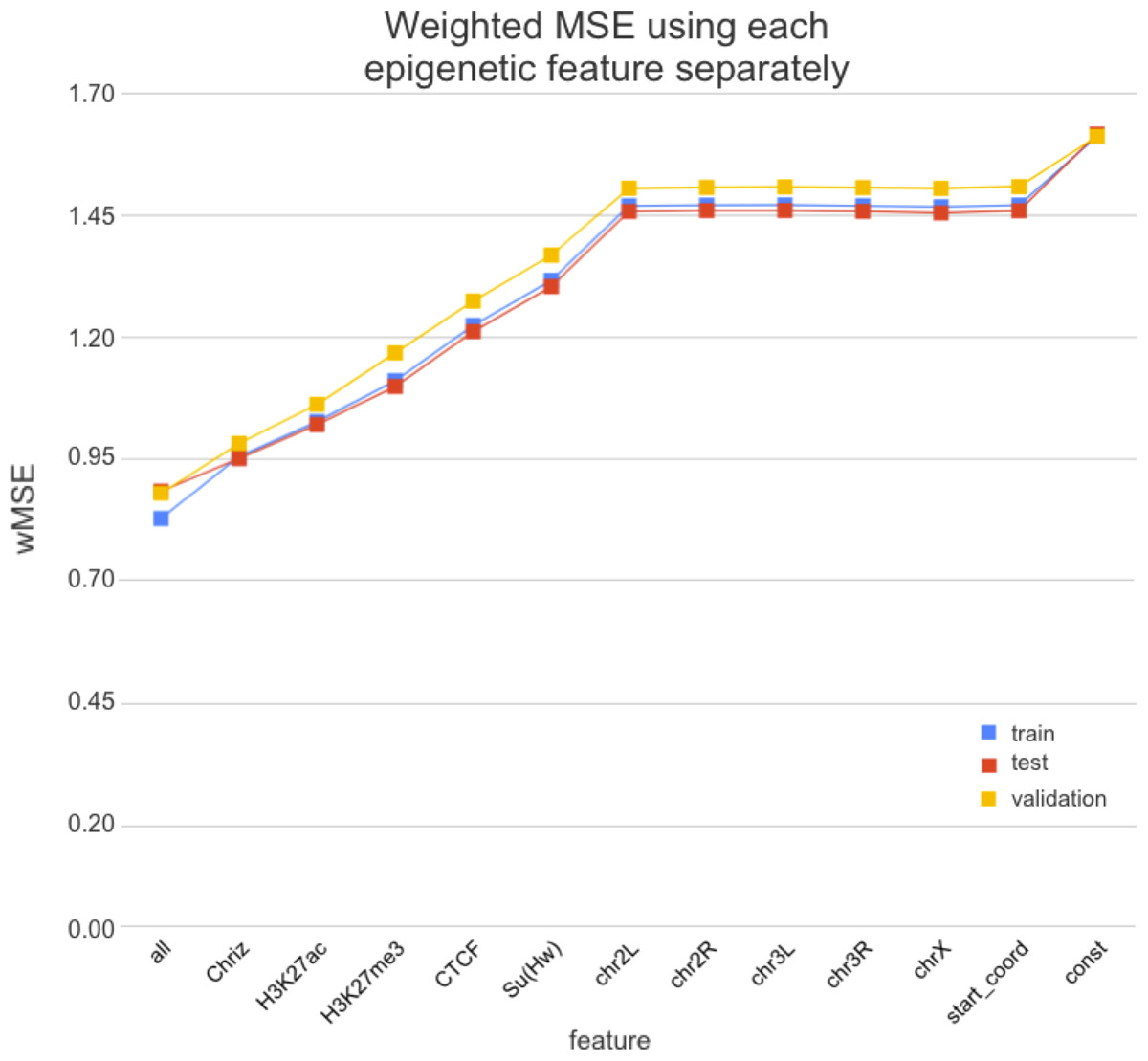
Может, ТАДы и не стоит искать?
Wang et al. 2018
5. Находим "визуальное" подтверждение
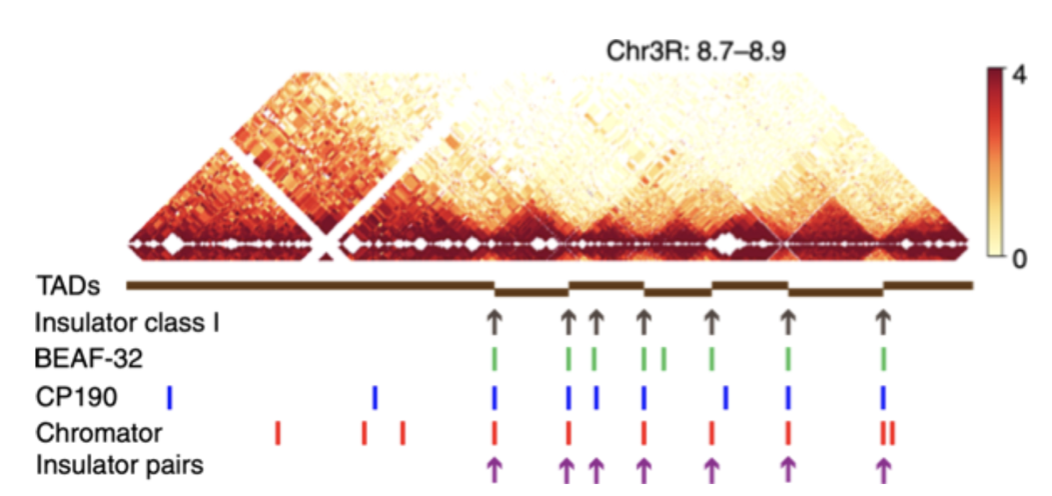
Компартменты
Bonev et al. 2016 Nature Reviews
одна из гипотетических моделей:
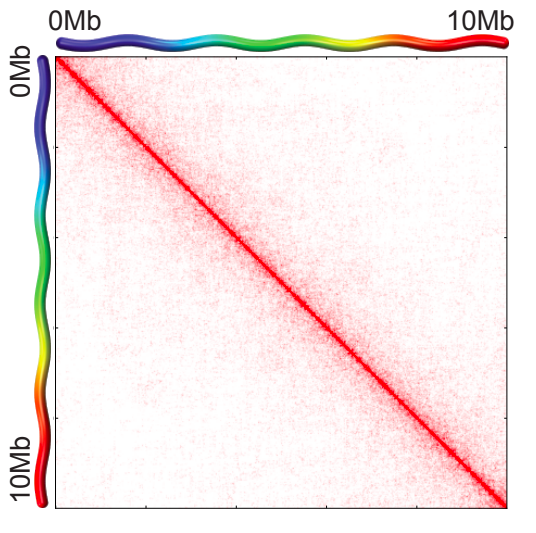
Вспоминаем: графики P(s)
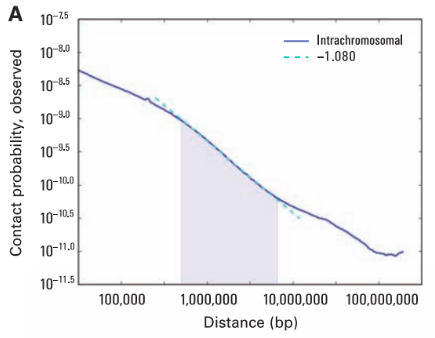
Lieberman-Aiden, 2009
Зависимость частоты контактов от геномного расстояния:
Алгоритм поиска компартментов
Lieberman-Aiden et al. Nature 2009
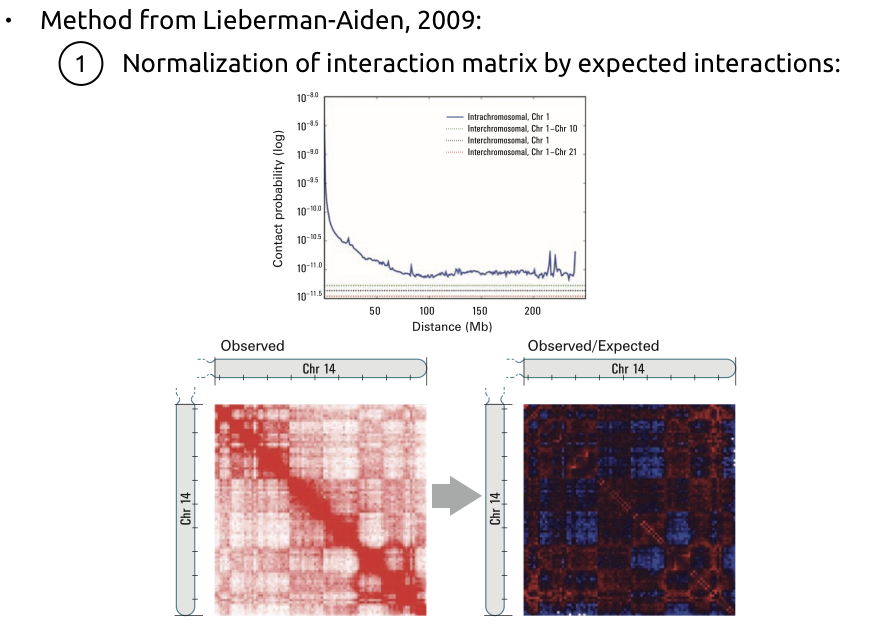
1. Нормализация матрицы контактов на P(s):
Алгоритм поиска компартментов
Lieberman-Aiden et al. Nature 2009
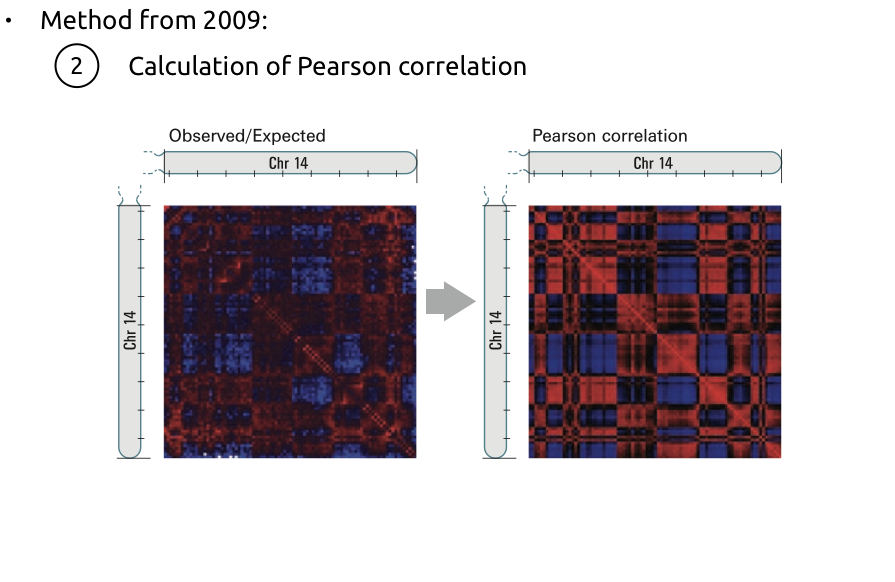
2. Усиление контрастности:
Алгоритм поиска компартментов
Lieberman-Aiden et al. Nature 2009
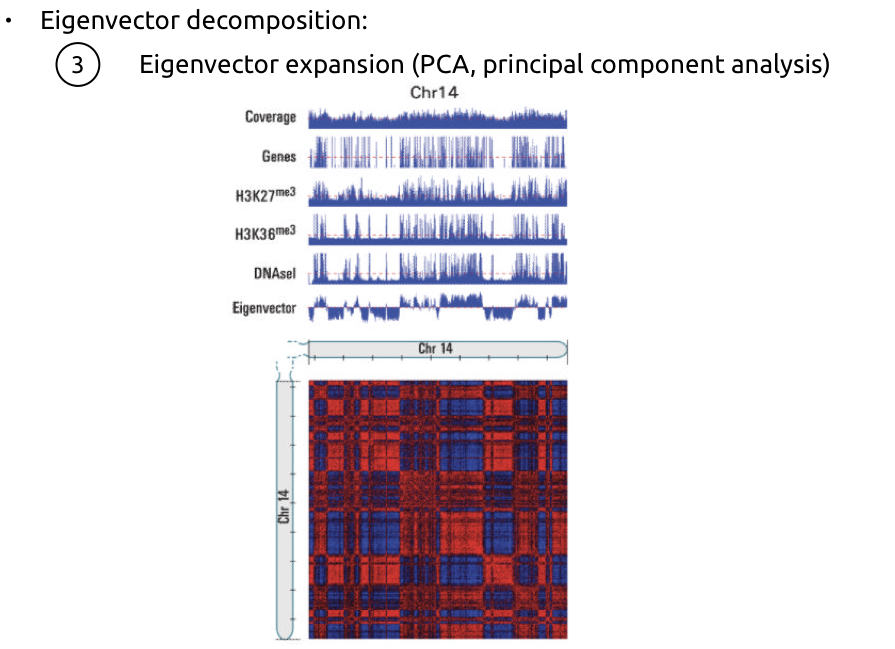
2. Оценка сходства между каждой парой строк:
(очень упрощая)
Оценка выраженности компартментов
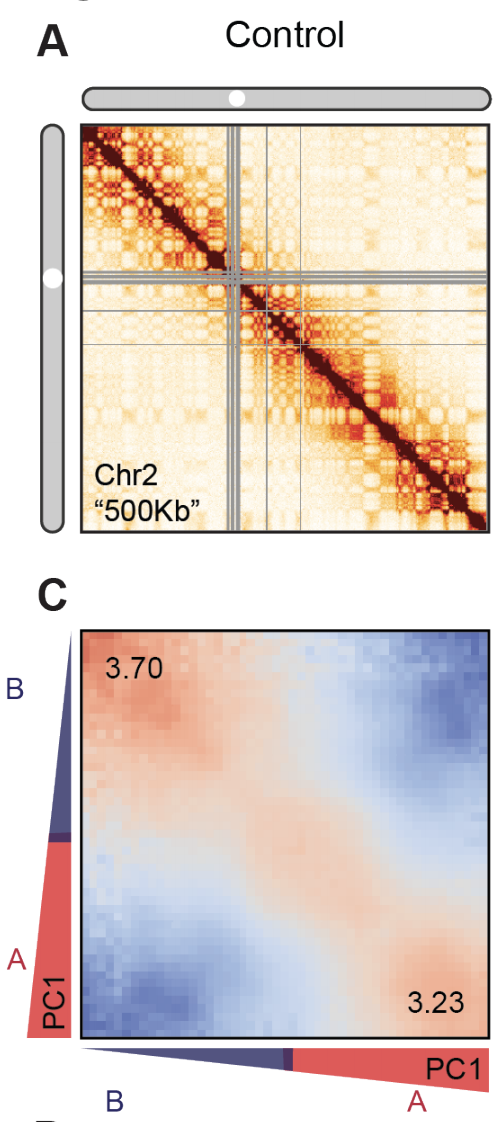

чем выше сигнал по углам карты,
тем более выражены компартменты
1. Найдем в Hi-C компартменты,
2. Нормируем Hi-C на P(s),
3. Переставим местами строки так, чтобы похожие были рядом,
4. Огрубим карту с помощью усреднения соседних пикселей.
Компартменты


Bonev et al. 2016 Nature Reviews
одна из гипотетических (!) моделей:
Шаг в сторону: single-cell Hi-C

Flyamer et al. Nature 2017

Single-cell Hi-C - протокол, похожий на Hi-C, но для единичных клеток:
Single-cell Hi-C, моделирование ДНК
Stevens et al. Nature 2017

Сила моделирования:
- хромосомные территории,
- активный хроматин (компартмент A) расположен
на периферии:
Всегда ли активный хроматин на периферии?
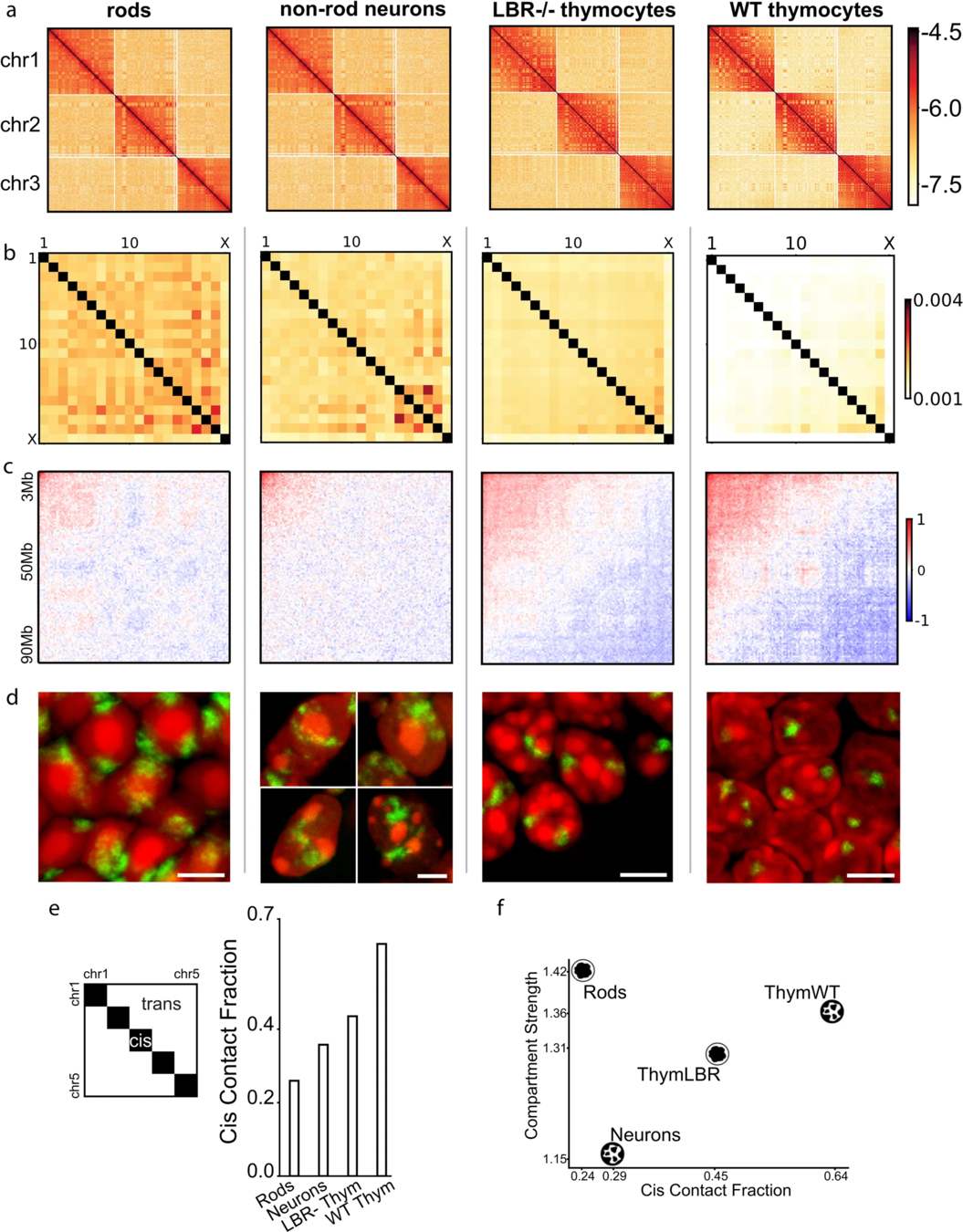
Falk et al. Nature 2019
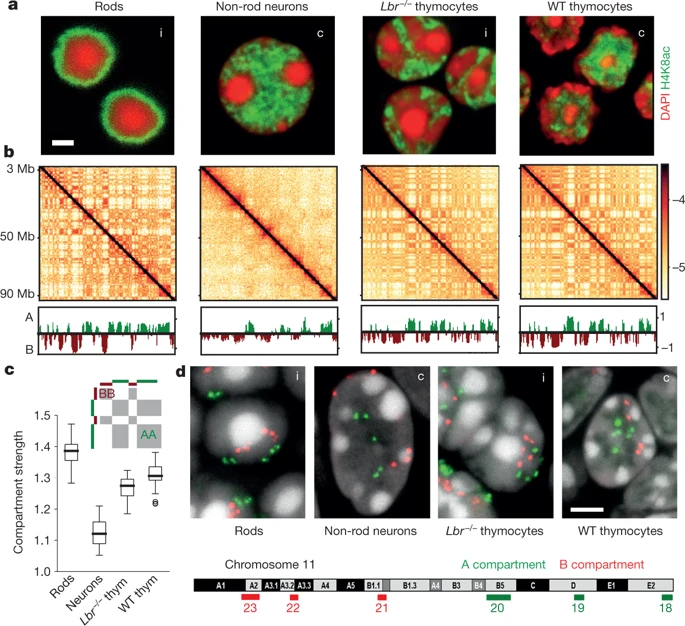
Результаты для типичных клеток:

нейроны
тимоциты
Флуоресцентная микроскопия
Карта Hi-C
Компартменты
Falk et al. Nature 2019



обычное ядро
На карте Hi-C видим одинаковые компартменты, но принципиально разные результаты микроскопии!
Всегда ли активный хроматин на периферии?
мутантные тимоциты
тимоциты
Флуоресцентная микроскопия
Карта Hi-C
Компартменты
инвертированные ядра
колбочки глаза
Solovei, 2009
Рассмотрим колбочки глаза:
Когда активный хроматин не на периферии ядра?
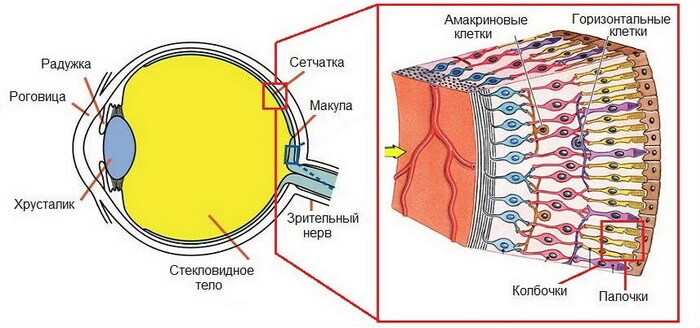
Solovei, 2009
Ядра колбочек мышей - инвертированы!
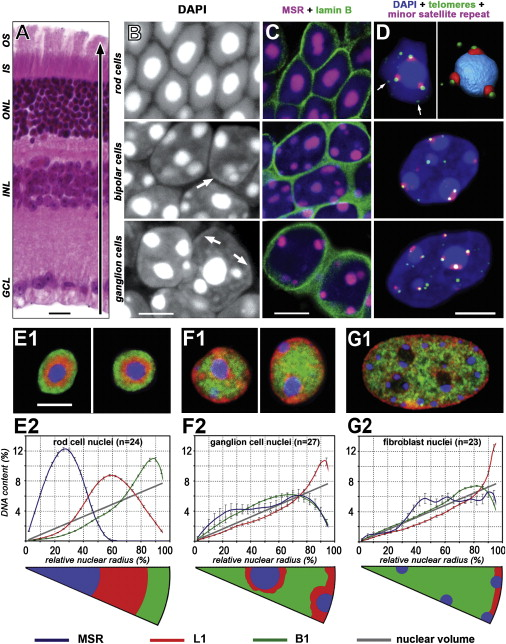
Когда активный хроматин не на периферии ядра?

колбочки
(тела клеток)
биполярные клетки
ганглиолярные клетки
срез сетчатки
колбочки
(ядра клеток)
колбочки
ганглиолярные клетки
фибробласты
Solovei, 2009
Ядра колбочек у ночных
животных - инвертированы!
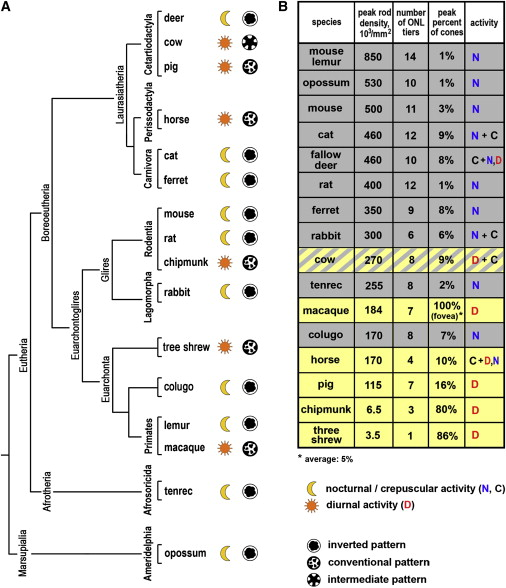
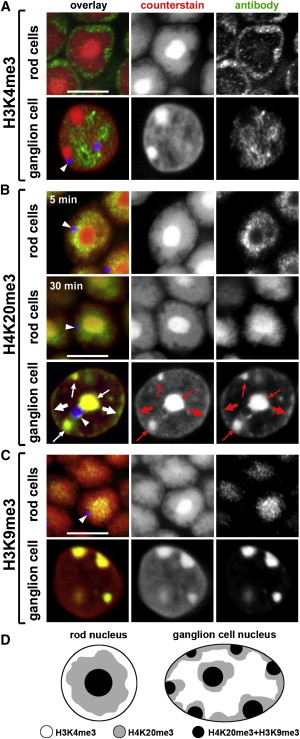
Когда активный хроматин не на периферии ядра?
Пример для лимфобластоидной клетки GM12878 (обращайте внимание на сокращения названий линий клеток - это ключ к пониманию их биологии):
- компартменты выражены
- не можем отличить инвертированное ядро от обычного.
Пример из реальной жизни
Перерыв?
Обогащенные взаимодействия (петли)
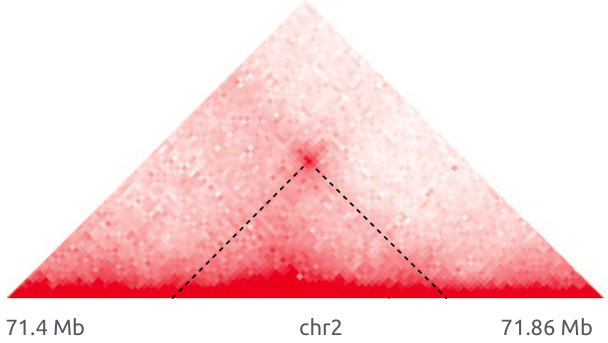
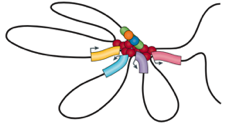
Архитектурные петли
Промотор-энхансерные взаимодействия
Polycomb-петли
Bonev et al. 2016 Nature Reviews
Алгоритм поиска петель: HiCCUPS
Rao et al. Cell 2014
Hi-C Computational Unbiased Peak Search
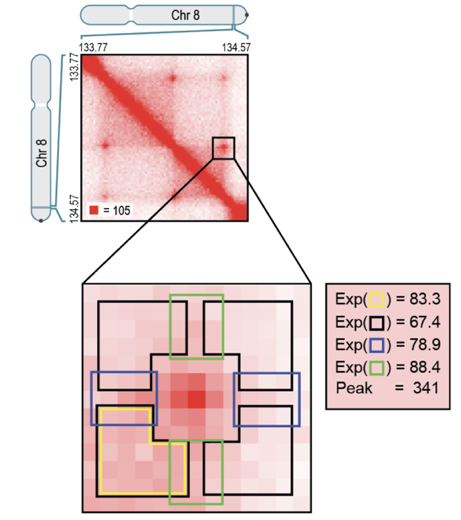
Свойства петель хроматина
Rao et al. Cell 2014
Позиции петель млекопитающих часто содержат мотив связывания фактора CTCF:
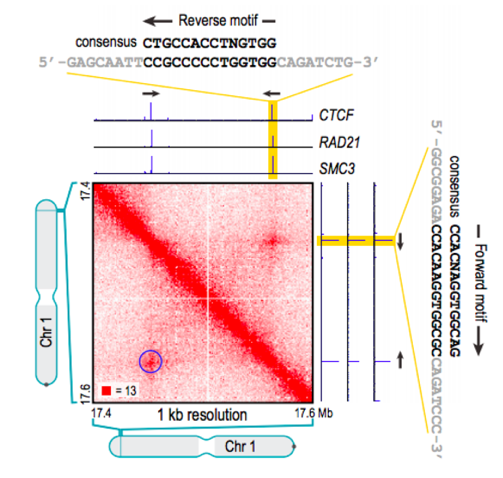
Свойства петель хроматина
Li et al. Nature 2020
CTCF важен потому, что останавливает экструзию
(см. Лекцию 1)
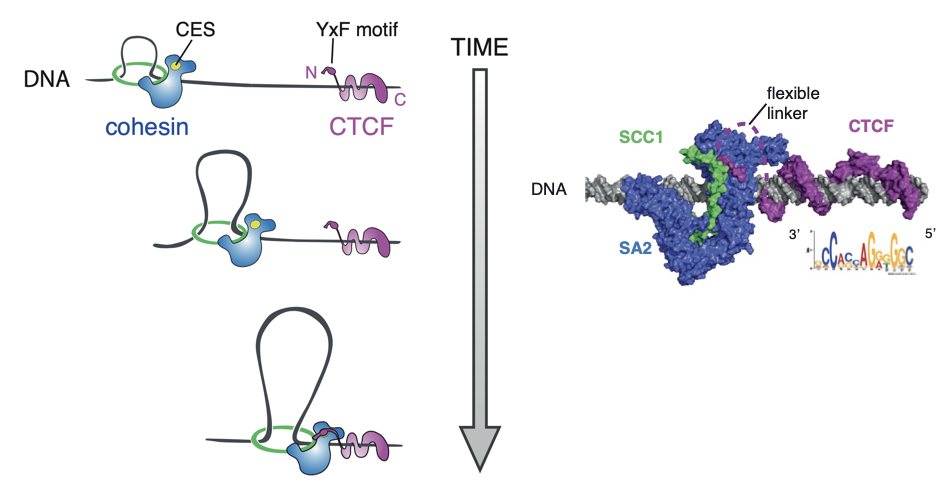
Свойства петель хроматина


Свойства петель хроматина
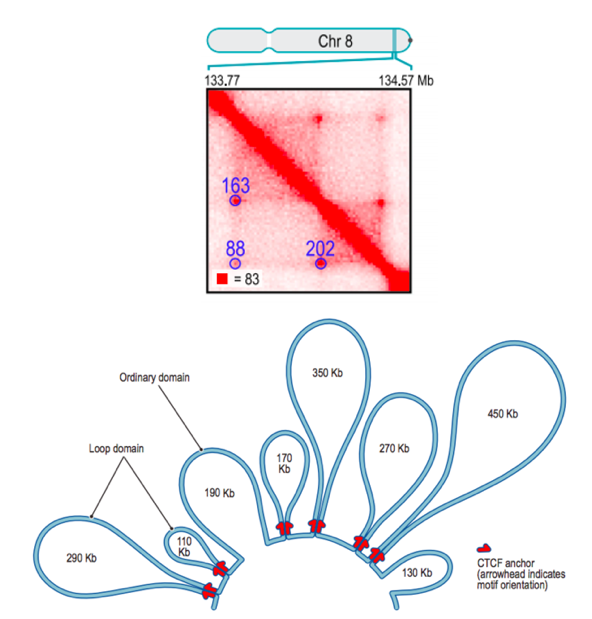
Rao et al. Cell 2014
Петли образуют более интересные структуры...
(вспоминаем пример с Лекции 1)
Разнообразие алгоритмов поиска петель
Forcato et al. Nature Methods 2017
Огромное количество алгоритмов, как и в случае поиска ТАДов:
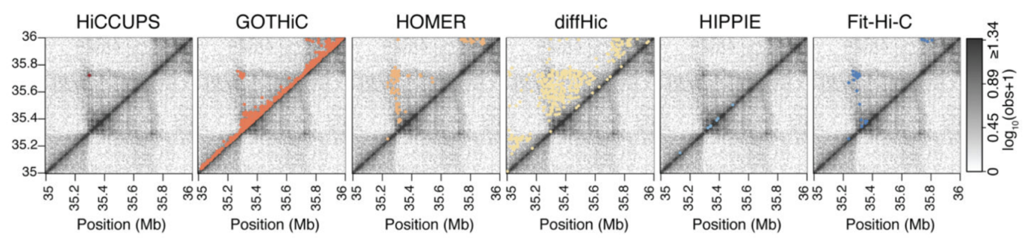
Всегда ли нужно искать петли?
Flyamer Bioinformatics 2019
Можем взять все позиции связывания CTCF и построить среднюю картину петли:
https://github.com/Phlya/coolpuppy


позиции CTCF
средняя петля
Усреднение - мощный инструмент
Ulyanov, Galitsyna et al. 2020, Nature Communications, in press
На примере single-cell Hi-C данных мухи, очень мало данных:
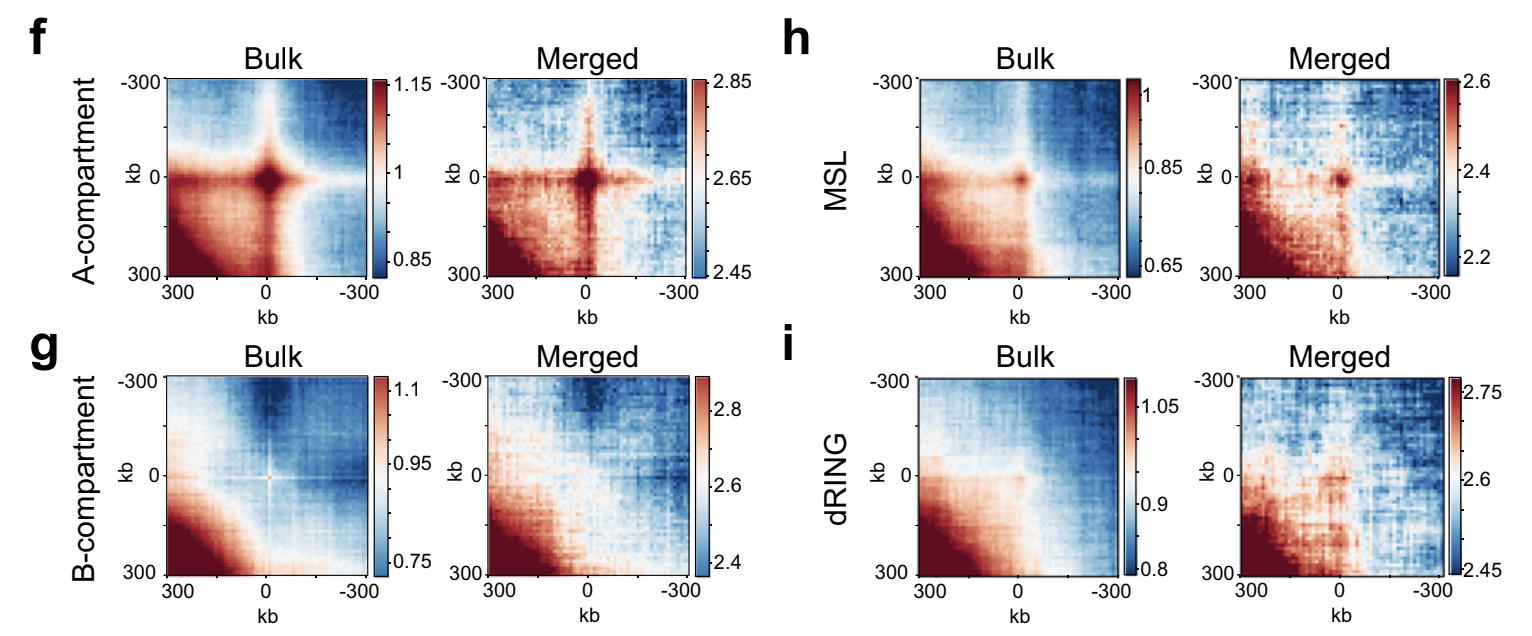
Консультация
по домашней работе
FAQ
Q: Будет ли еще одна домашняя работа по теме хроматина?
A: Нет, не будет. Единственная домашняя работа была выдана 3 декабря, дедлайн для ее выполнения - 09:00 утра 17 декабря.
Q: Когда будет квиз?
A: Следующий квиз будет 17 декабря.
Q: Я пропустил(а) квиз. Будет ли пересдача?
A: Нет, не будет.
Q: Я скачал(а) данные из публичной папки на кластере, однако в моем наборе нет файлов с геномными индексами. Что делать?
A: Действительно, такая проблема наблюдалась пару дней назад. Сейчас файлы на месте: проверьте еще раз. (для продвинутых: используйте bwa mem, чтобы получить свои индексированные файлы!)
Перерыв
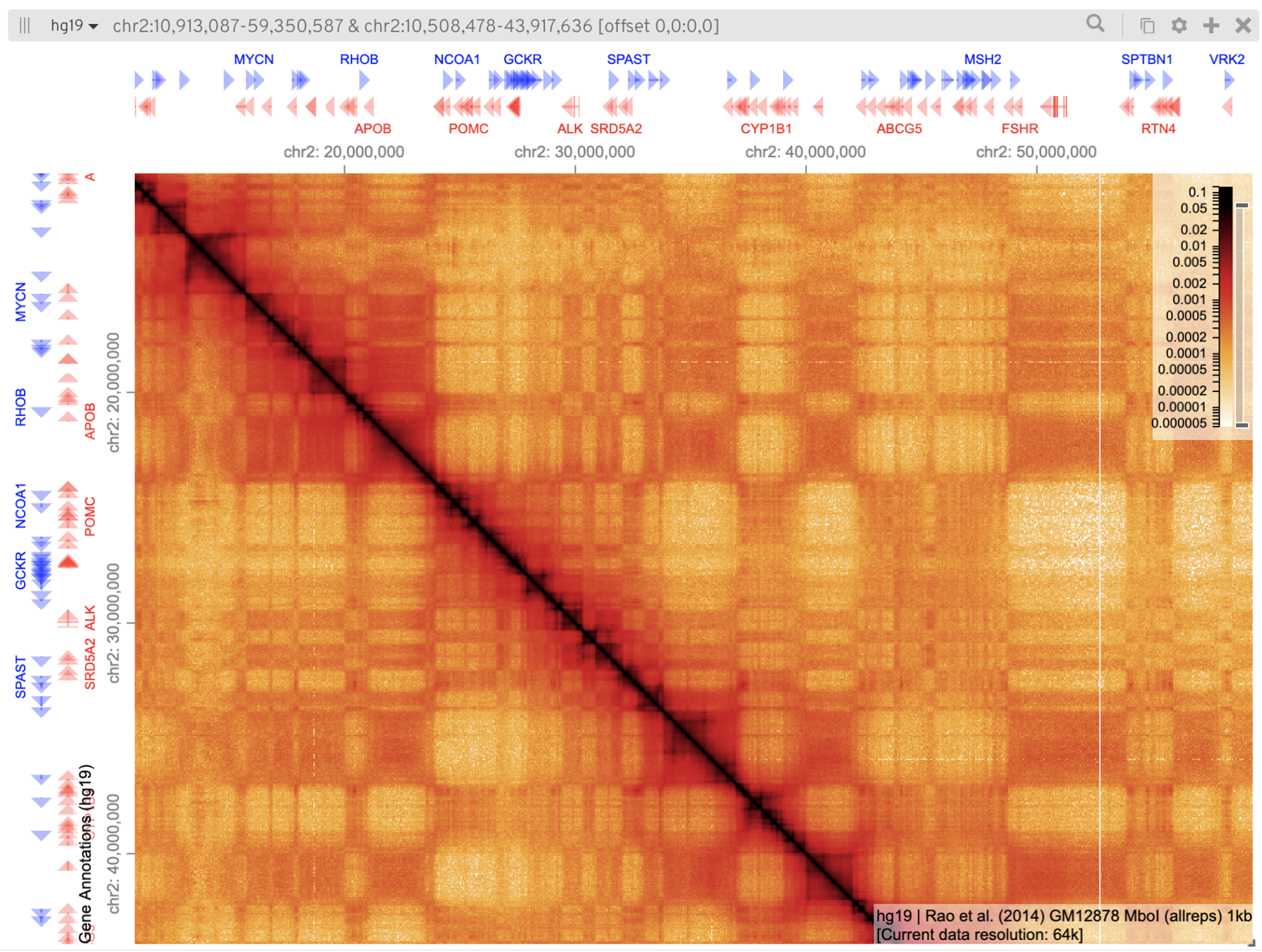
Работа с браузерами Hi-C: Juicebox
Разбираемся с петлями и ТАДами с помощью Juicebox:
https://aidenlab.org/juicebox/
1. Загрузите данные для клеточной линии GM12878 из Juicebox Archive
2. Приблизьте карту на регион одной хромосомы. Поменяйте цветовую шкалу при необходимости
3. Найдите хотя бы одну петлю и хотя бы один ТАД.
Какое разрешение подходит для их визуализации?
4. Загрузите 2D аннотации (петли и домены типа Combined).
Как связаны между собой разметки петель и ТАДов?
5. Добавьте данные RNA-Seq. Как они соотносятся с позициями петель и ТАДов?
Работа с браузерами Hi-C: HiGlass
Разбираемся с разными экспериментами через HiGlass:
1. Перейдите на Two Linked View раздел.
Какие типы клеток перед вами?
2. Одинаковая ли структура хроматина в заданном регионе?
3. В каких типах клеток (из двух) гены OXR1, ABRA, ANGPT1 находятся в "активном" хроматине?
4. Какие структуры хроматина ассоциированы с активными генами?
Вопрос: Используя информацию из литературы и публичных баз данных, как можно объяснить дифференциальную активность генов OXR1, ABRA, ANGPT1 между двумя приведенными клеточными типами?
Жду развернутый ответ на agalitzina@gmail.com или Telegram @agalicina. Верные и научно обоснованные гипотезы будут засчитаны вместо квиза 17 декабря.



