Трехмерная организация структуры ДНК
Александра Галицына
Цикл лекций о структуре хроматина
для Высшей Школы Экономики
25 ноября 2021
Что предстоит?
25 ноября:
- одна очень важная лекция, которая познакомит с областью и актуальными задачами
- домашняя работа с письменным ответом
2 декабря:
- квиз (обязательный и ограниченный по времени)
- лекция о методах работы с данными о 3D-геноме
- практическая работа на сервере, по которой будет домашняя работа
9 декабря:
- квиз (обязательный и ограниченный по времени)
- заключительная лекция
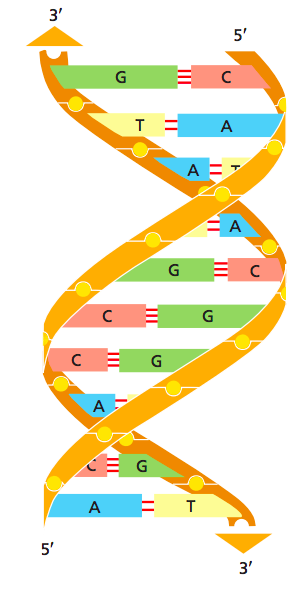
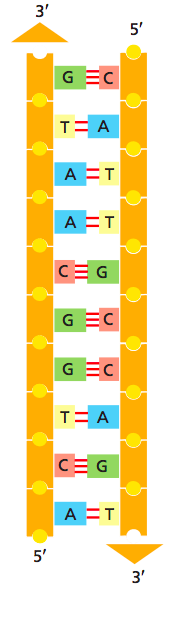
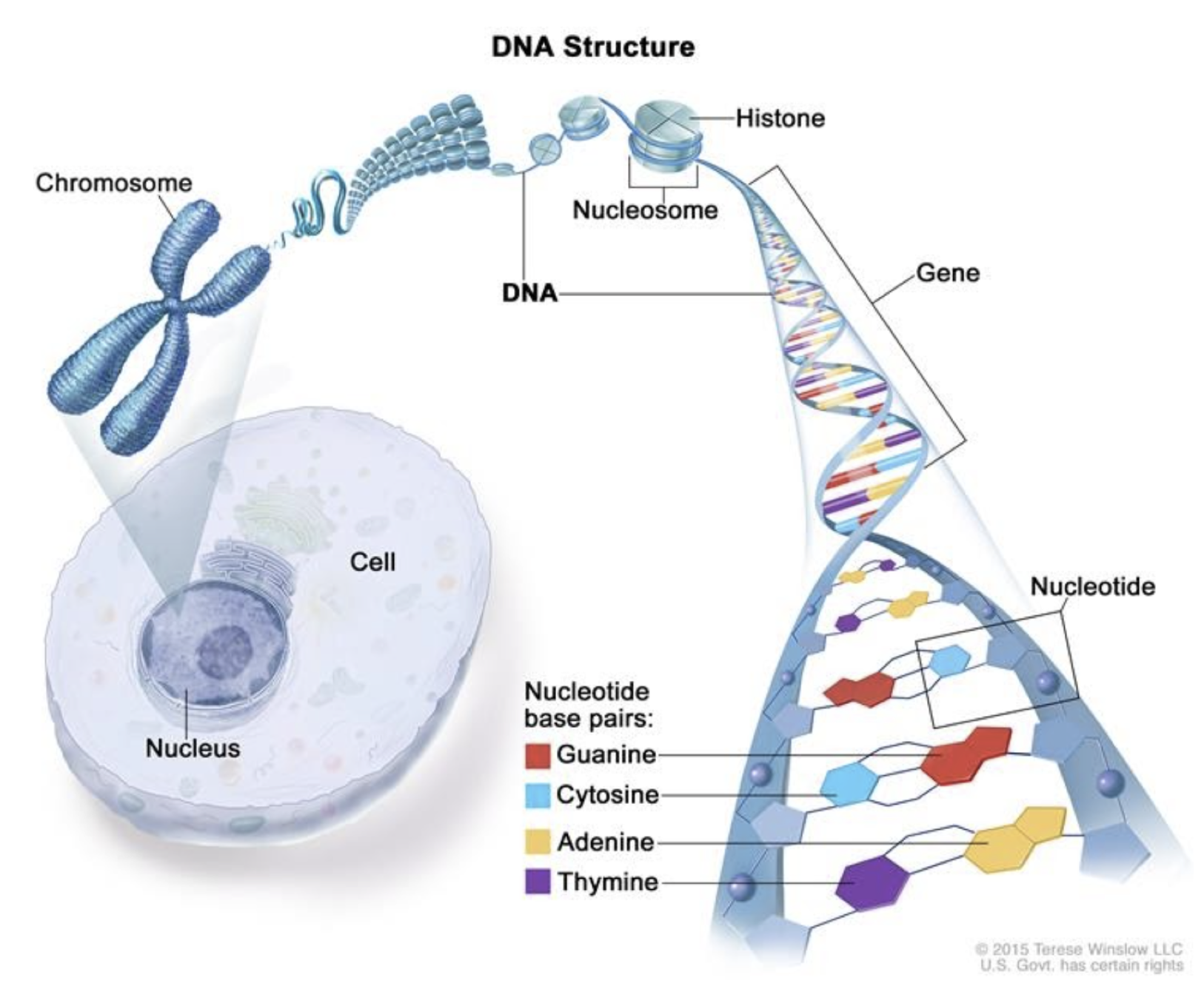
Классические представления о ДНК
- ДНК - это носитель генетической информации, линейно упорядоченная молекула биополимера нуклеиновых кислот.

ДНК как двойная спираль
- Уотсон, Крик и Франклин: рентгеноструктурный анализ (1953)

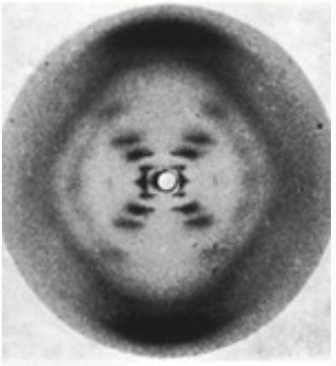
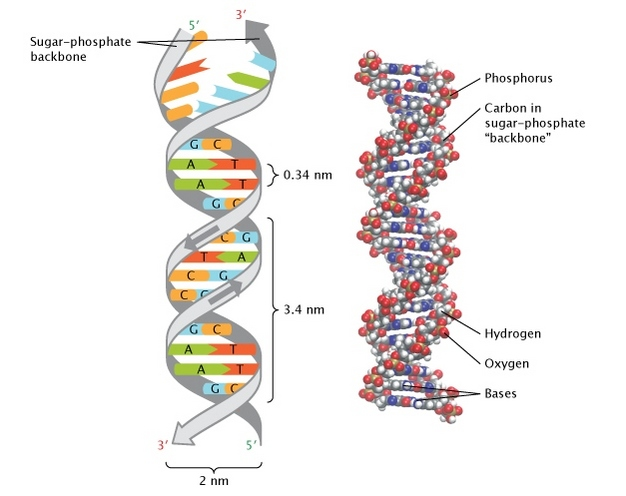
Pray Nature Education 2008
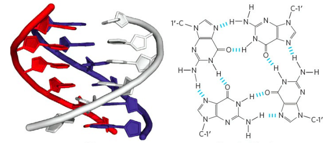


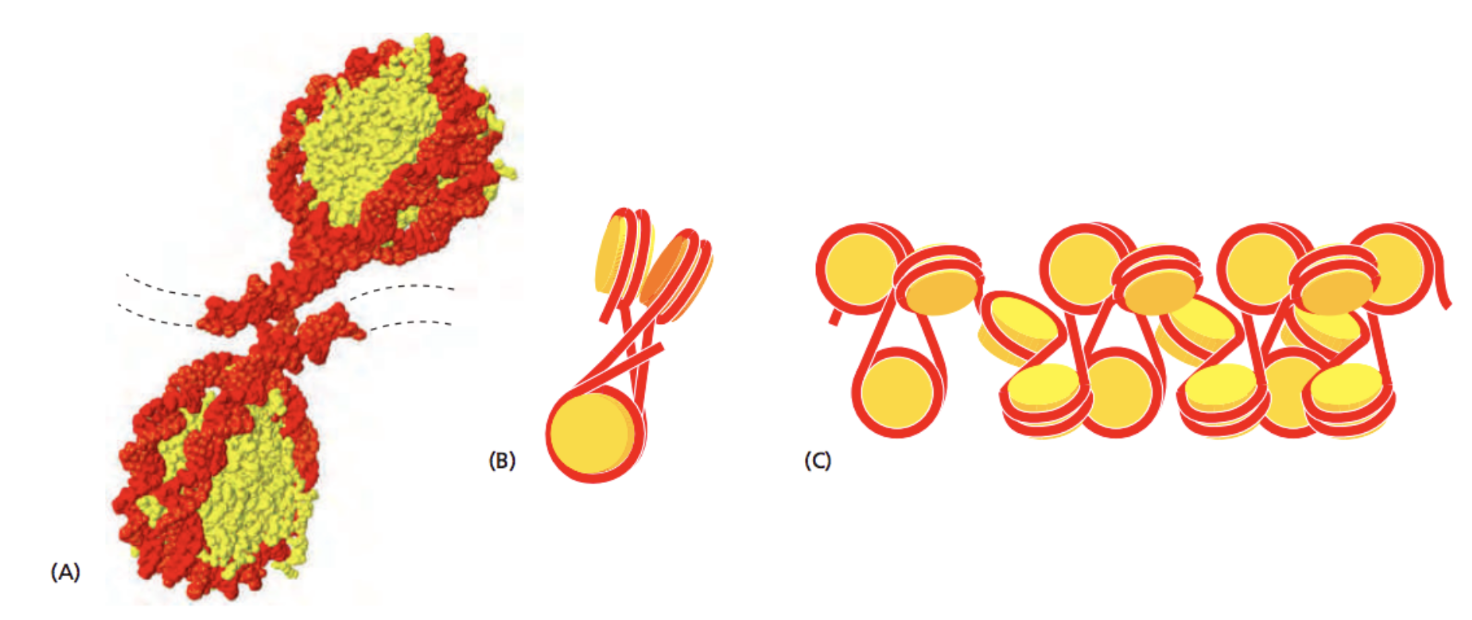
Уровни организации хроматина
Первичная структура
Вторичная структура
A-форма
спирали
B-форма
Z-форма
G-квадруплекс
ДНК-шпилька
Хроматин - комплекс ДНК и белков
ДНК образует более сложные структуры за счет связывания белков. В первую очередь, это гистоновые белки (у эукариот):
Alberts "Molecular Biology of the Cell" 6th edition 2015
Хроматин - комплекс ДНК и белков
Robinson, 2016
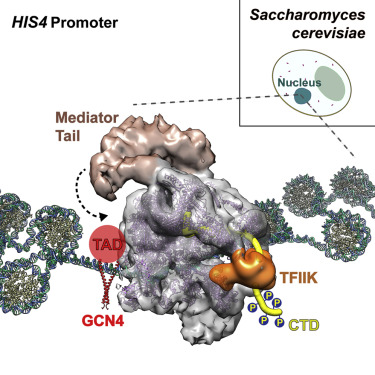
Регуляторы экспрессии генов: энхансеры
Для активации экспрессии необходима сборка белкового комплекса на специальных регуляторных последовательностях ДНК - энхансерах.
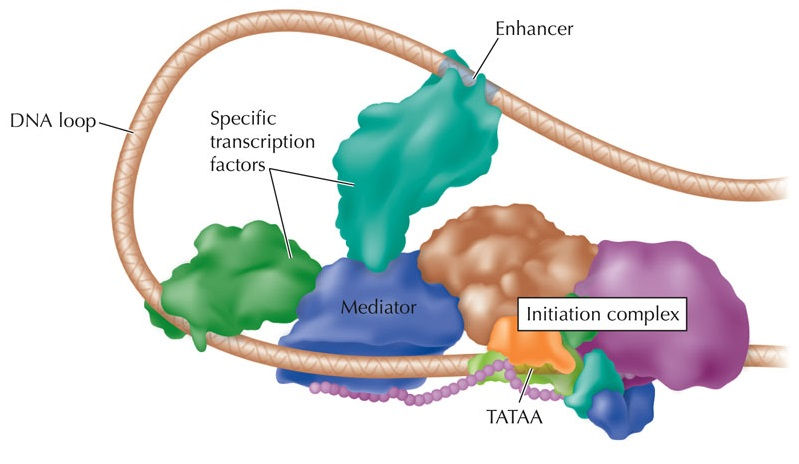
Энхансер
Промотор
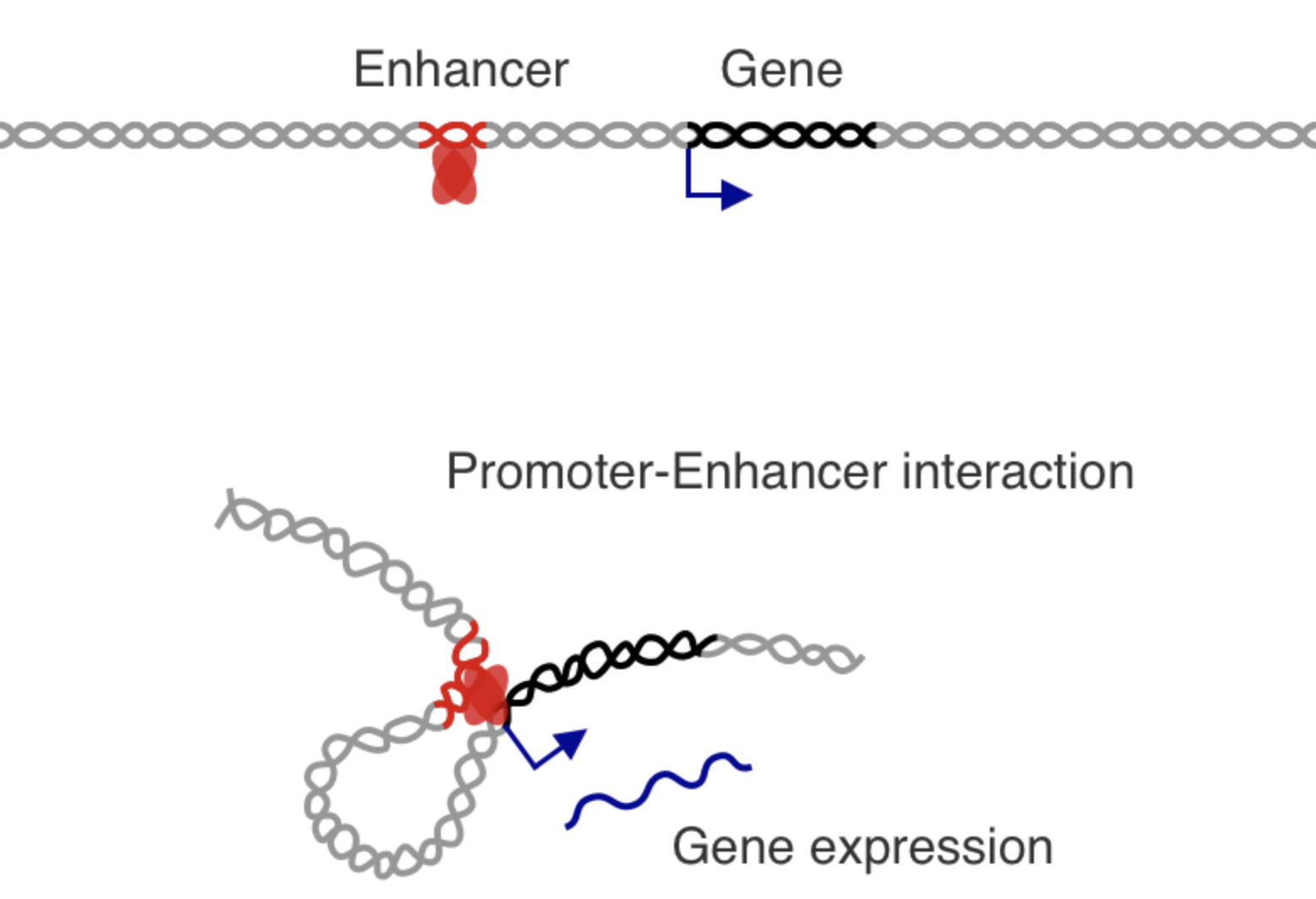
Как привести во взаимодействие промотор и энхансер?
Dean 2006
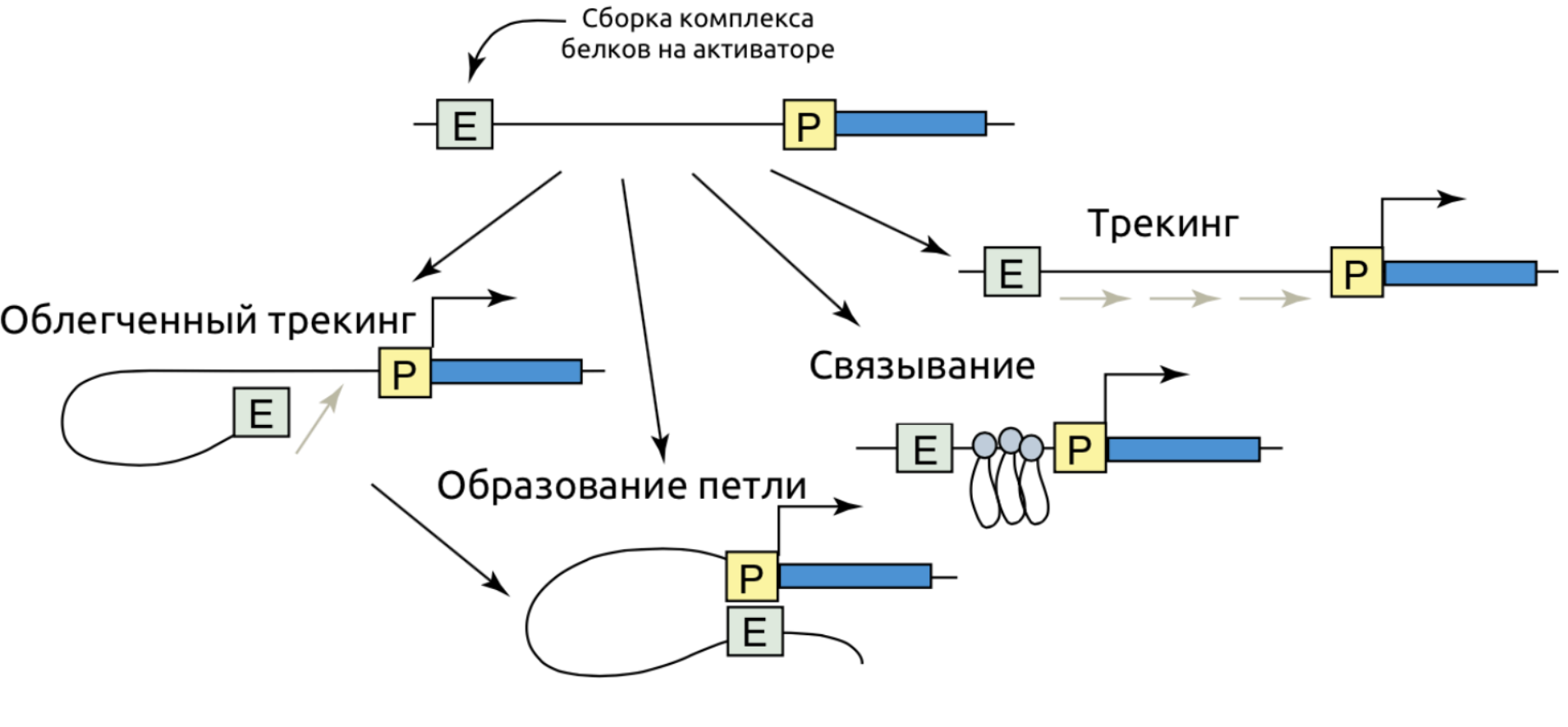
Wasserman & Sandelin. Applied bioinformatics for the identification of regulatory elements. Nat. Rev. Genet. 2004
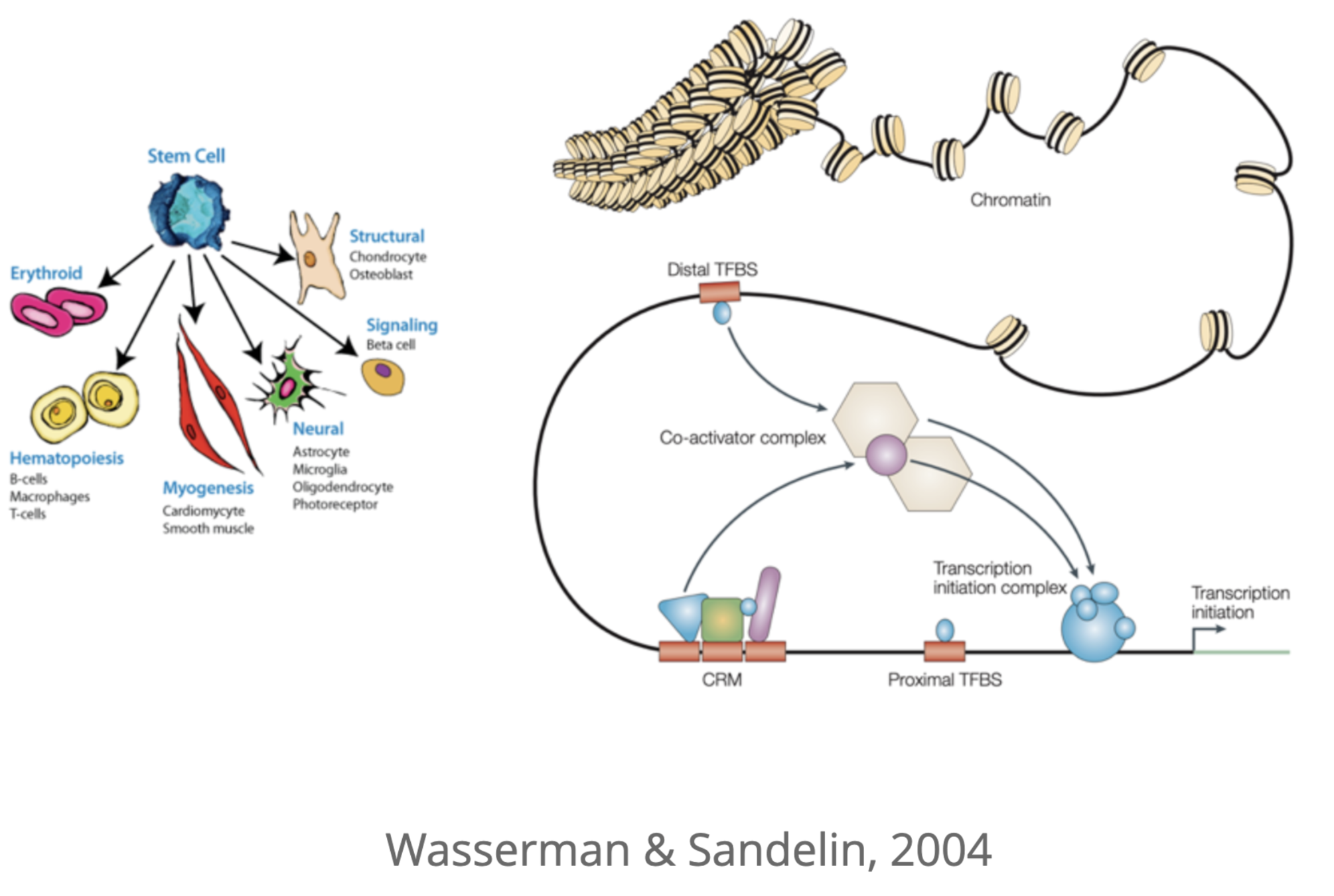
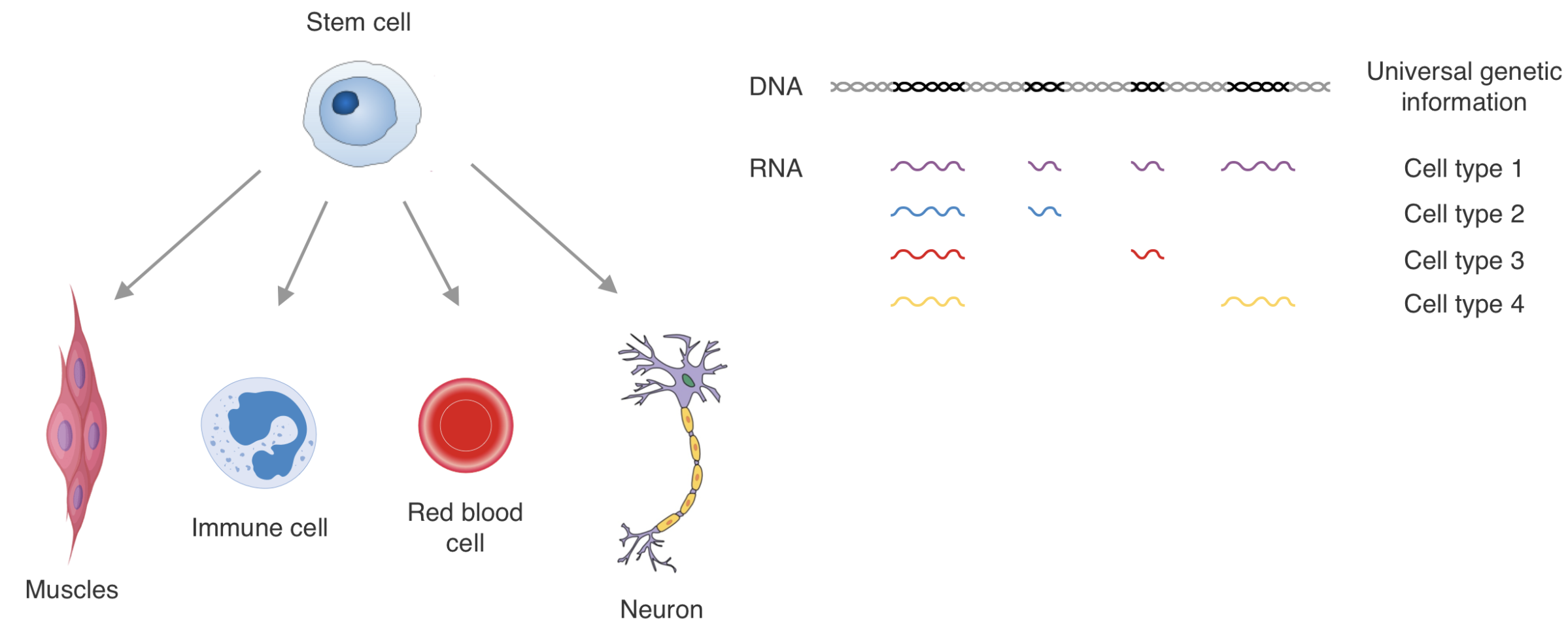
Регуляторные сети энхансеров
Один ген может регулироваться несколькими энхансерами,
один энхансер может регулировать несколько генов:
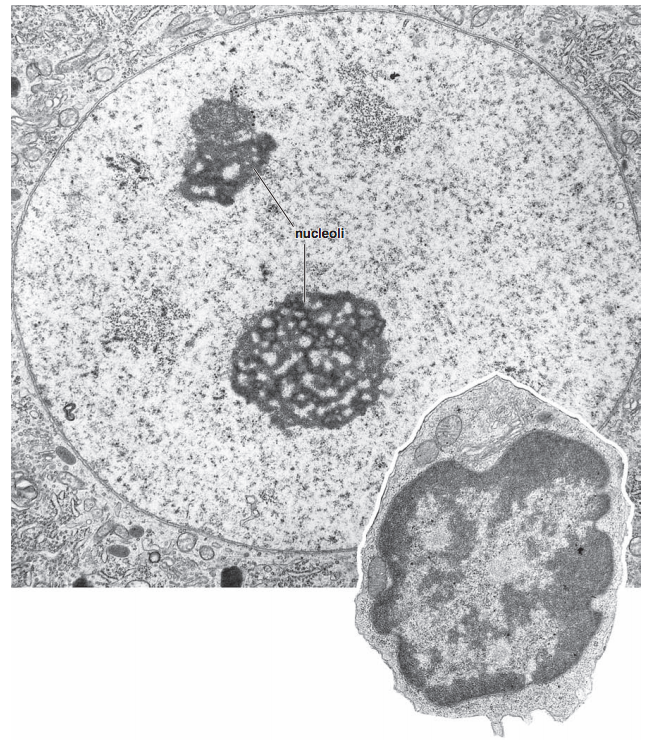
Два подхода: микроскопия и молекулярная биология




Микроскопия ядра
Ros 2006 "Histology Atlas with Correlated Cell and Molecular Biology"
FISH-микроскопия
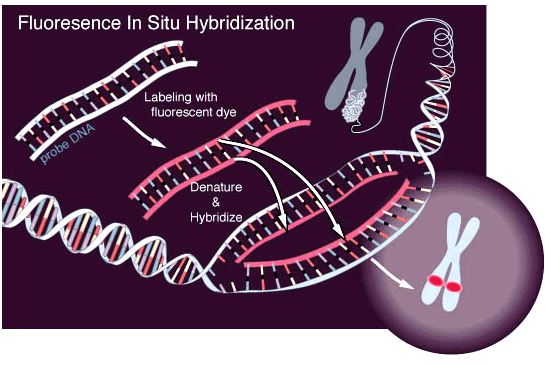
Fluorescent in situ hybridization
FISH-микроскопия
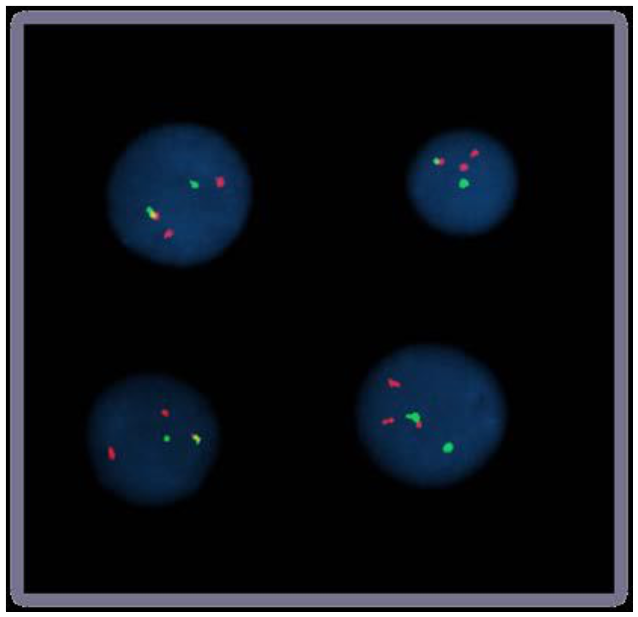
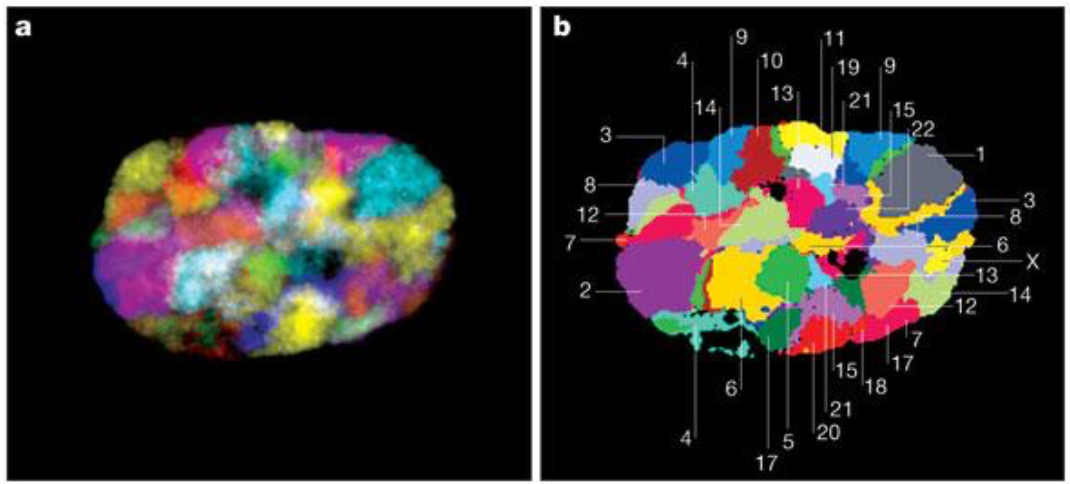
Bolzer et al., PLoS Biol. 2005
Speicher & Carter Nature 2005
Fluorescent in situ hybridization
Подходы к исследованию хроматина и "дальней" регуляции
4DN Nucleome
4DN Nucleome
The goal of the Common Fund’s 4D Nucleome (4DN) program is to study the three-dimensional organization of the nucleus in space and time (the 4th dimension).
...
The 4DN program has generated a variety of tools and resources so scientists can continue to learn about the importance of nuclear organization. Program deliverables currently available through the public 4DN Portal include nearly 2000 datasets from hundreds of experiments, 52 software packages and 23 protocols and reagents for researchers to use.
(2020)
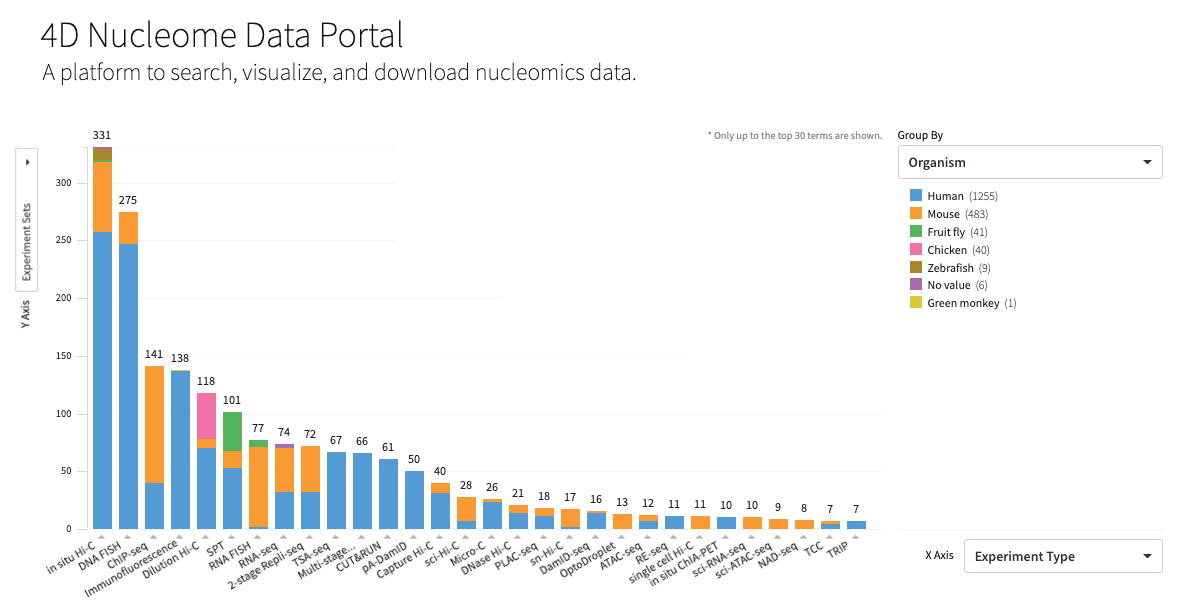
4DN Nucleome
Data Portal:
Фиксация конформации хромосом: 3C
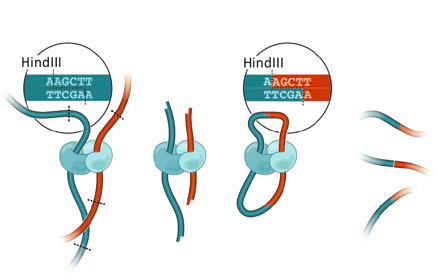
Фиксация формальдегидом
Рестрикция ДНК
Лигирование
Очистка ДНК
Библиотека ДНК-ДНК контактов
3C: Dekker et al. 2002 Science
Hi-C: высокопроизводительная фиксация конформации хромосом
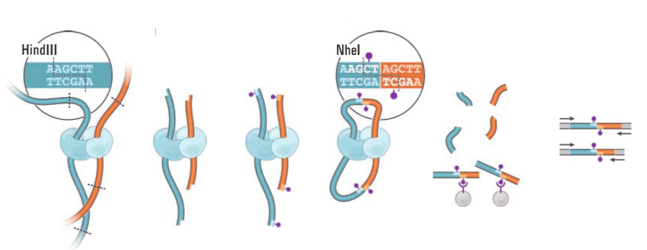
Фиксация формальдегидом
Рестрикция ДНК
Лигирование
Очистка ДНК
Секвенирование
Картирование
Lieberman-Aiden et al. 2009 Science
Лигирование
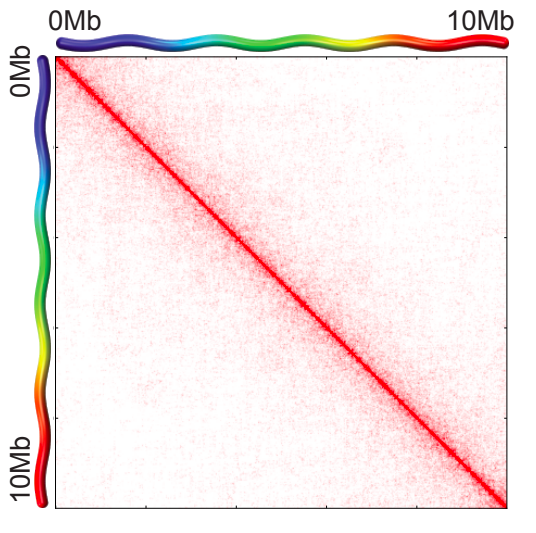
Теплокарта ДНК-ДНК контактов
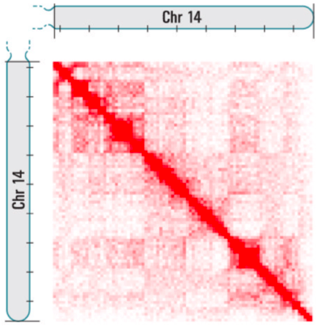
Lieberman-Aiden et al. 2009 Science
Цвет: частота взаимодействий регионов ДНК
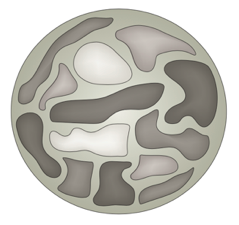
Хромосомные территории
Bonev et al. 2016 Nature Reviews
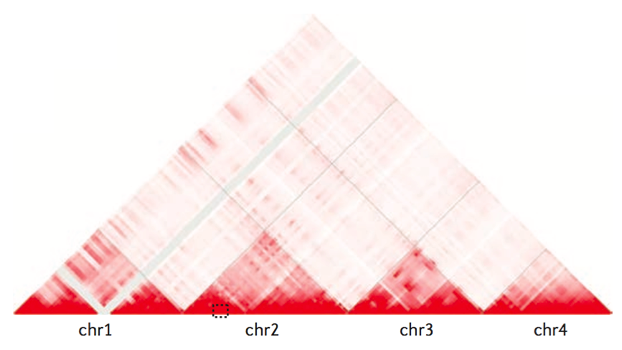

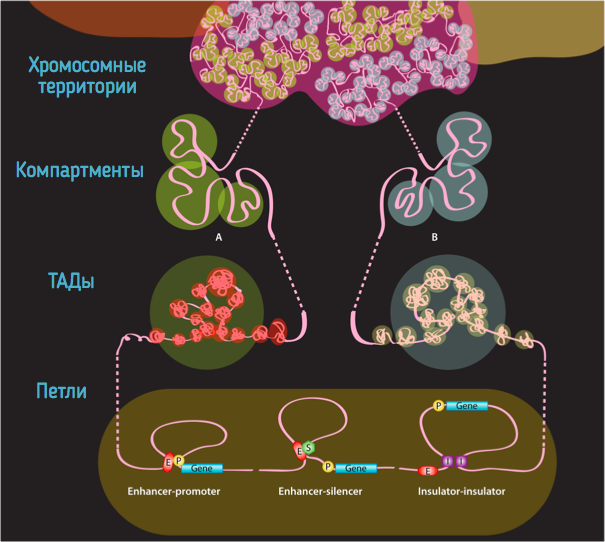
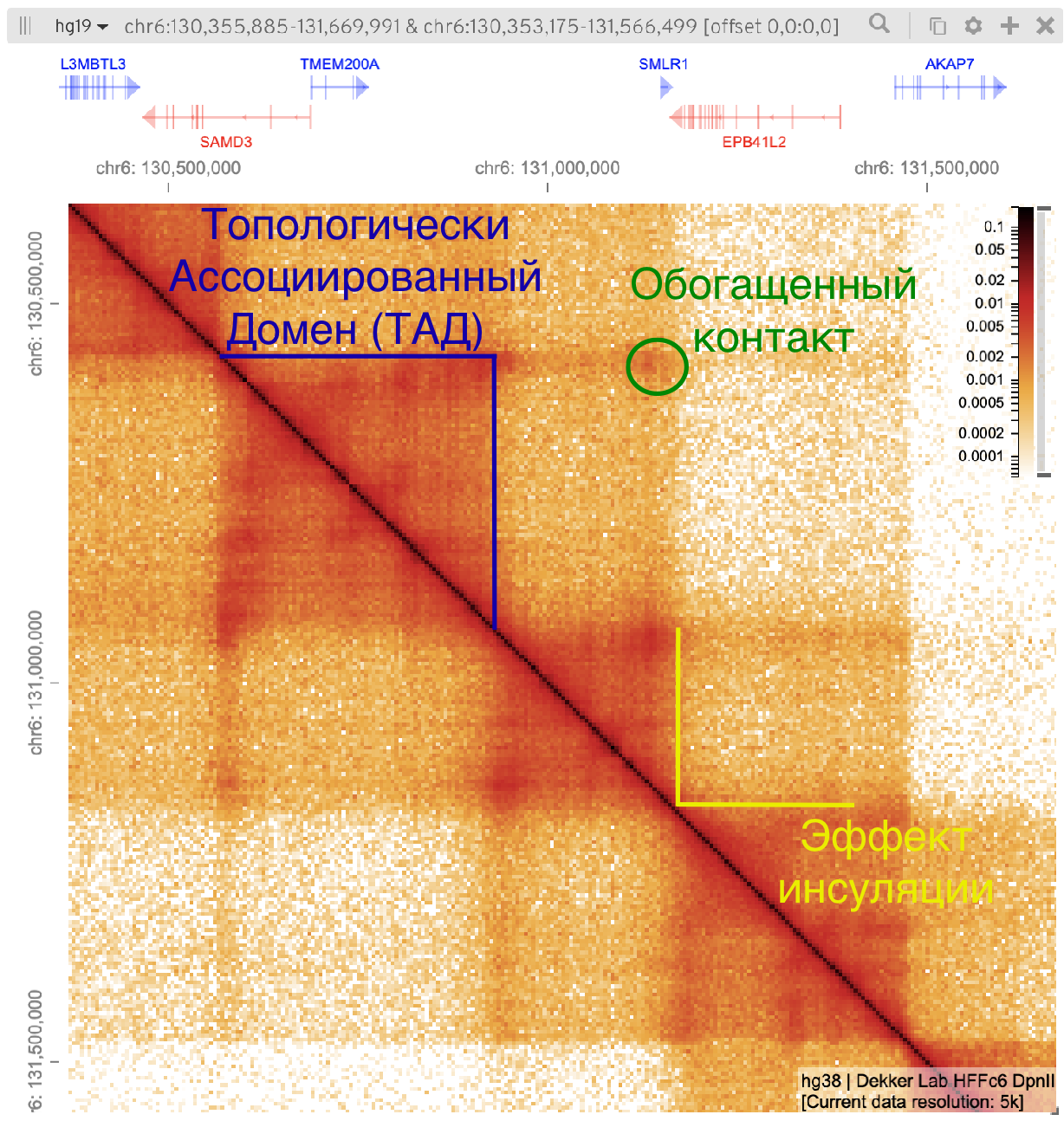
Топологически ассоциированные домены (ТАДы)

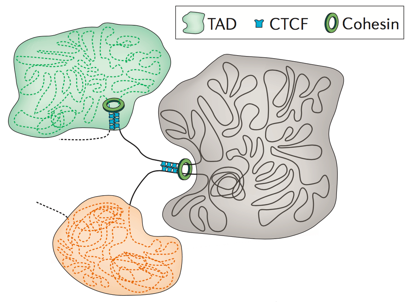
Bonev et al. 2016 Nature Reviews
Компартменты
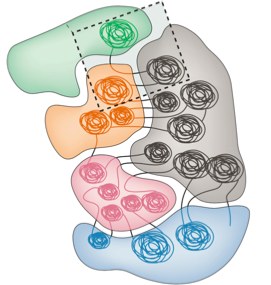
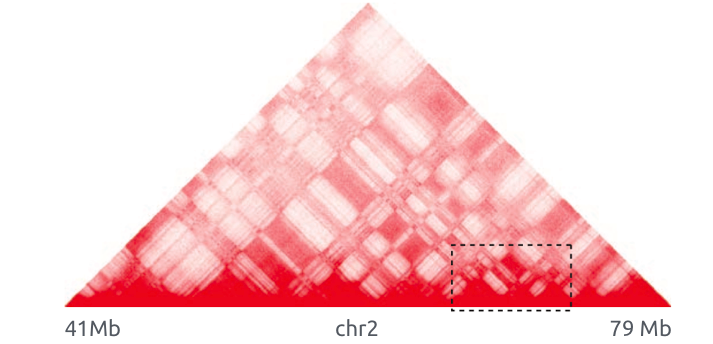
Bonev et al. 2016 Nature Reviews
Обогащенные взаимодействия
Архитектурные контакты
Промотор-энхансерные взаимодействия
Polycomb-петли
Bonev et al. 2016 Nature Reviews
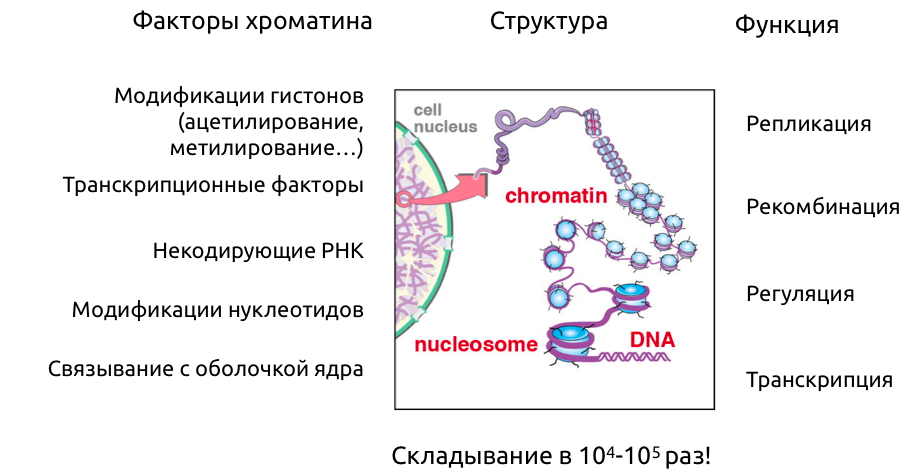
Уровни иерархии хроматина
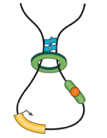
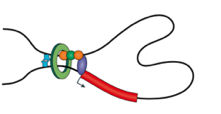
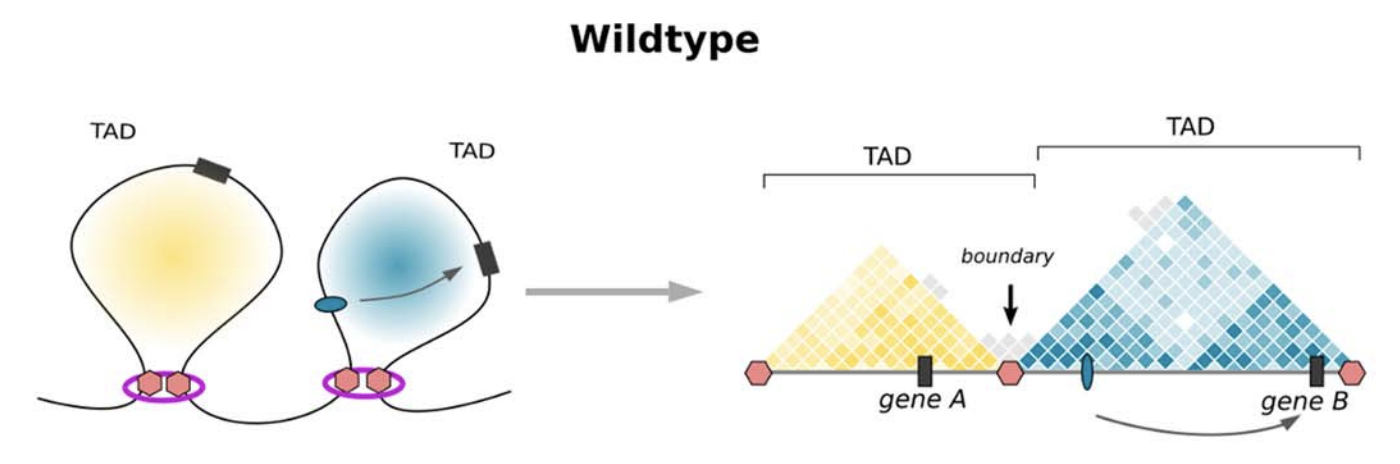
Модель выпетливания
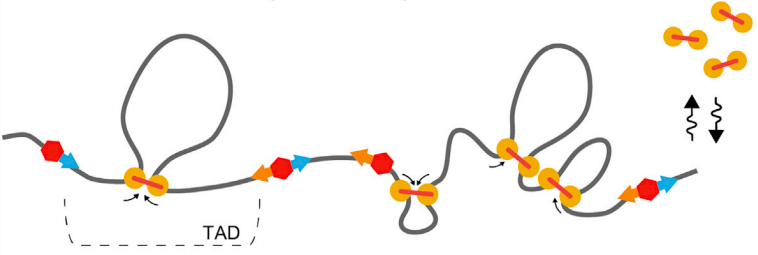
Fudenberg et al. 2016 Cell Reports

когезин - выпетливающий фактор
CTCF - граничный элемент
Экструзия петель - основная гипотеза формирования структуры хроматина
MirnyLab Youtube channel
Упрощенная визуализация ключевых компонентов:
ДНК, экструдера (когезин) и барьерного элемента (CTCF)
Ganji et al. 2018 Science
Ganji et al. 2018 Science
Прямое подтверждение экструзии
Модель активной компактизации хромосом
Mirny Lab Youtube channel
ДНК как активно живущий "город", наполненный активными машинками-экструдерами
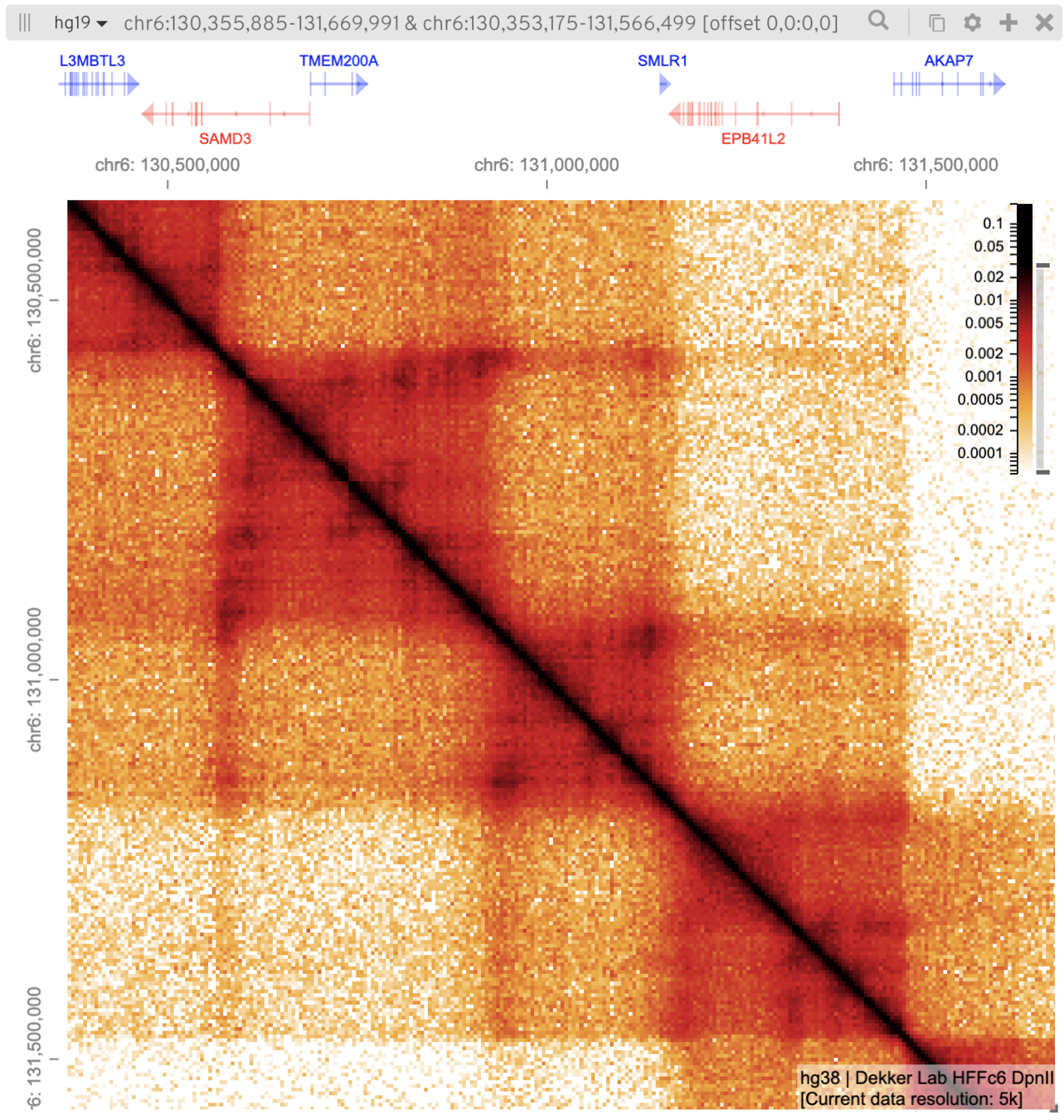
Архитектурный белок хроматина CTCF
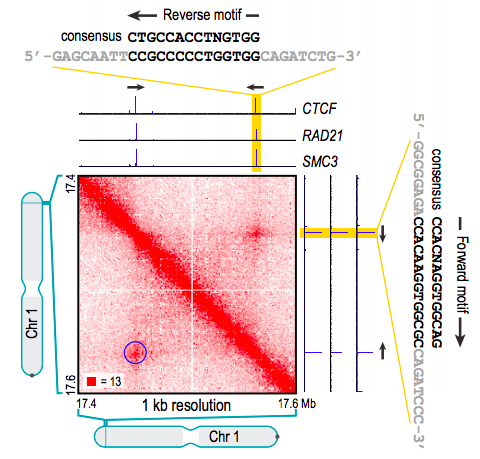
Rao et al. 2014 Cell
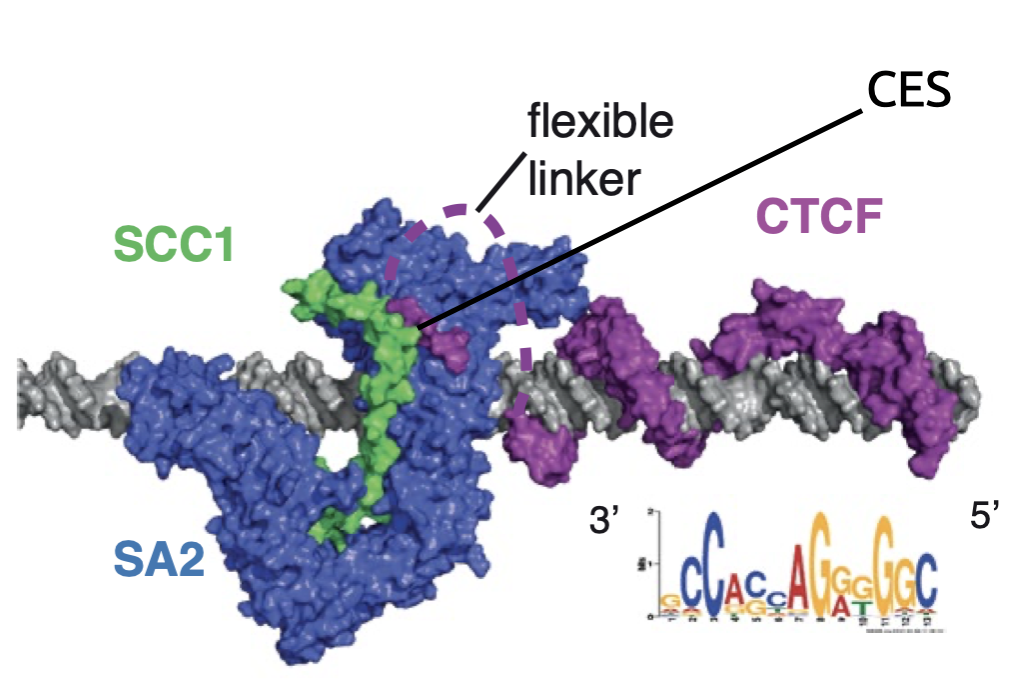


когезин - выпетливающий фактор
CTCF - граничный элемент
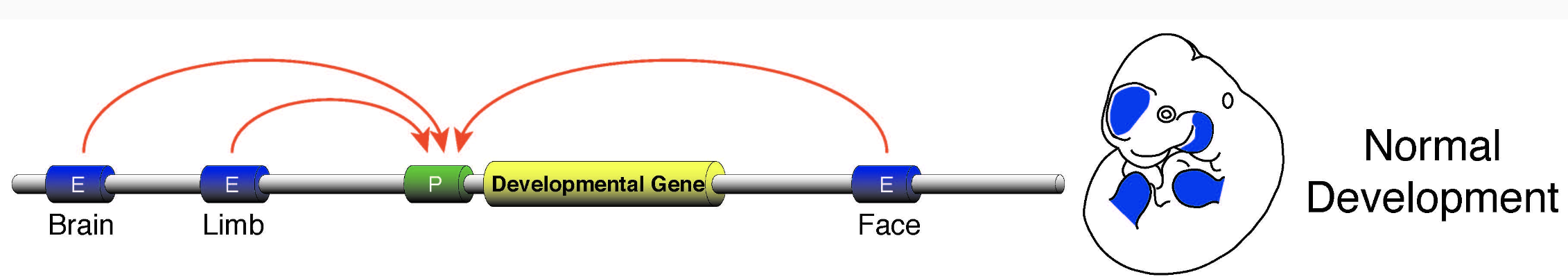
Клинические примеры
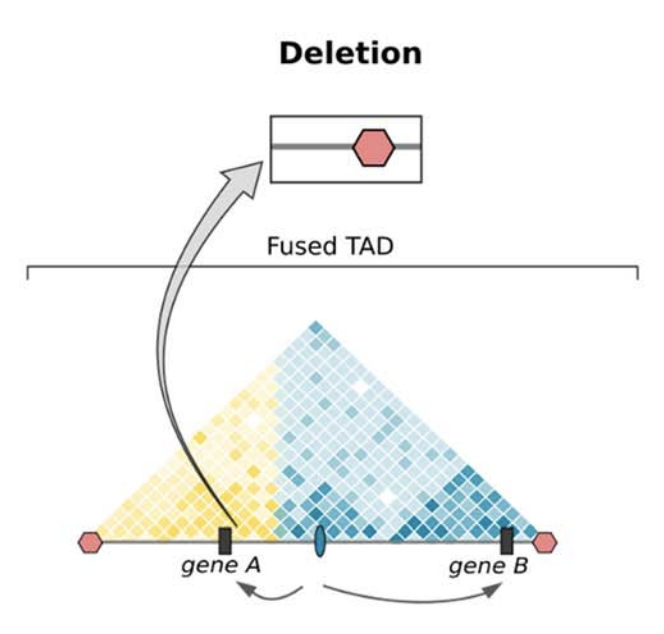
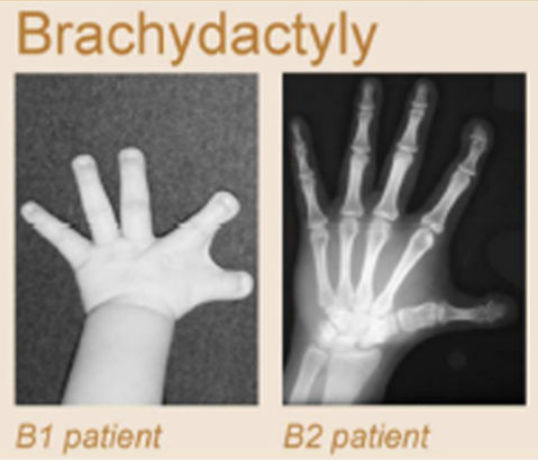
Anania and Lupiáñez, 2020; Lupiáñez et al. 2015
Брахидактилия - генетическое врожденное заболевания, патология формирование кисти
Клинические примеры
Anania and Lupiáñez, 2020; Lupiáñez et al. 2015

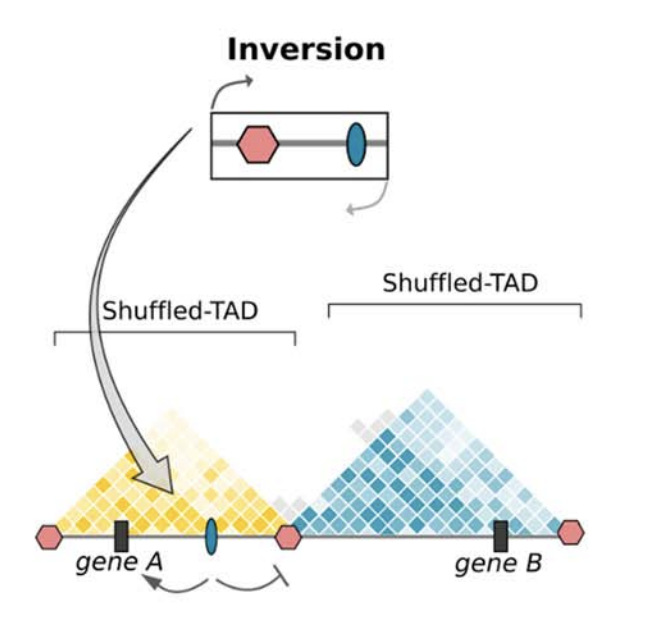
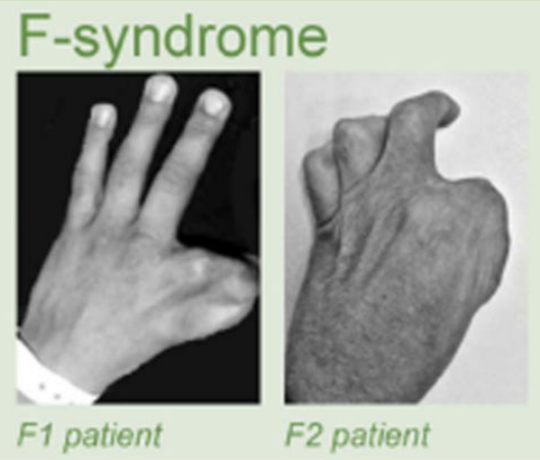
F-рука
Клинические примеры

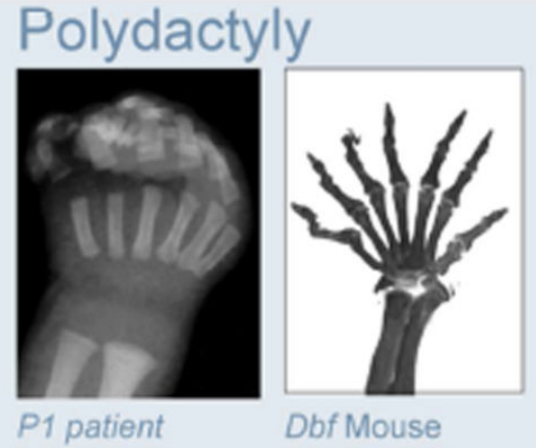
Anania and Lupiáñez, 2020; Lupiáñez et al. 2015
Полидактилия

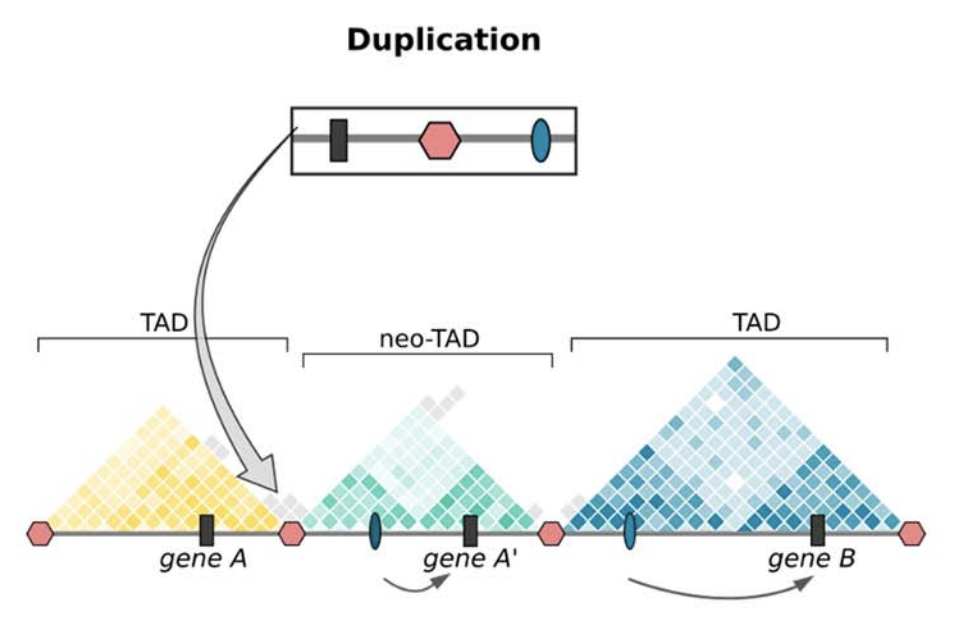
Несколько петель
"Hi-C" карта
3D-структура

Домен (ТАД)

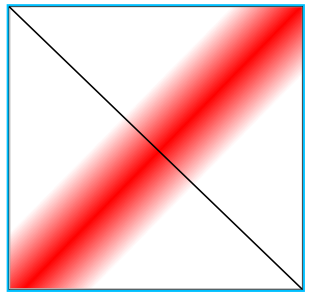
Шпилька
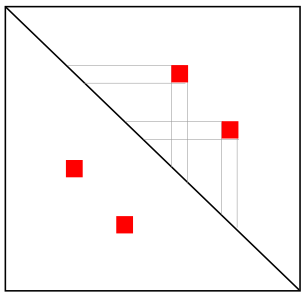
Псевдоузел (практически не встречается)
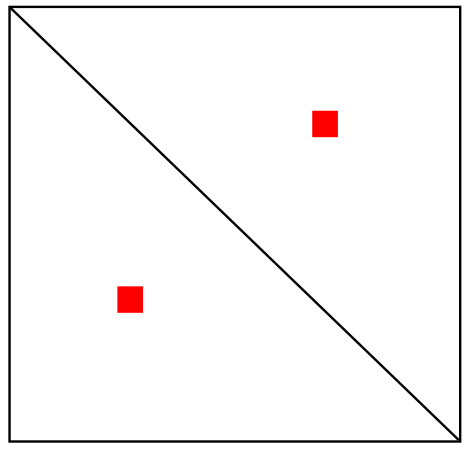

Соответствие?
Одиночная
петля
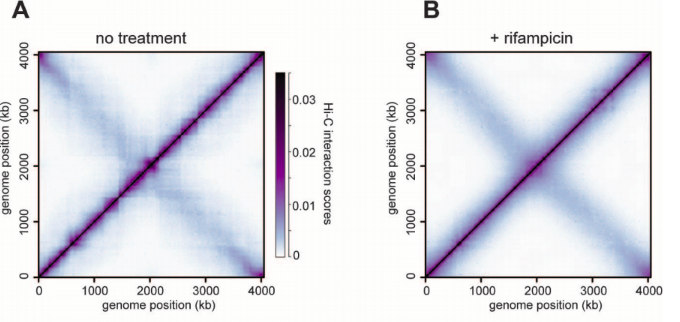
Настоящие шпильки хроматина
Caulobacter crescentus
(бактерия)

Настоящие шпильки хроматина
Caulobacter crescentus
Человек: 2 м ДНК в 10 мкм ядро
100-этажный дом в рисовое зерно

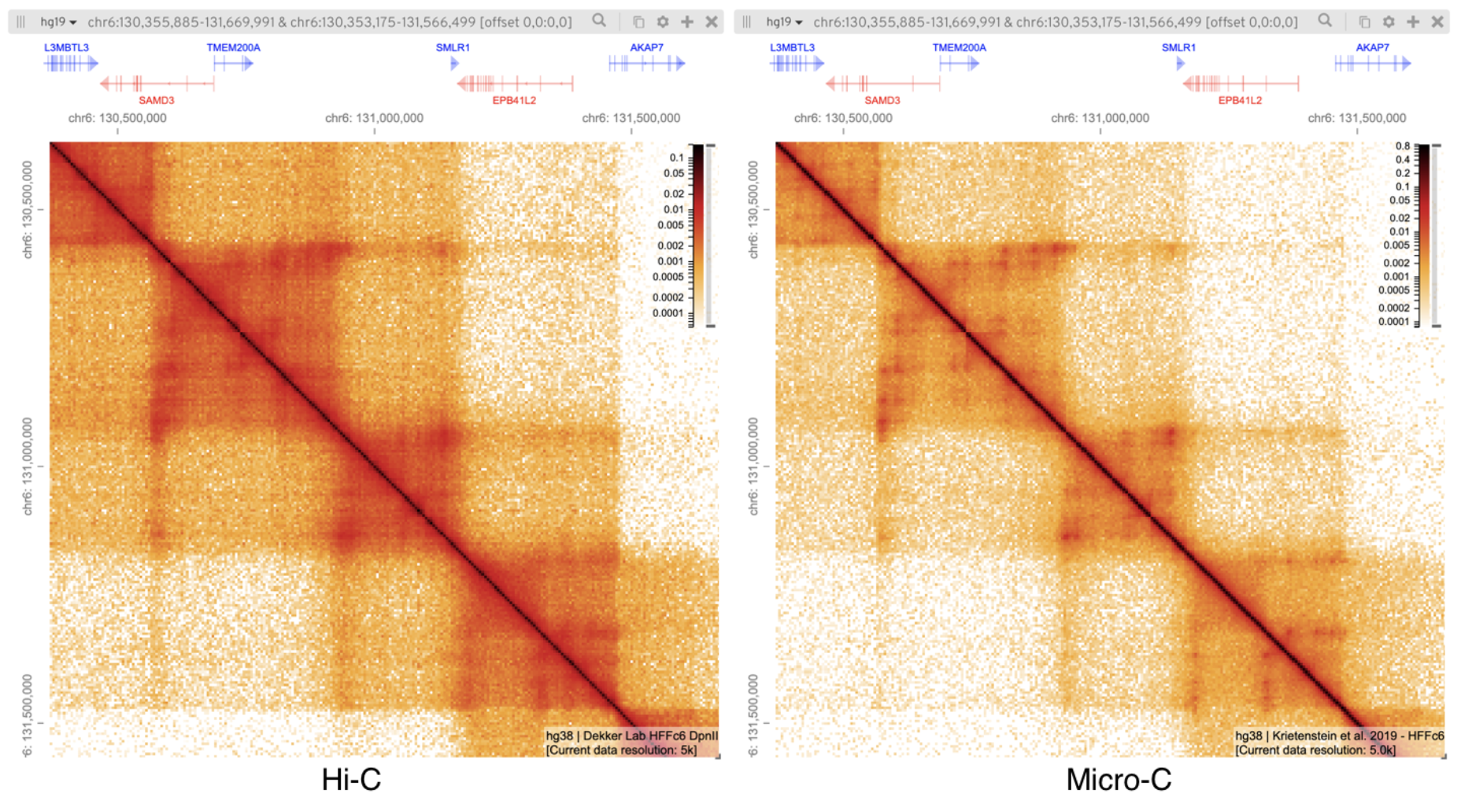
Другие методы исследования
структуры хроматина
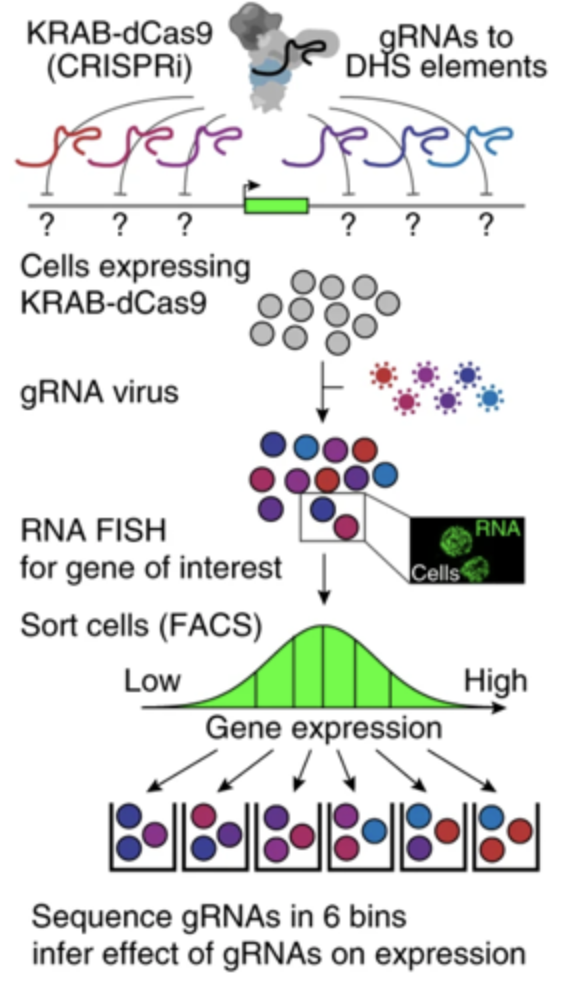
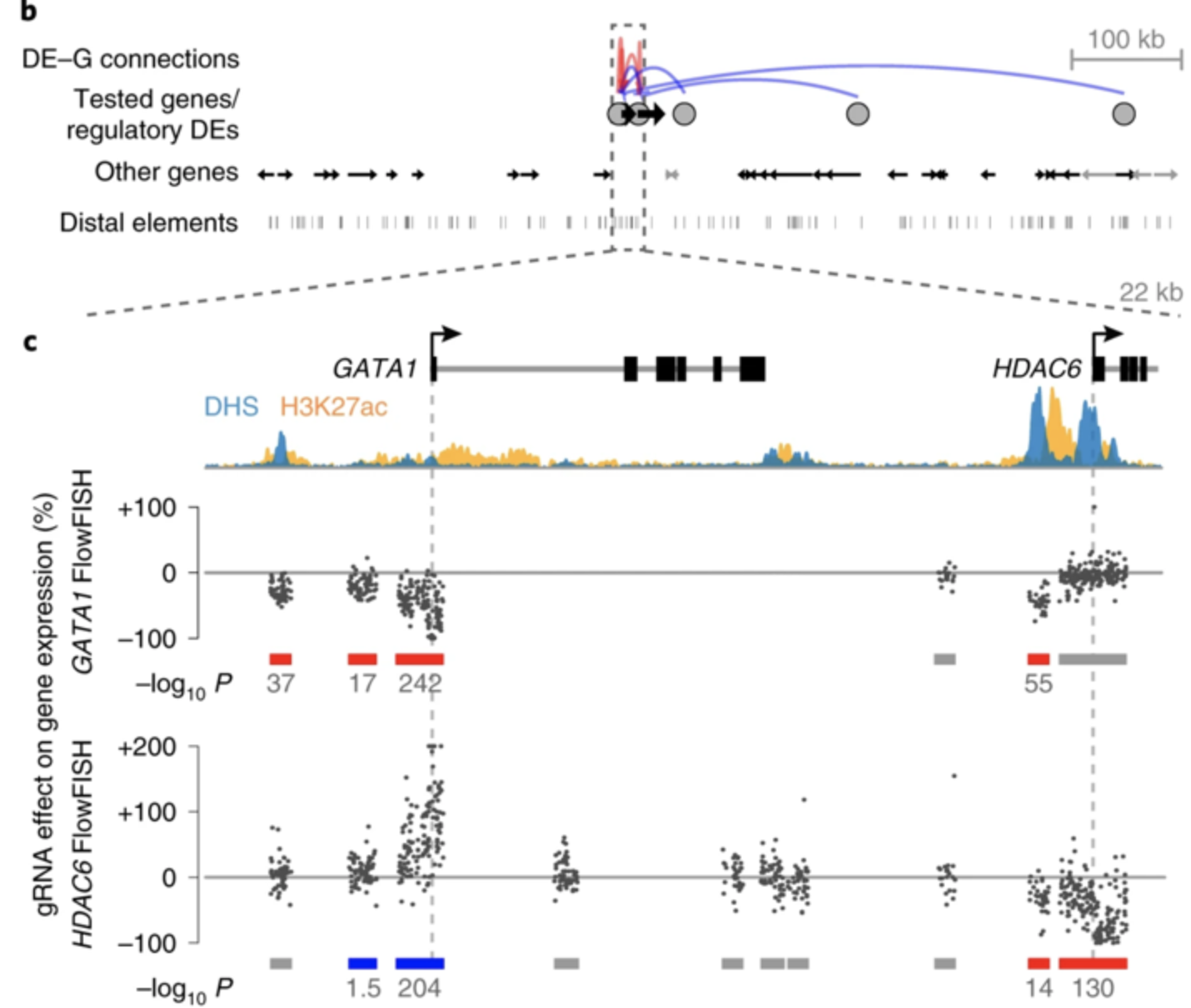
Fulco et al. 2019
In situ метод поиска
промотор-энхансерных взаимодействий:
CRISPRi-FlowFISH
Beagrie et al. 2017
Genome Architecture Mapping
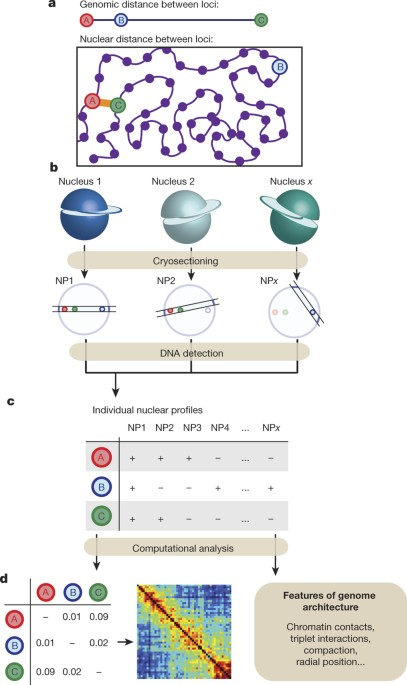
Nguyen et al. 2020
In situ sequencing approach
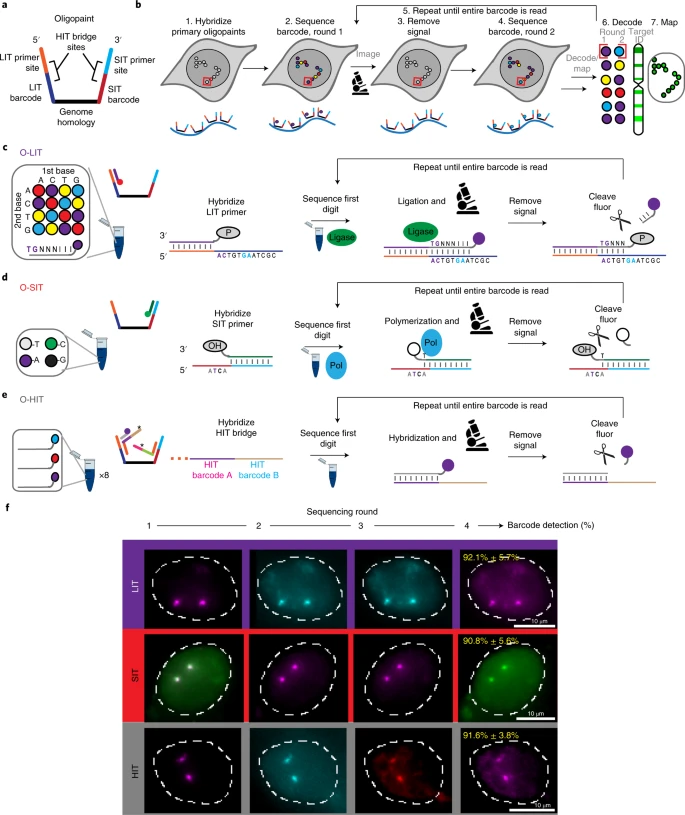
Hi-C - это МНОГО данных
Статистика депозита данных Hi-C в публичную базу SRA:
By Geoffrey Fudenberg for Open2C talk at Keystone Symposia 2021
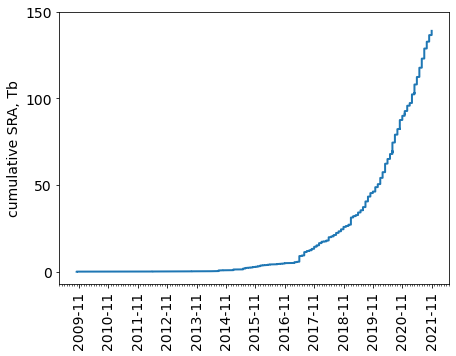
Hi-C - это МНОГО данных
Немного простой математики (для одного эксперимента Hi-C)
длина ДНК клетки человека:
~2 м
National Human Genome Research Institute www.genome.gov
Hi-C - это МНОГО данных
длина ДНК клетки человека:
~2 м
длина генома человека:
National Human Genome Research Institute www.genome.gov
~1 м
Немного простой математики (для одного эксперимента Hi-C)
Hi-C - это МНОГО данных
длина ДНК клетки человека:
размер генома человека:
количество всевозможных попарных контактов ДНК:
в 100Kb-бинах
в нуклеотидах
длина ДНК клетки человека:
~2 м
длина генома человека:
~1 м
размер одного нуклеотида = 0.34 нм = 0.00000000034 м
~3000000000 нт
3000000000·3000000000 =
9000000000000000000 нт
компьютерное представление:
~10 Эксабайт
(19 нулей)
Немного простой математики (для одного эксперимента Hi-C)
Hi-C - это МНОГО данных
Немного простой математики (для одного эксперимента Hi-C)
длина ДНК клетки человека:
длина ДНК клетки человека:
~2 м
длина генома человека:
~1 м
~ 1 Гигабайт
размер генома человека:
количество всевозможных попарных контактов ДНК:
в 100Kb-бинах
в нуклеотидах
~3000000000 нт
30000·30000 =
900000000 нт
компьютерное представление:
~10 Эксабайт
(19 нулей)
~30000 бинов
3000000000·3000000000 =
9000000000000000000 нт
Подходы для хранения Hi-C
Варианты хранения:
- плотная матрица (dense)
- разреженная матрица (sparse)

игрушечный пример:
[[2,0,1], [0,2,0], [1,0,2]]
left right n_contacts 1 1 2 1 3 1 2 2 2 3 3 2
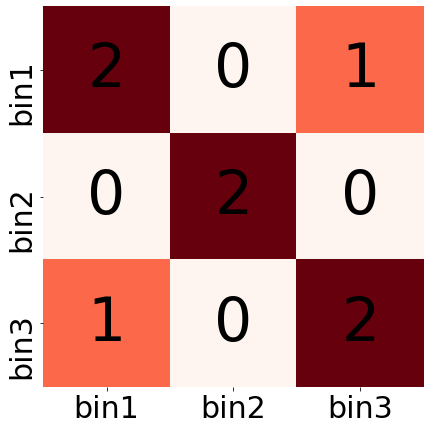
Подходы для хранения Hi-C
Варианты хранения:
- плотная матрица
- разреженная матрица .cool файл

игрушечный пример:
[[2,0,1], [0,2,0], [1,0,2]]
left right n_contacts 1 1 2 1 3 1 2 2 2 3 3 2


Подходы для обработки данных о взаимодействии ДНК
- Выбрать только заведомо важные контакты



+
- точечные контакты промоторов и энхансеров
Подходы для обработки данных о взаимодействии ДНК
- Выбрать только заведомо важные контакты

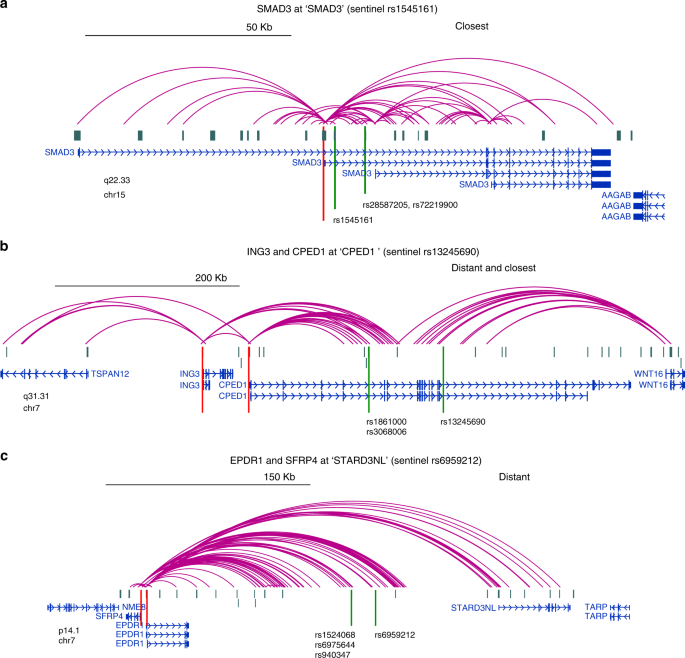
Chesi et al. 2019
- точечные контакты промоторов и энхансеров
- на уровне экспериментальной техники: Capture C
Подходы для обработки данных о взаимодействии ДНК
- Выбрать только заведомо важные контакты
2. Анализировать особенности (фичи, features) данных Hi-C

- точечные контакты промоторов и энхансеров
- на уровне экспериментальной техники: Capture C




}
Особенности структуры хроматина
Fraser et al. Microbiology and Molecular Biology Reviews 2015

Хромосомные территории
Bonev et al. 2016 Nature Reviews



Хромосомные территории
Falk et al. Nature 2019
^^^ Сделали усреднение ^^^
получили средние контакты между парами хромосомам
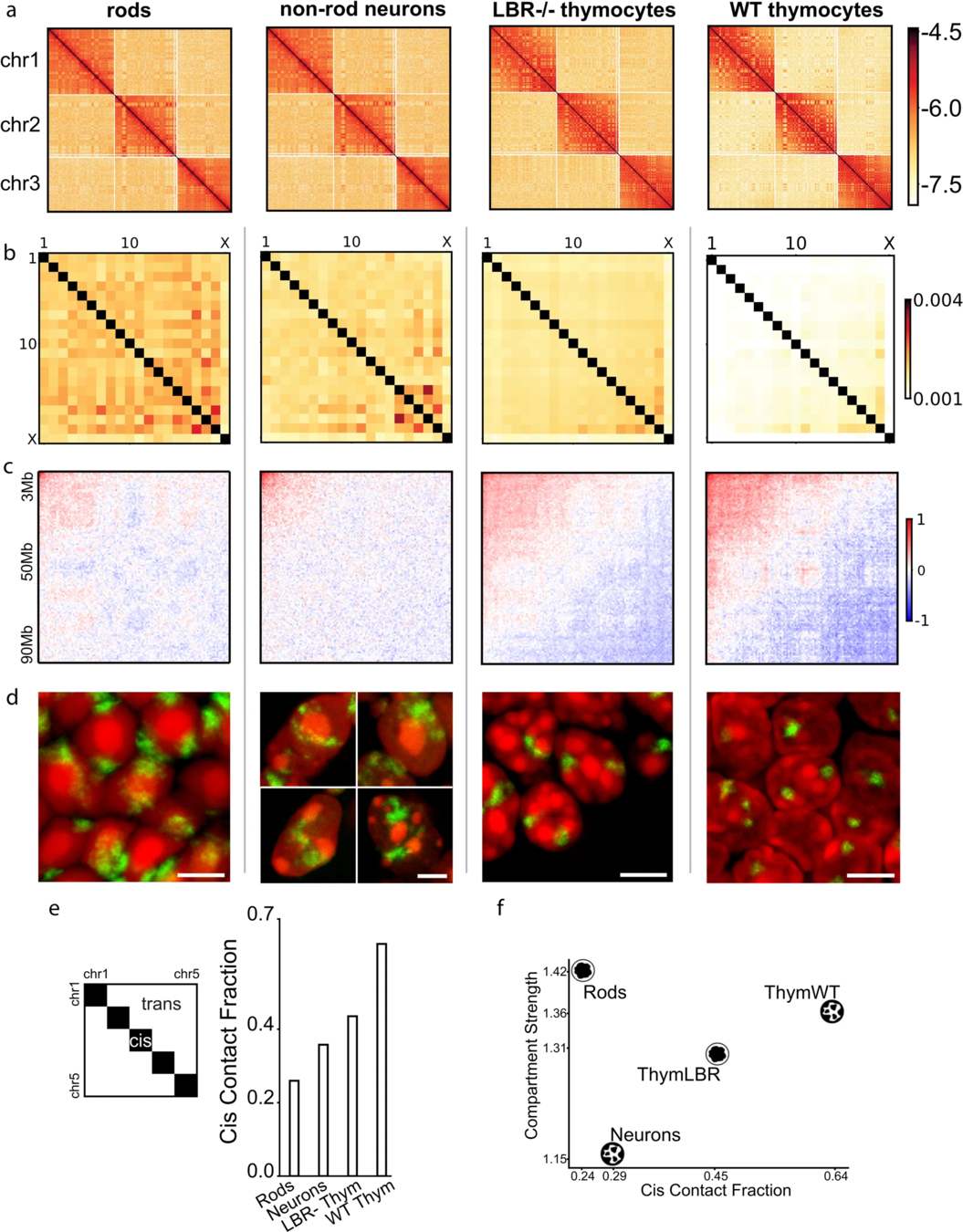
Hi-C карты для трех хромосом:
Топологически ассоциированные домены (ТАДы)


Bonev et al. 2016 Nature Reviews
одна из гипотетических моделей:
ТАДы сложнее
Filippova et al. Algorithms for Molecular Biology 2014
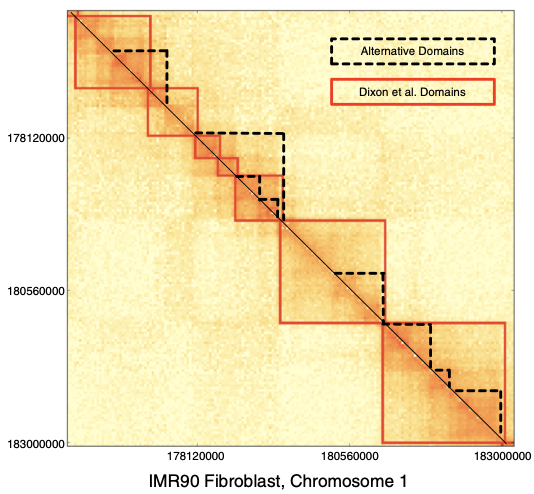
чем "квадраты" вдоль диагонали:
- Решается с помощью динамического программирования, например,
Armatus
Много способов искать ТАДы...
Forcato et al. Nature Methods 2017
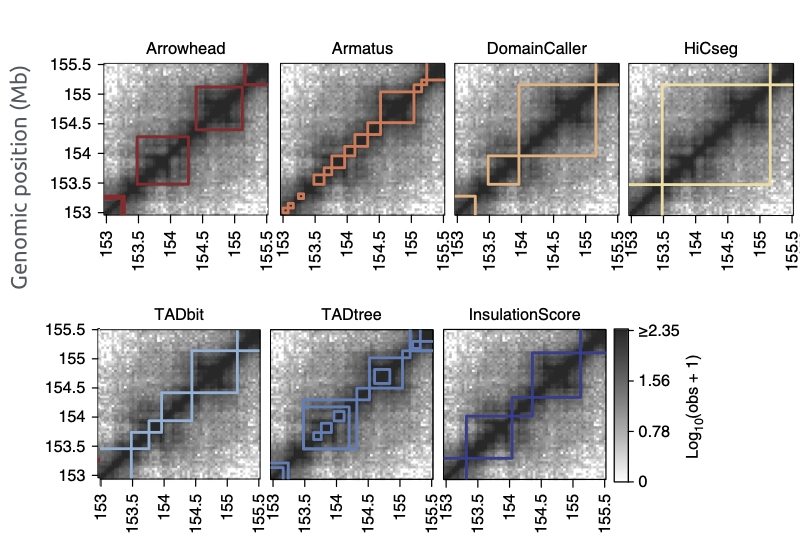
Разберем пример поиска ТАДов
based on Crane, 2015
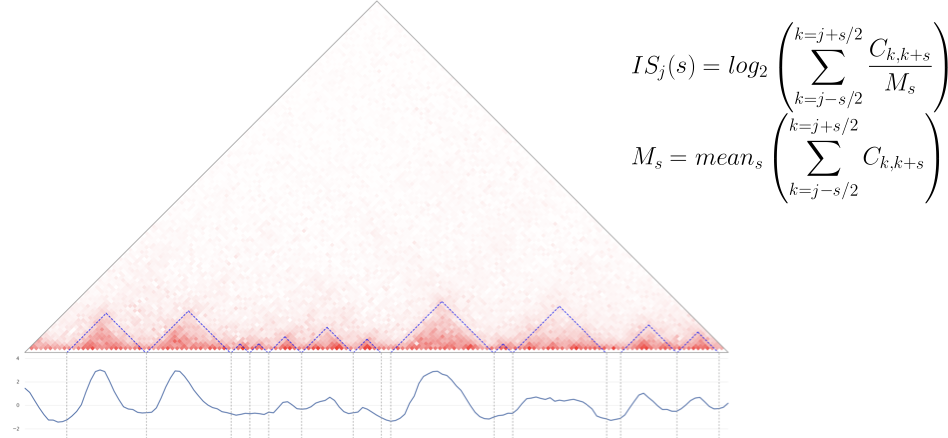
- Показатель инсуляции - один из простейших подходов:
- Подсчитать меру инсуляции для каждого бина,
- Найти локальные минимумы:
Инструмент для Python: cooltools insulation with tutorial
Стоит ли искать ТАДы?
- Использование меры Gamma transitional (близкая к IS)
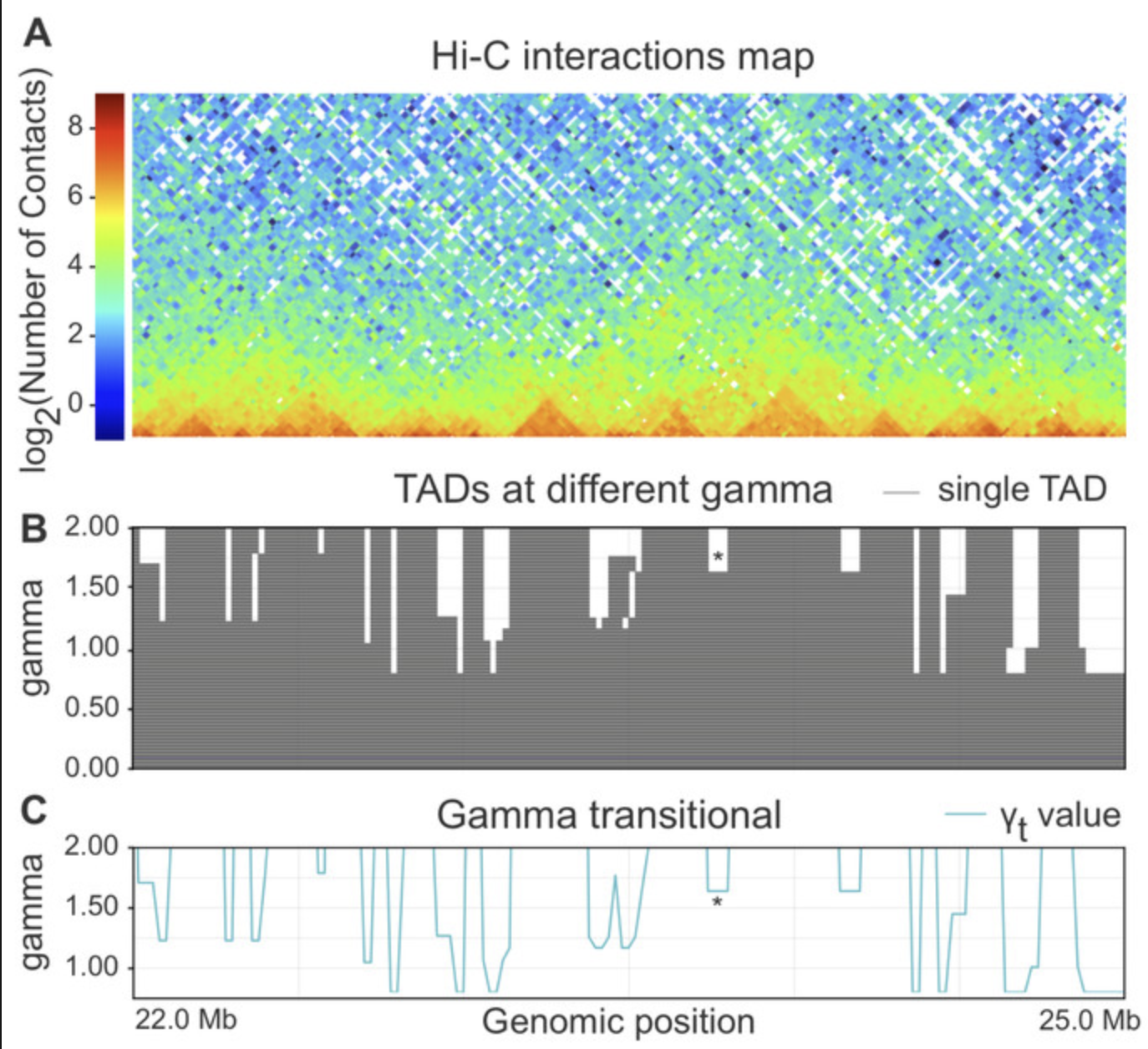
1. для каждого бина генома получаем
некоторую меру инсуляции
2. берем набор эпигенетических данных
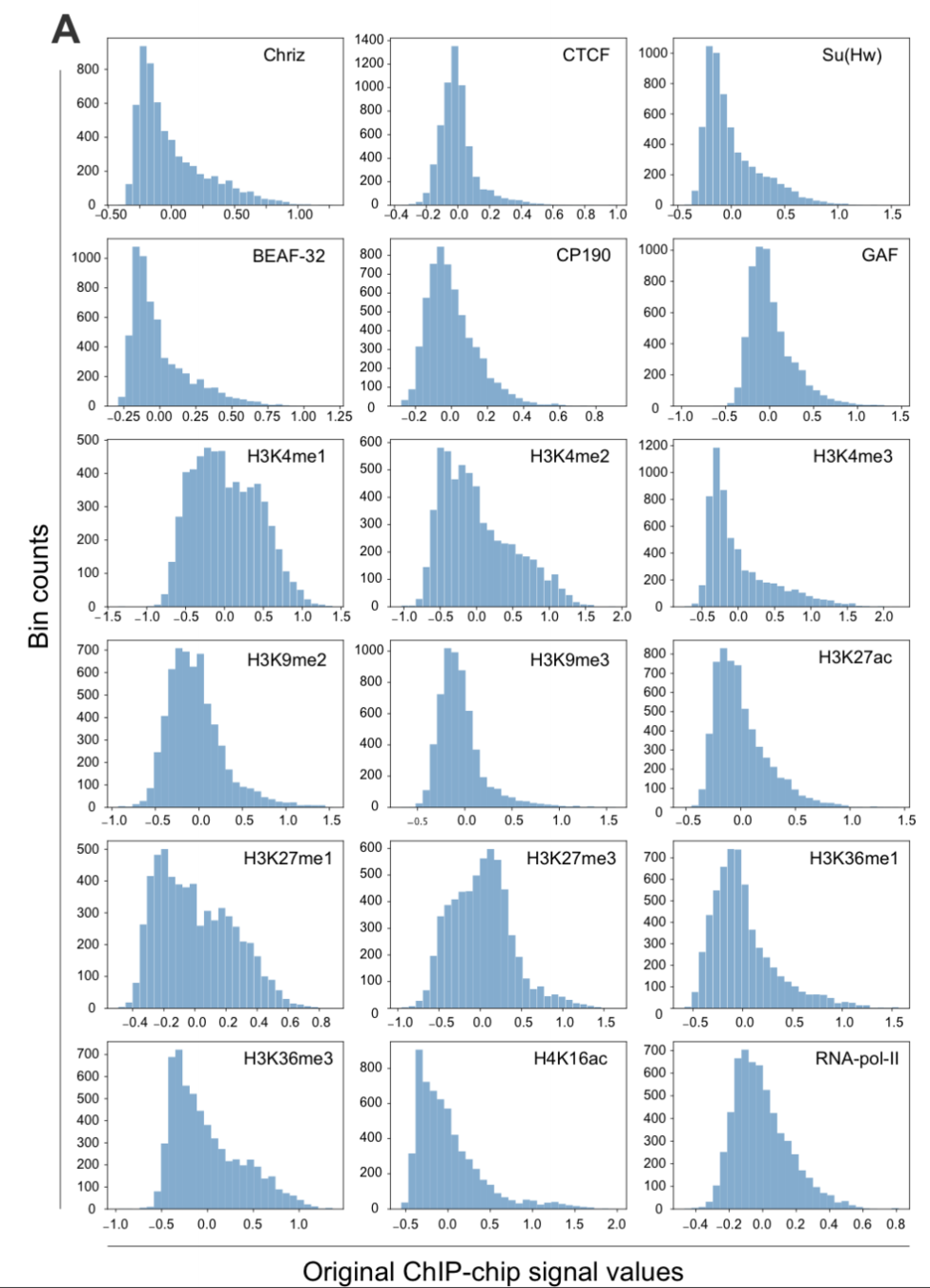
Стоит ли искать ТАДы?
- Использование меры Gamma transitional (близкая к IS)
3. подбираем и обучаем модель машинного обучения
(в данном случае biLSTM):
4. получаем ранжирование эпигенетических факторов по важности для формирования структуры хроматина:
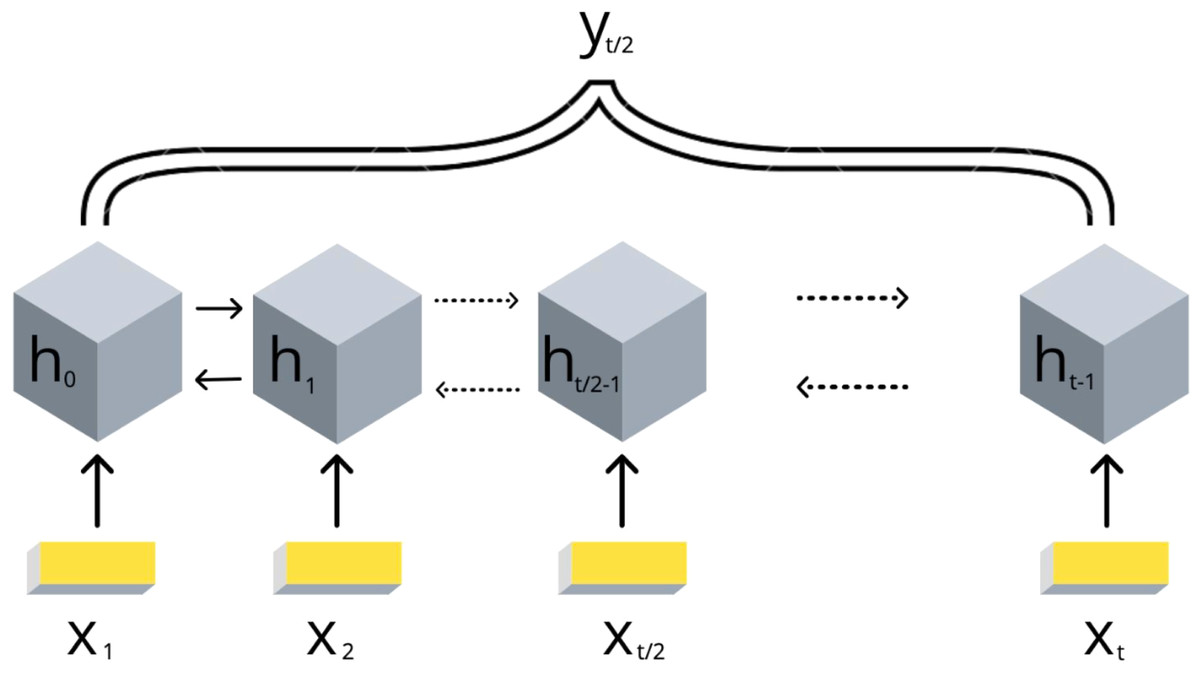
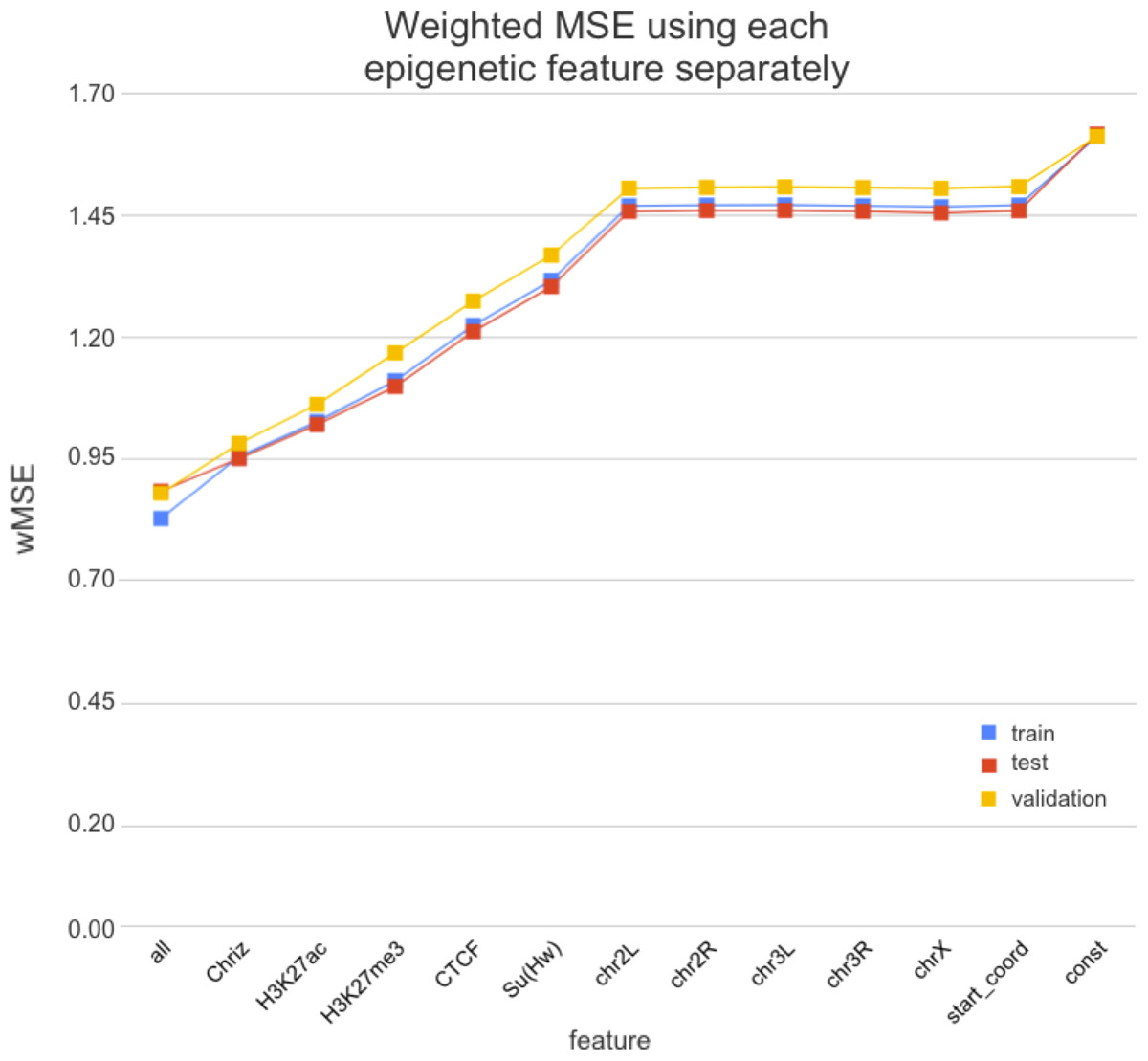
Стоит ли искать ТАДы?
Wang et al. 2018
5. Позиционирование факторов предсказывает границы ТАДов:
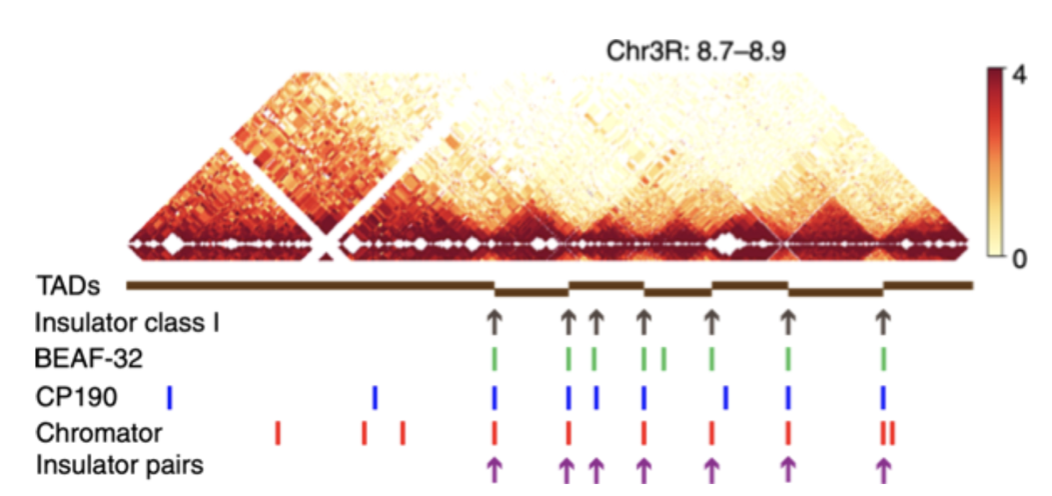
Компартменты


Bonev et al. 2016 Nature Reviews
одна из гипотетических моделей:
Вспоминаем: графики P(s)
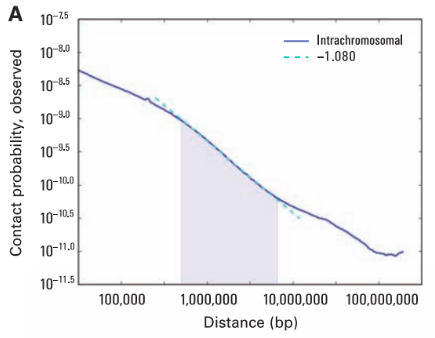
Lieberman-Aiden, 2009
Зависимость частоты контактов от геномного расстояния:
Алгоритм поиска компартментов
Lieberman-Aiden et al. Nature 2009
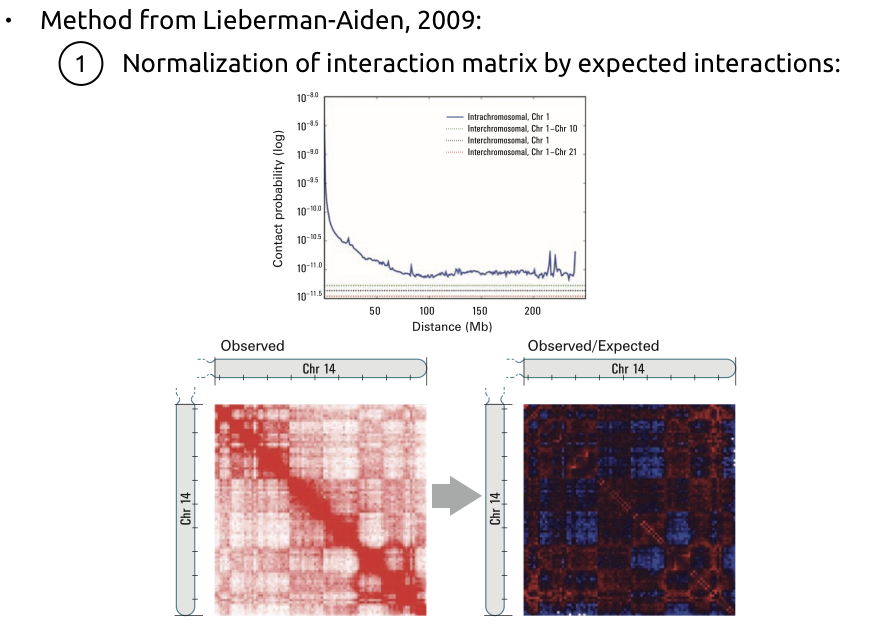
1. Нормализация матрицы контактов на P(s):
Алгоритм поиска компартментов
Lieberman-Aiden et al. Nature 2009
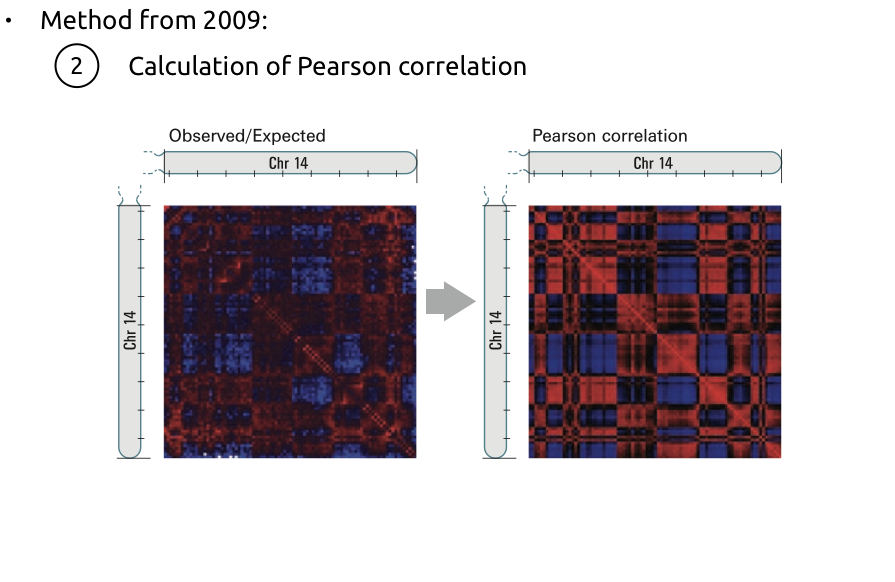
2. Усиление контрастности:
Алгоритм поиска компартментов
Lieberman-Aiden et al. Nature 2009
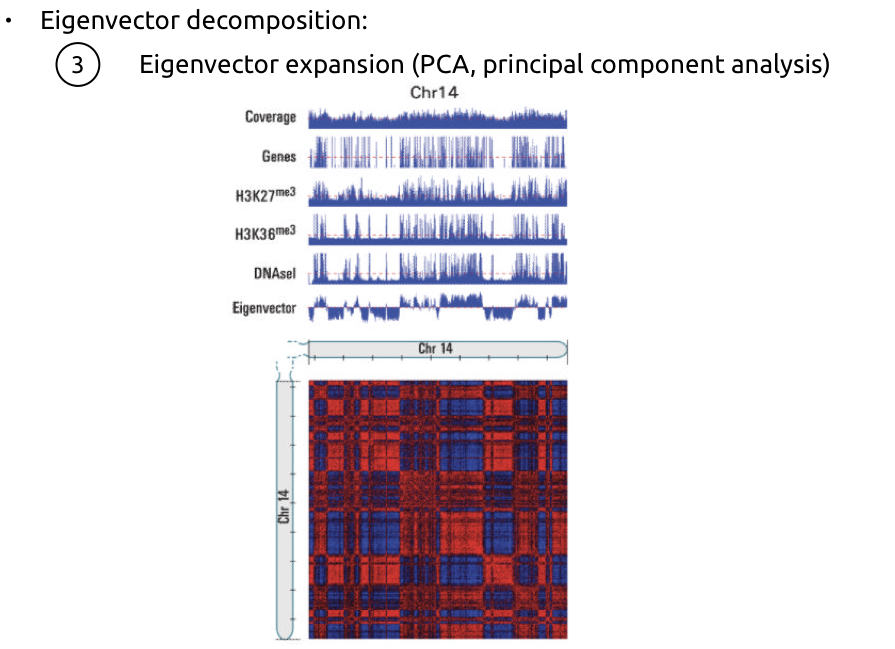
2. Оценка сходства между каждой парой строк:
(очень упрощая)
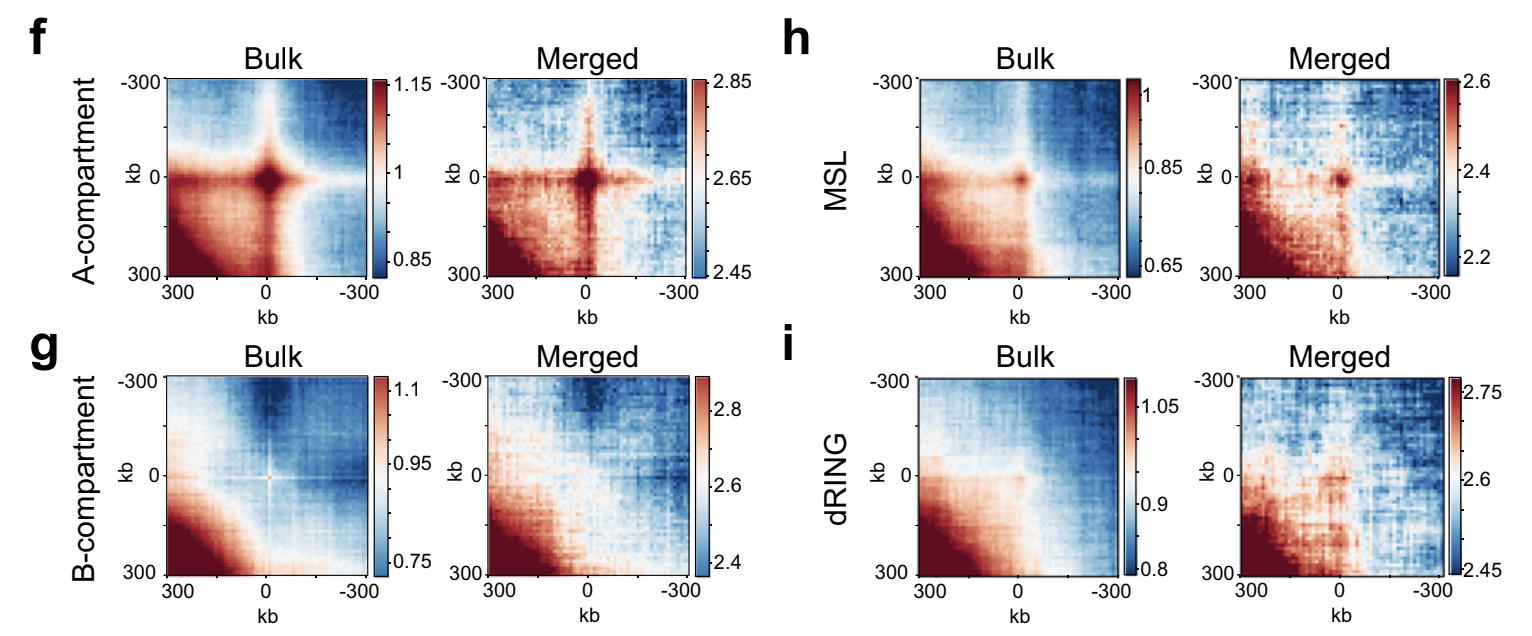
Оценка выраженности компартментов
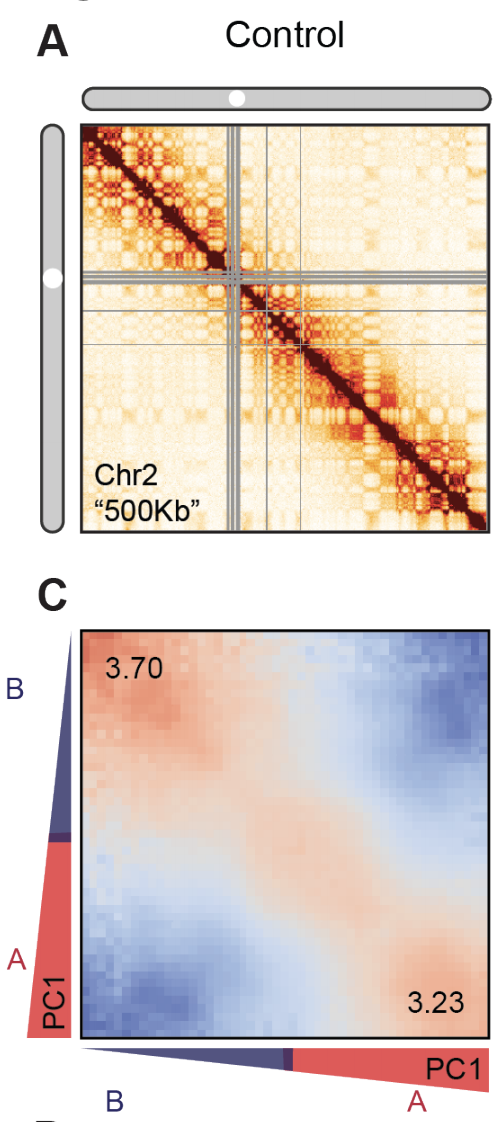

чем выше сигнал по углам карты,
тем более выражены компартменты
1. Найдем в Hi-C компартменты,
2. Нормируем Hi-C на P(s),
3. Переставим местами строки так, чтобы похожие были рядом,
4. Огрубим карту с помощью усреднения соседних пикселей.
Инструмент для Python: cooltools compartments with tutorial
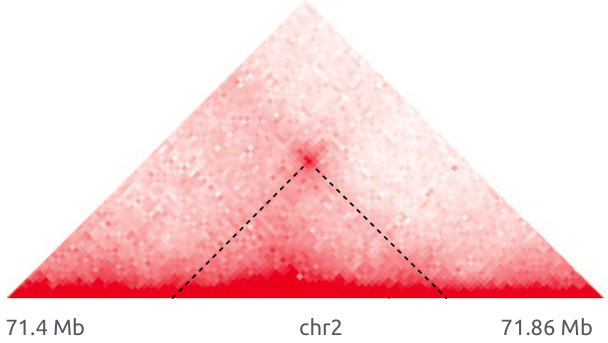
Обогащенные взаимодействия (петли)




Архитектурные петли
Промотор-энхансерные взаимодействия
Polycomb-петли
Bonev et al. 2016 Nature Reviews
Алгоритм поиска петель: HiCCUPS
Rao et al. Cell 2014
Hi-C Computational Unbiased Peak Search
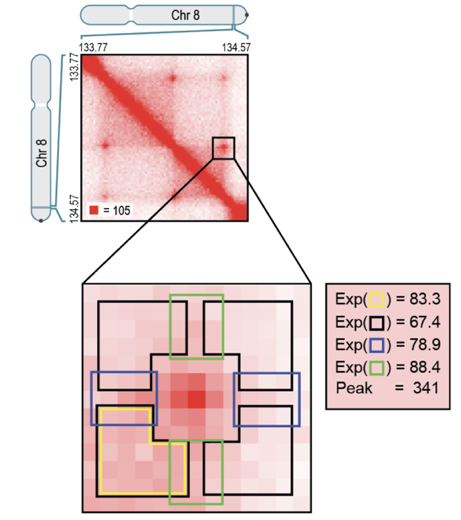
Свойства петель хроматина
Rao et al. Cell 2014
Позиции петель млекопитающих часто содержат мотив связывания фактора CTCF:
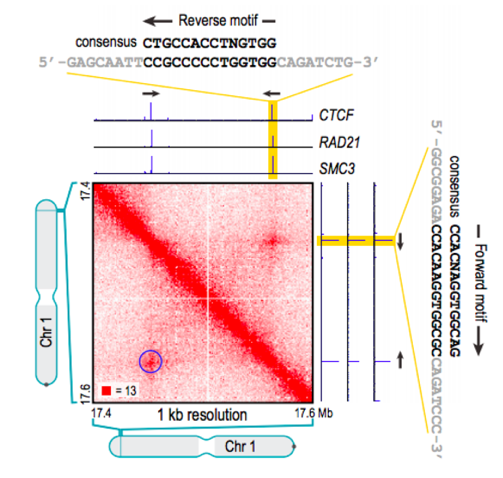
Разнообразие алгоритмов поиска петель
Forcato et al. Nature Methods 2017
Огромное количество алгоритмов, как и в случае поиска ТАДов:
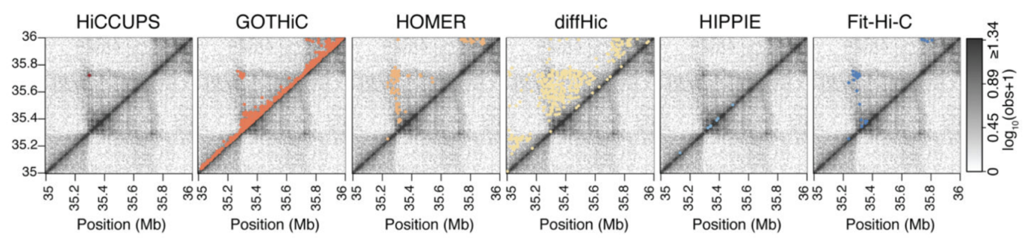
Всегда ли нужно искать петли?
Flyamer Bioinformatics 2019
Можем взять все позиции связывания CTCF и построить среднюю картину петли:
https://github.com/Phlya/coolpuppy


позиции CTCF
средняя петля
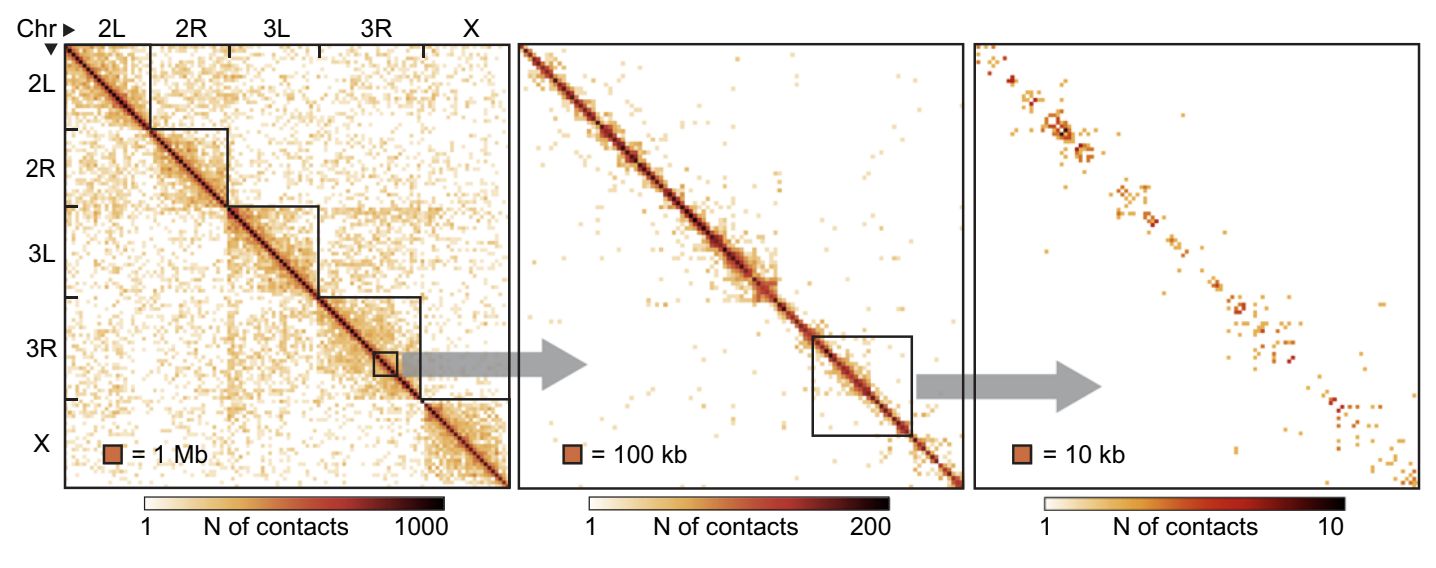
Шаг в сторону: single-cell Hi-C

Flyamer et al. Nature 2017

Single-cell Hi-C - протокол, похожий на Hi-C, но для единичных клеток:
Усреднение - мощный инструмент
Ulyanov, Galitsyna et al. 2021, Nature Communications
На примере single-cell Hi-C данных мухи, очень мало данных:
Письменное домашнее задание
Работа с браузерами Hi-C: Juicebox
Разбираемся с петлями и ТАДами с помощью Juicebox:
https://aidenlab.org/juicebox/
1. Загрузите данные для клеточной линии GM12878 из Juicebox Archive.
2. Приблизьте карту на регион одной хромосомы. Поменяйте цветовую шкалу при необходимости
3. Найдите хотя бы одну петлю и хотя бы один ТАД.
Какое разрешение подходит для их визуализации?
4. Загрузите 2D аннотации (петли и домены типа Combined).
Как связаны между собой разметки петель и ТАДов?
5. Добавьте данные RNA-Seq. Как они соотносятся с позициями петель и ТАДов?
Работа с браузерами Hi-C: HiGlass
Разбираемся с разными экспериментами через HiGlass:
1. Перейдите на Two Linked View раздел.
Какие типы клеток перед вами?
2. Одинаковая ли структура хроматина в заданном регионе?
3. В каких типах клеток (из двух) гены OXR1, ABRA, ANGPT1 находятся в "активном" хроматине?
4. Какие структуры хроматина ассоциированы с активными генами?
Вопрос: Используя информацию из литературы и публичных баз данных, как можно объяснить дифференциальную активность генов OXR1, ABRA, ANGPT1 между двумя приведенными клеточными типами?
Ссылка на форму для развернутого ответа:
https://docs.google.com/forms/d/1F6N47Wu8MFJZ6_d8cHYsAZRWwKa3R6ps4gSPqbgEeYg
(ответы принимаются до 23:59 4 декабря)



