Bioinformática desde Cero


Un poco de Blackground sobre biología
hace falta ¿No?




Luego hay que elegir un curso que nos guste entre las decenas que hay
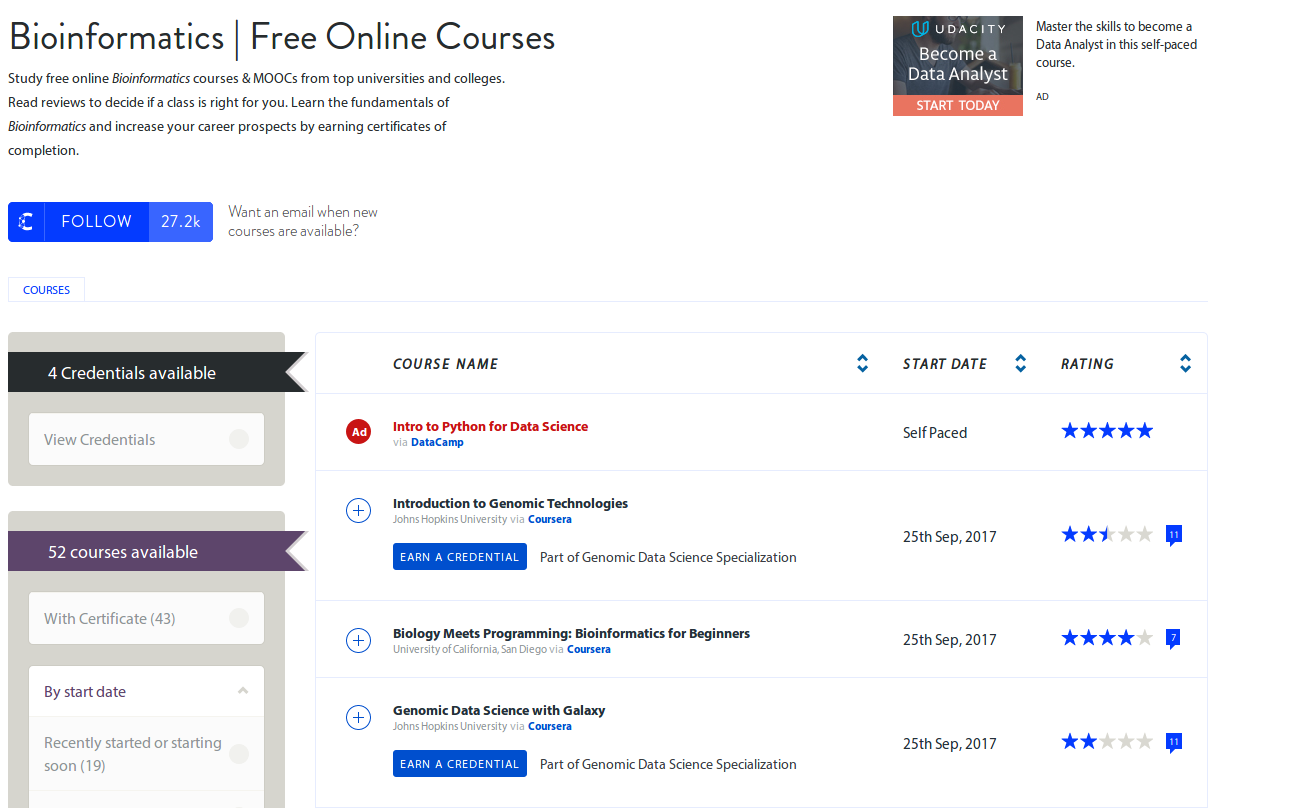
Mi elección, basada en mis gustos y usos como programador en Python, fue esta, aunque también hay muchos otros cursos para Java, R, etc ...
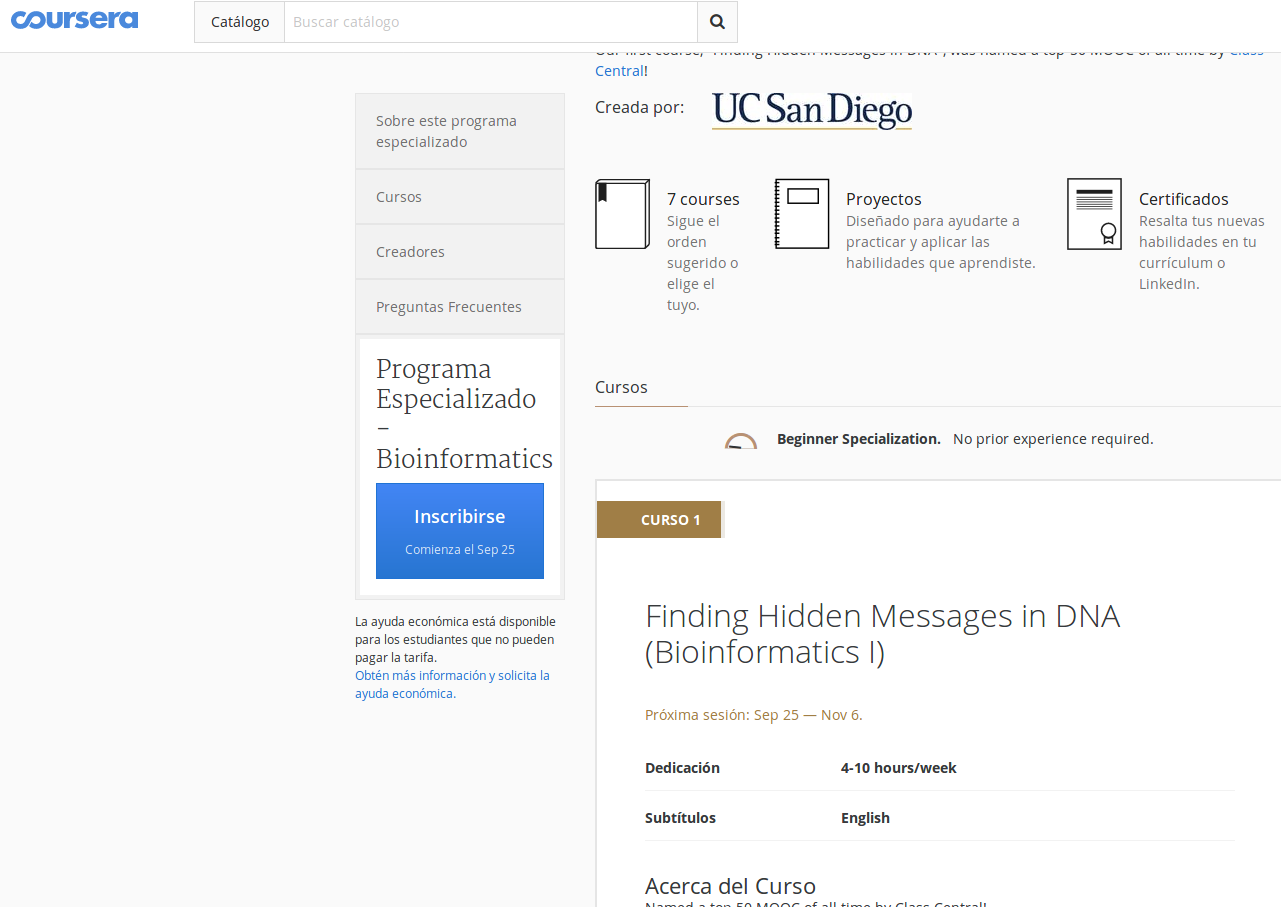
Programa especializado de Coursera de 7 MOOCs sobre BioInformática con Python
El MOOC en concreto sobre el que voy a hablar es ...
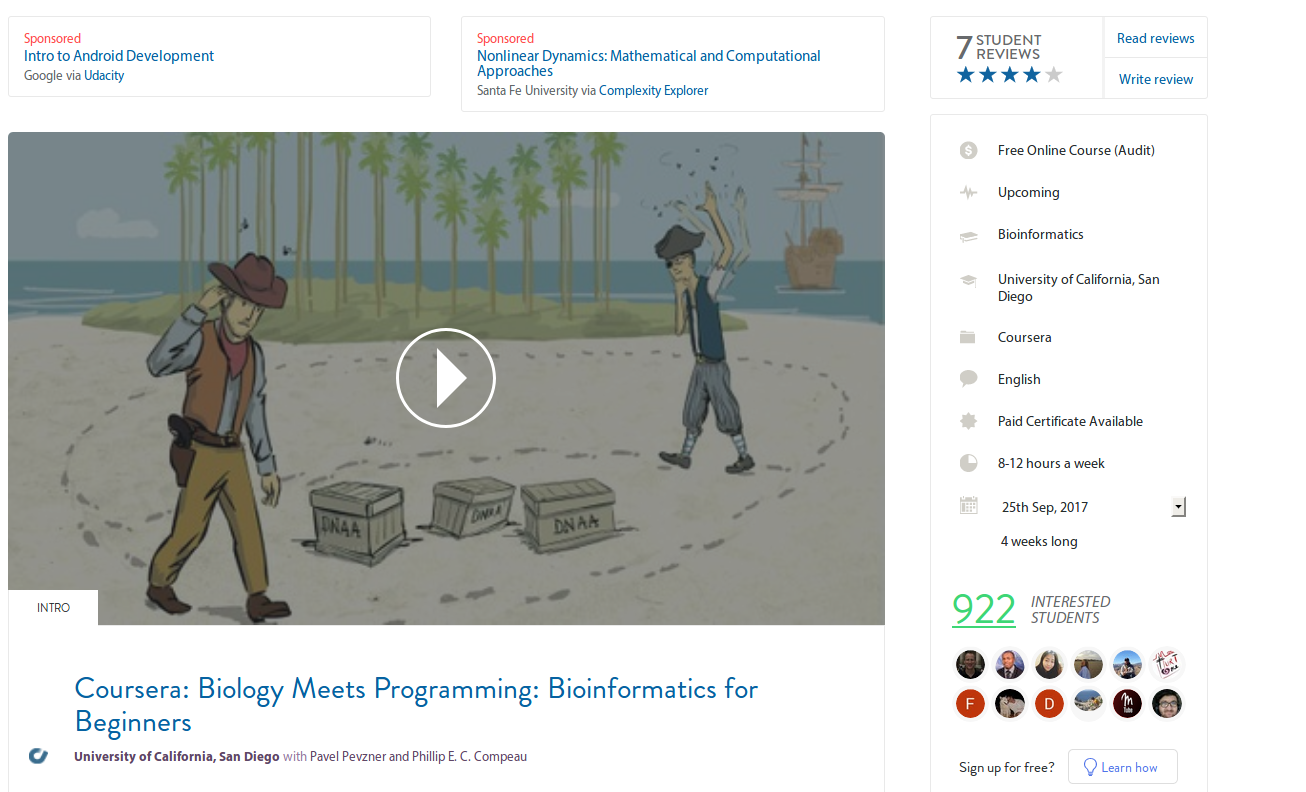
Los Apuntes del Curso en formato Notebook de Jupyter se pueden encontrar en el Github
de vencejo
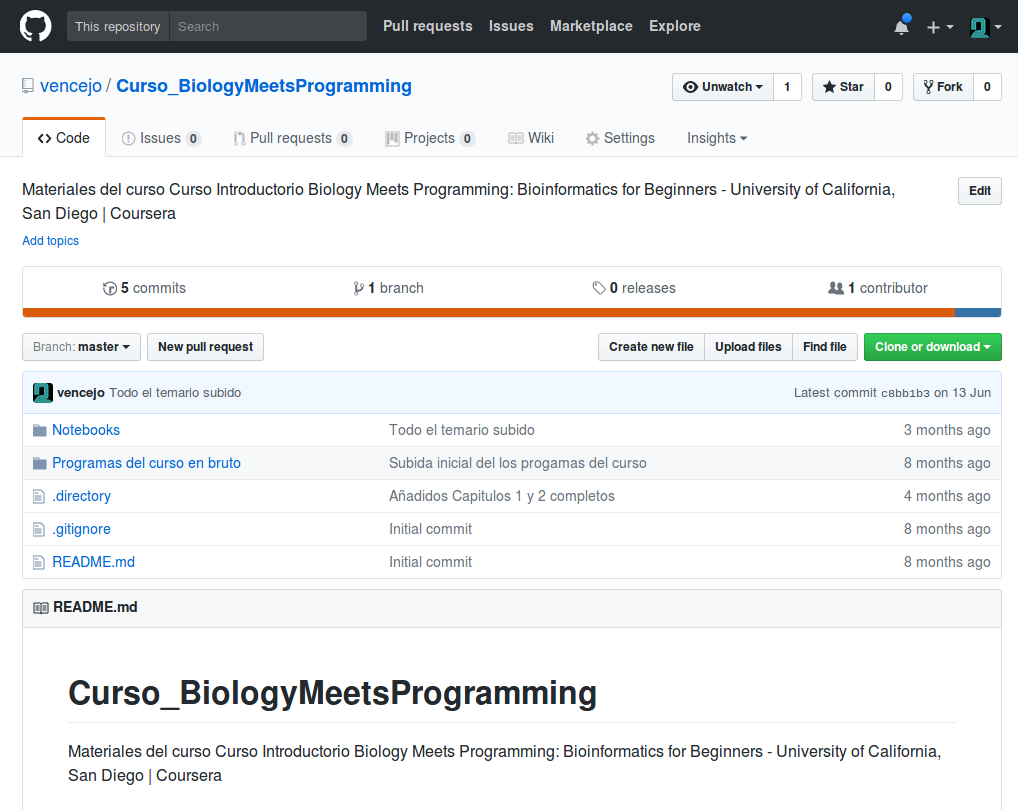
Indice del Curso

El Curso se estructura para dar respuesta a dos grandes problemas
-
Búsqueda de las regiones ORi
-
Búsqueda de los lugares de enganche de las proteínas reguladoras (Motifs)
Primer problema del curso
-
Búsqueda de las regiones ORi
En el primero de los 4 módulos del curso se trabaja buscando las regiones de ADN donde se inicia la replicación (Zonas Ori)

¿Que es lo que identifica los ORi? Dada una cadena de ADN, ¿Como podemos saber si hay un ORi en ella?
Un primer vistazo a como se complementan la MicroBiología tradicional con la BioInformática

Para los nobeles totales en programación:
Curso paralelo en Codecademy

Caso práctico de Ori en Vibrio Cholerae

El curso tiene una curva de aprendizaje suave, además los programas son muy "de abajo a arriba" , muy modulares, aquí por ejemplo está la primera función que cuenta palabras en el ADN


Los k-mer: Un k-mer (camero?) es la palabra de longitud k que mas veces aparece en una cadena dada de ADN

Los k-mer en el ORi de Vibrio Cholerae

Segundo problema del curso
Búsqueda de los lugares de enganche de las proteínas reguladoras (Motifs)
El problema de la búsqueda de los lugares de enganche de las proteínas reguladoras
(Regulatory Motifs o Transcription factor binding site)

Lugares de enganche de las proteínas reguladoras
(Regulatory Motifs )
El ejemplo de la Tuberculosis

Lugares de enganche (Binding Sites)
de las proteínas reguladoras
(Regulatory Motifs) : Problema -> Hay muchas formas operativas del mismo Motif

Seria fácil encontrar los lugares de enganche de las proteínas reguladoras
(Regulatory Motifs) si estos tuviesen una forma única

El problema es que estos lugares de enganche de las proteínas reguladoras
(Regulatory Motifs) tienen formas equivalentes "mutadas" que se dan en la realidad

Para empezar a crear algoritmos que busquen Motifs, primero hay que tener formas de cuantificarlos
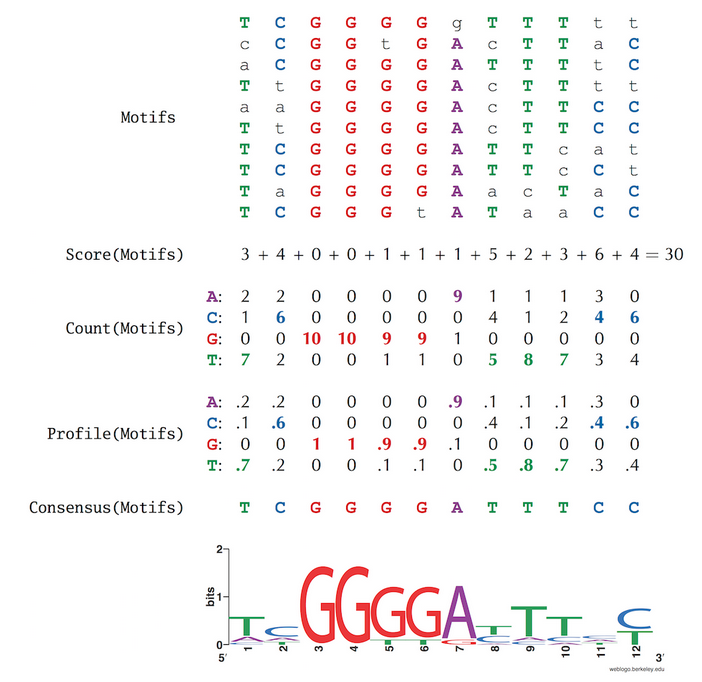
Después hay que definir de manera computacionalmente rigurosa el problema

Para luego darnos cuenta de que un enfoque de fuerza bruta es inviable

Un algoritmo bastante rudimentario, pero que sirve como una primera aproximación es el "avaricioso" que trabaja eligiendo siempre , en cada iteración, la opción que "maximiza la ganancia"

"Maximizar la ganancia" en este contexto consiste en ir eligiendo las opciones que maximicen su probabilidad dado un Motif determinado (En forma de Profile o matriz de probabilidades) , para luego actualizar dicho profile con la nueva información y volver a empezar el ciclo

El algoritmo avaricioso tiene unos resultados mediocres,pero es muy sensible a los valores iniciales y es muy rápido, lo que lo hace idoneo para aplicarle sucesivas fases de randomización que lo convierte en un algoritmo decente

Armado con estos algoritmos el curso nos permite aplicarlos en genomas ya conocidos como el de la Tuberculosis o la E.Coli
Un párrafo que resume mi opinión global sobre este MOOC
Si , en un primer curso introductorio, se han tratado problemas tan apasionantes como estos, de una manera tan amena y efectiva ,
¿Que nos pueden deparar los restantes siete cursos de los que se compone el track de BioInformática con Python de Coursera
Una reflexión final respecto al proceso de aprendizaje de las ciencias mediante una aproximación computacional


Muchas gracias por su atención,
¿alguna pregunta ?

