Uczenie maszynowe
w medycynie
Michał Bieroński, 218324
Mateusz Burniak, 218321
Agenda
-
Czym jest uczenie maszynowe?
- Kategorie
-
Algorytmy
- Regresja liniowa
- Drzewa decyzyjne
- Random Forest
- KNN
-
Zastosowania
- Obrazowanie siatkówki
- Rak skóry
- Rak piersi + demo
-
Ciekawostki
- Kaggle, YOLO, MarI/O, Muzyka
Uczenie maszynowe

Dziedziny zastosowania
- Usługi finansowe (oszutwa, analiza giełdy)
- Rząd (przestępstwa, analiza budżetu)
- Medycyna (diagnozowanie chorób)
- Marketing (spersonalizowane reklamy)
- Przemysł energetyczny (posz. nowych źr. energii)

Kategorie
- Nadzorowane (Supervised)
- Nienadzorowane (Unsupervised)
- Przez wzmacnianie (Reinforcement)
Uczenie nadzorowane
Znane są poprawne wartości dla danych uczących.
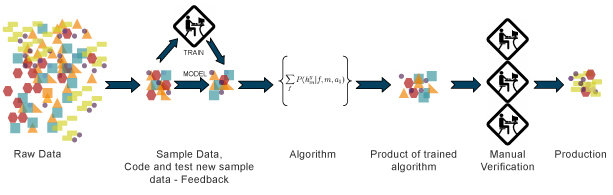
- regresja
- drzewa decyzyjne
- random forest
- KNN
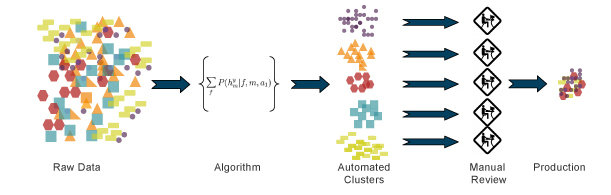
Uczenie nienadzorowane
Nie są znane dane wyjściowe.
Uczenie przez wzmacnianie
Metoda prób i błędów.
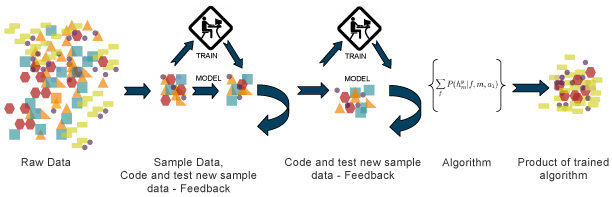
Algorytmy
Regresja liniowa
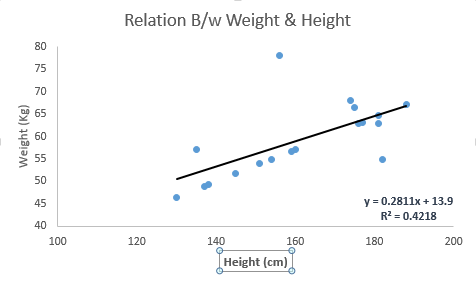

Szukamy funkcji opisującej zależność jednej zmiennej w zależności od innych zmiennych (często pozornie nie związanych ze sobą)
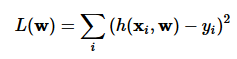
Drzewa decyzyjne
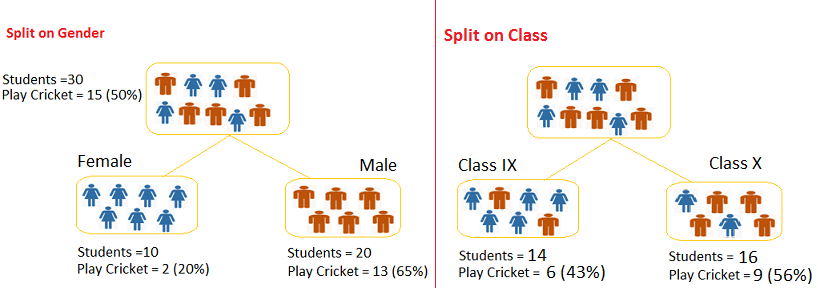
Podział danych względem
najbardziej znaczących atrybutów.

Drzewa decyzyjne
Random Forest
Połączenie wielu drzew decyzyjnych w multiklasyfikator.

KNN

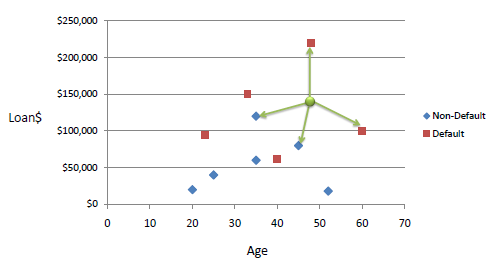
- Jeden z najprostszych klasyfikatorów
- Dobra jakość predykcji, lecz wolny
A teraz przykłady...
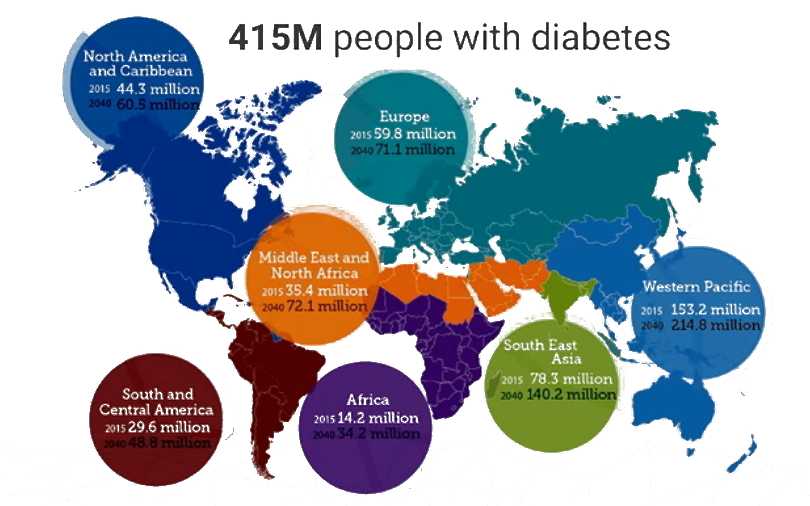
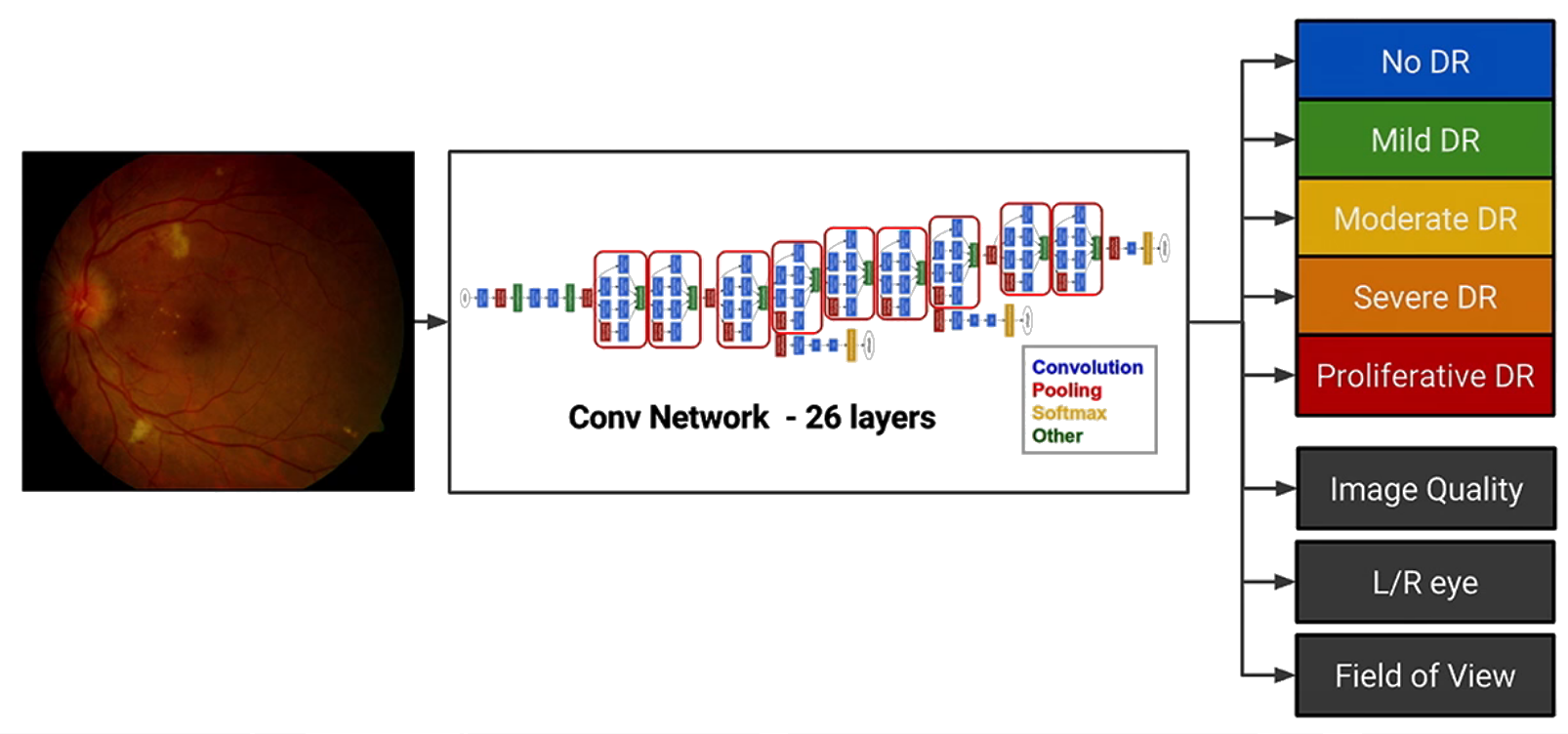
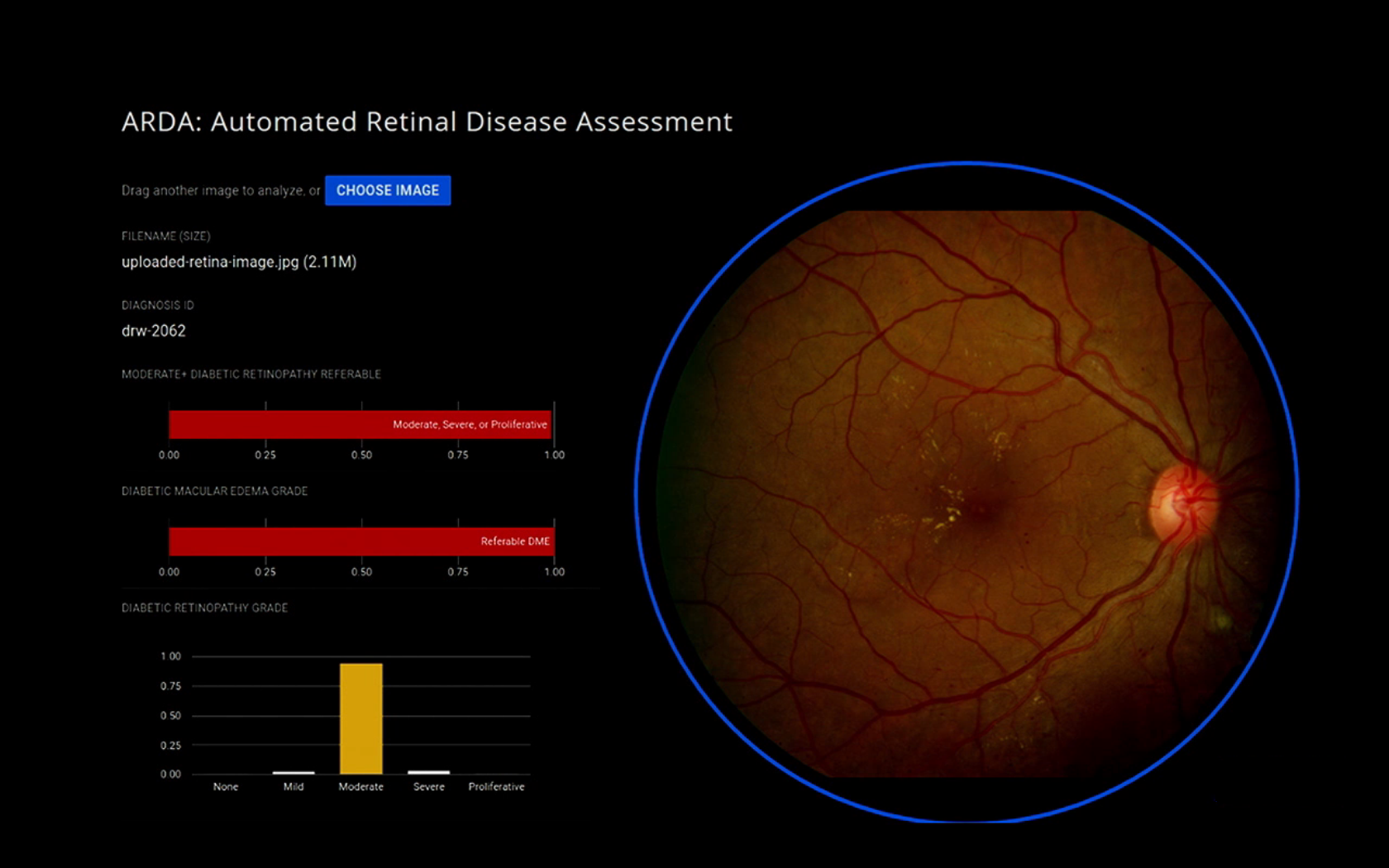
Obrazowanie siatkówki
Retinopatia cukrzycowa
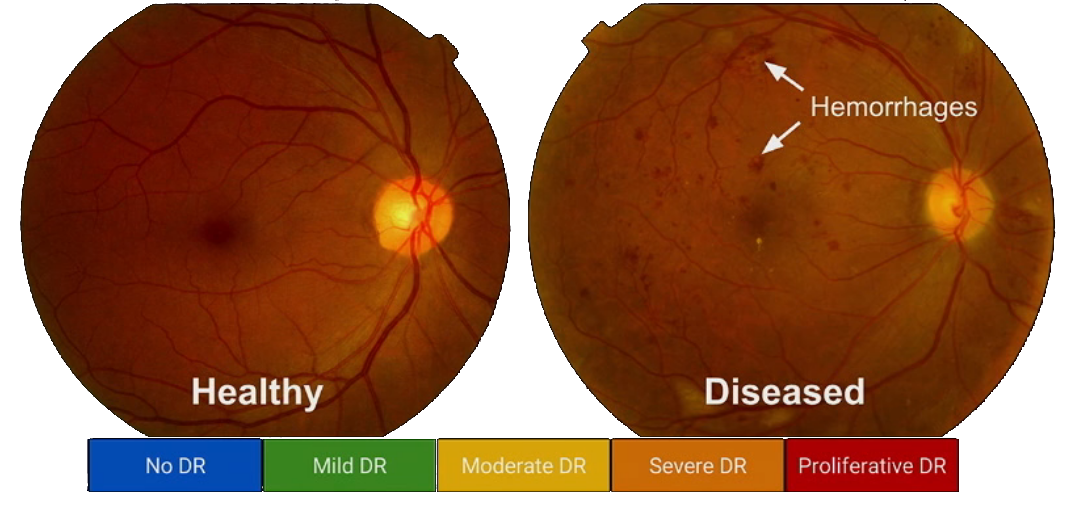
Obrazowanie siatkówki
Retinopatia cukrzycowa
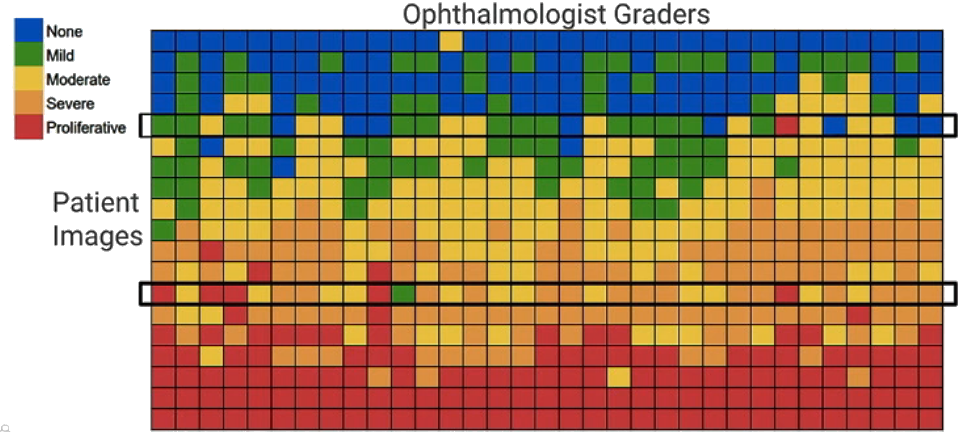
Obrazowanie siatkówki
Retinopatia cukrzycowa
Obrazowanie siatkówki
Retinopatia cukrzycowa
Rak piersi
Jednym z zastosowań uczenia maszynowego w medycynie jest klasyfikacja raka piersi
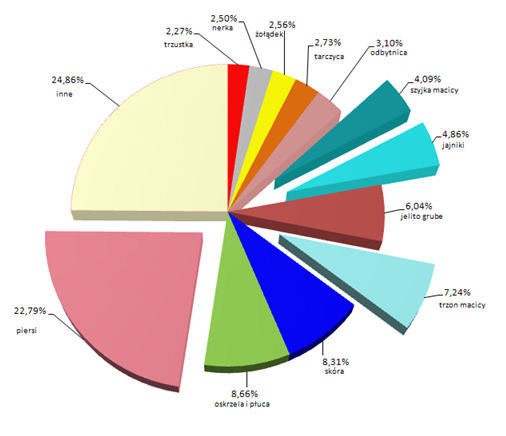
Procentowy rozkład zachorowań na nowotwory
ML w praktyce


Problem klasyfikacji złośliwości raka piersi
- złośliwy
- łagodny
Zestaw cech w pliku csv
- id
- diagnoza (M = złośliwy, B = łagodny)
- promień
- tekstura
- obwód
- obszar
- gładkość
- zawartość
- wklęsłość
- punkty wklęsłe
- symetria
- wymiar fraktalny
#header oznacza, że wiersz 0 będzie zawierać etykiety dla naszych danych
data = pd.read_csv("../input/data.csv",header=0)
#możemy teraz wypisać sobie przykładowo 2 wiersze danych
print(data.head(2))
#teraz chcielibyśmy uzyskać trochę informacji o wczytanych danych
data.info()
<class 'pandas.core.frame.DataFrame'>
RangeIndex: 569 entries, 0 to 568
Data columns (total 33 columns):
id 569 non-null int64
diagnosis 569 non-null object
radius_mean 569 non-null float64
texture_mean 569 non-null float64
perimeter_mean 569 non-null float64
area_mean 569 non-null float64
smoothness_mean 569 non-null float64
...
texture_worst 569 non-null float64
concave points_worst 569 non-null float64
symmetry_worst 569 non-null float64
fractal_dimension_worst 569 non-null float64
Unnamed: 32 0 non-null float64
dtypes: float64(31), int64(1), object(1)
memory usage: 146.8+ KB
Let's code!
#usuwamy pustą, nadmiarową kolumnę o nazwie "Unnamed: 32"
data.drop("Unnamed: 32",axis=1,inplace=True)
#sprawdźmy czy kolumna została porzucona
data.columns
Index(['id', 'diagnosis', 'radius_mean', 'texture_mean', 'perimeter_mean',
'area_mean', 'smoothness_mean', 'compactness_mean', 'concavity_mean',
'concave points_mean', 'symmetry_mean', 'fractal_dimension_mean',
'radius_se', 'texture_se', 'perimeter_se', 'area_se', 'smoothness_se',
'compactness_se', 'concavity_se', 'concave points_se', 'symmetry_se',
'fractal_dimension_se', 'radius_worst', 'texture_worst',
'perimeter_worst', 'area_worst', 'smoothness_worst',
'compactness_worst', 'concavity_worst', 'concave points_worst',
'symmetry_worst', 'fractal_dimension_worst'],
dtype='object')
#analogicznie pozbywamy się kolumny id
data.drop("id",axis=1,inplace=True)Selekcja cech
#Dzielimy cechy według odpowiednich kategorii
features_mean = list(data.columns[1:11])
features_se = list(data.columns[11:20])
features_worst = list(data.columns[21:31])
print(features_mean)
print("-----------------------------------")
print(features_se)
print("------------------------------------")
print(features_worst)
['radius_mean', 'texture_mean', 'perimeter_mean', 'area_mean', 'smoothness_mean', 'compactness_mean', 'concavity_mean', 'concave points_mean', 'symmetry_mean', 'fractal_dimension_mean']
-----------------------------------
['radius_se', 'texture_se', 'perimeter_se', 'area_se', 'smoothness_se', 'compactness_se', 'concavity_se', 'concave points_se', 'symmetry_se']
------------------------------------
['radius_worst', 'texture_worst', 'perimeter_worst', 'area_worst', 'smoothness_worst', 'compactness_worst', 'concavity_worst', 'concave points_worst', 'symmetry_worst', 'fractal_dimension_worst']
#Na koniec mapujemy typy diagnozy na łatwiejsze do przetwarzania
data['diagnosis'] = data['diagnosis'].map({'M':1,'B':0})Selekcja cech
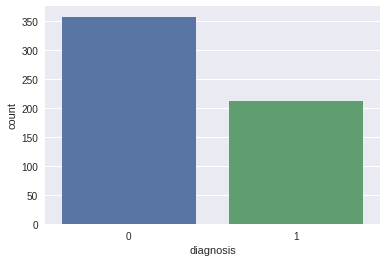
#Narysujmy sobie wykres częstości występowania danego typu diagnoz
sns.countplot(data['diagnosis'],label="Count")Analiza danych
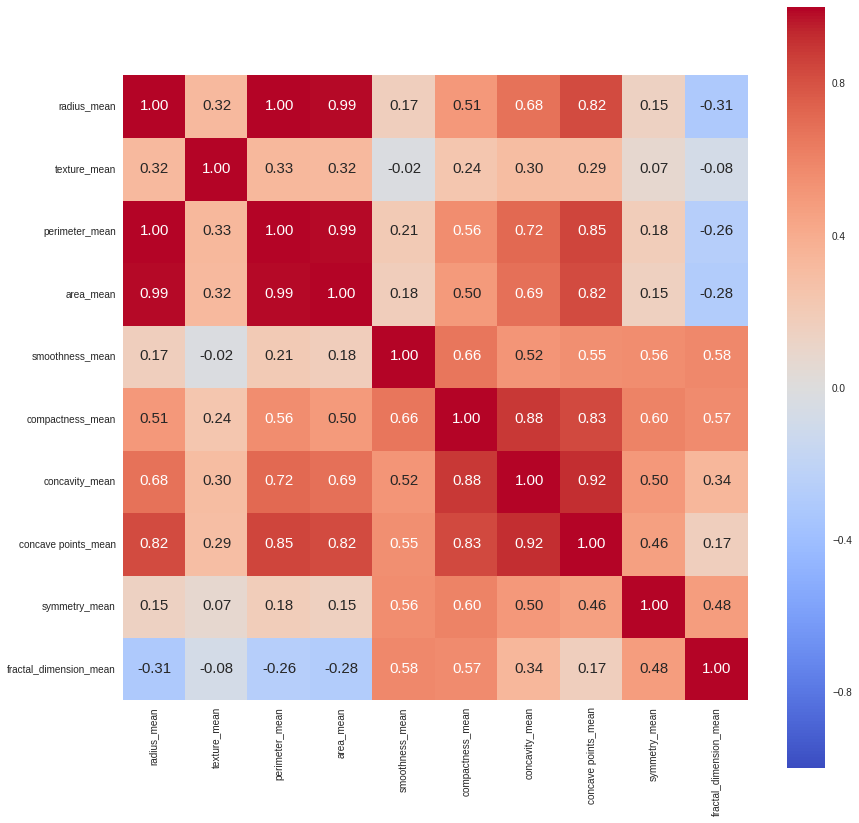
#Teraz wykorzystamy funkcje corr do znalezienia korelacji pomiędzy cechami
corr = data[features_mean].corr()
#A następnie utworzymy sobie tzw. heatmap'e,
#która ładnie nam też zależności przedstawi
plt.figure(figsize=(14,14))
sns.heatmap(corr, cbar = True,
square = True,
annot=True,
fmt= '.2f',
annot_kws={'size': 15},
xticklabels= features_mean,
yticklabels= features_mean,
cmap= 'coolwarm')Analiza danych
#od teraz będziemy używać tylko tych cech
prediction_var = ['texture_mean','perimeter_mean','smoothness_mean',
'compactness_mean','symmetry_mean']
#dzielimy zbiór danych
train, test = train_test_split(data, test_size = 0.3)
train_X = train[prediction_var] # cechy wejściowe
train_y=train.diagnosis # spodziewane wyjścia
#tak samo dla zbioru testowego
test_X= test[prediction_var]
test_y =test.diagnosis
- radius, parameter oraz area są mocno skorelowane, więc wystaczy wybrać jedną z nich
- analogicznie compactness_mean, concavity_mean and concavepoint_mean
Odrzucamy nadmiarowe cechy
#Tworzymy model klasyfikatora
model=RandomForestClassifier(n_estimators=100)
#Trenujemy model
model.fit(train_X,train_y)
#dokonujemy predykcji na zbiorze testowym
prediction = model.predict(test_X)
#Mierzymy otrzymaną poprawnosc klasyfikatora
metrics.accuracy_score(prediction,test_y)
0.92982456140350878Trenujemy i testujemy model

Nowotwór skóry
- Najpopularniejszy nowotwór w USA
- 1 na 5 Amerykanów będzie chorować
- W 2017 będzie 90k nowych zachorowań na czerniaka,
z czego 10k śmiertelnych
- Przeżywalność czerniaka to 98%
- W 2020 będzie w obiegu ponad 6 mld smartfonów
Nowotwór skóry
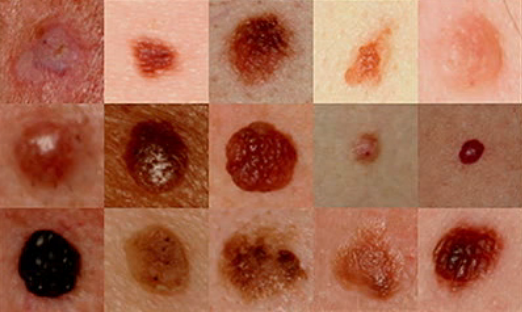
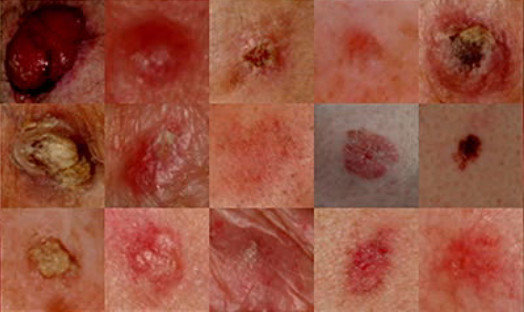
łagodny
złośliwy
Nowotwór skóry
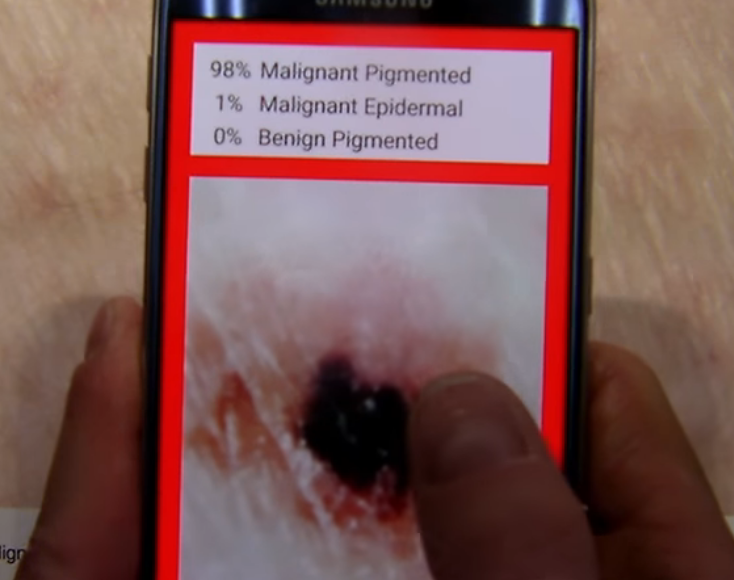
Ciekawostki
Ciekawostki
MarI/O



Ciekawostki
YOLO


Ciekawostki
Kompozycja muzyki

Zadanie domowe
Kaggle


Michał Bieroński, 218324
Mateusz Burniak, 218321