Mikel Egaña Aranguren
Candidato Profesor Adjunto
Dpto. Lenguajes y Sistemas Informáticos UPV/EHU
http://mikel-egana-aranguren.github.io
mikel.egana.aranguren@gmail.com
Méritos preferentes
Estancias en centros diferentes a UPV/EHU














Experiencia laboral y becas
Investigador contratado predoctoral
Puesto: Investigador predoctoral a tiempo completo
Institución: VIB (Vlaams Instituut voor Biotechnologie), Bélgica
Financiación: Marie Curie Early Stage Research Training (EU)
Desarrollo de Cell Cycle Ontology, incluyendo aplicación de patrones de diseño
[01/05/2006 - 01/10/2006]
Investigador contratado postdoctoral
[01/04/2014 - 31/03/2015]
Puesto: Investigador postdoctoral a tiempo parcial (80%)
Institución: Grupo de investigación Genomic Resources, departamento de Genética, Antropología Física, y Fisiología Animal, UPV/EHU
Financiación: Grupo consolidado UPV/EHU
Desarrollo de pipelines de metagenómica, Docker, SADI
Puesto: Investigador postdoctoral a tiempo completo
Institución: Grupo de investigación Biological Informatic Group, Centro de Biotecnología y Genómica de Plantas (CBGP); Grupo de Investigación Ontology Engineering Group, Departamento de Inteligenica Artificial, UPM
Financiación: Marie Curie Cofund Unite (EU)
Desarrollo de pipelines de metagenómica, Docker, SADI, ingeniería ontologías, Linked Data
[14/02/2011 - 14/02/2014]
[01/12/2010 - 01/02/2011]


Puesto: Investigador postdoctoral a tiempo completo
Institución: Facultad de Informática de la Universidad de Murcia
Financiación: Plataforma Web para Gestión de conocimiento guiada por ontologías en genómica funcional (Región de Murcia)
Ingeniería ontologías
Experiencia fuera del ámbito docente e investigador universitario
[14/01/2016 - Presente]
Puesto: Analista técnico a tiempo completo
Institución: Eurohelp Consulting S.L.
Financiación: 35% Programa Torres Quevedo MINECO (14/01/2016 – 14/01/2019)
Proyectos Linked Open Data: SOLDAGE, REPLICATE, Linked Open Data Euskadi (EJIE), LODgen, ENGIMU
Formación interna y externa
Diseño y desarrollo de arquitecturas Linked Data
[27/04/2015 - 14/01/2016]
Puesto: Analista técnico a tiempo completo
Institución: Eurohelp Consulting S.L.
Proyectos Linked Open Data: SOLDAGE, REPLICATE, Linked Open Data Euskadi (EJIE), LODgen, ENGIMU
Formación interna y externa
Diseño y desarrollo de arquitecturas Linked Data
[04/10/2016 - 25/02/2017]
Puesto: Consultor externo
Institución: Intellimedis, Luxemburgo
Desarrollo de programa de consulta de historiales médicos basado en ontologías
[01/08/2018 - 31/07/2018]
Puesto: Consultor externo
Institución: Universidad de Murcia
Redacción de Pliego Técnico para el Proyecto Hércules (4.000.000€)
Otras becas y ayudas


EPSRC/Universidad de Manchester para Doctorado en informática (UK)
Erasmus (UE, BBK), Canterbury Christ Church, UK
[2005 - 2008]
[2001]
Méritos de investigación
Estancias en centros de investigación
2005 European Bioinformatics Institute (UK)
Financiado por Network of Excellence on Semantic Interoperability and Data Mining in Biomedicine (EU)
Desarrollo Gene Ontology
Colaborador en proyectos
Proyecto: REPLICATE (Renaissance of Places with Innovative Citizenship And Technology)
Financiación: EU (Consorcio: 24.000.000€; Eurohelp: 300.000€)
Aplicación de tecnología Smart City en Donostia, Bristol, y Florencia
Publicación de datos resultantes mediante Linked Data
[2016 - 2020]
Proyecto: Servicios OpenLinkedData
Financiación: Contrato con EJIE, Gobierno Vasco, tras concurso (90.000€)
Publicación de datos de Open Data Euskadi y contenido Web en Linked Open Data
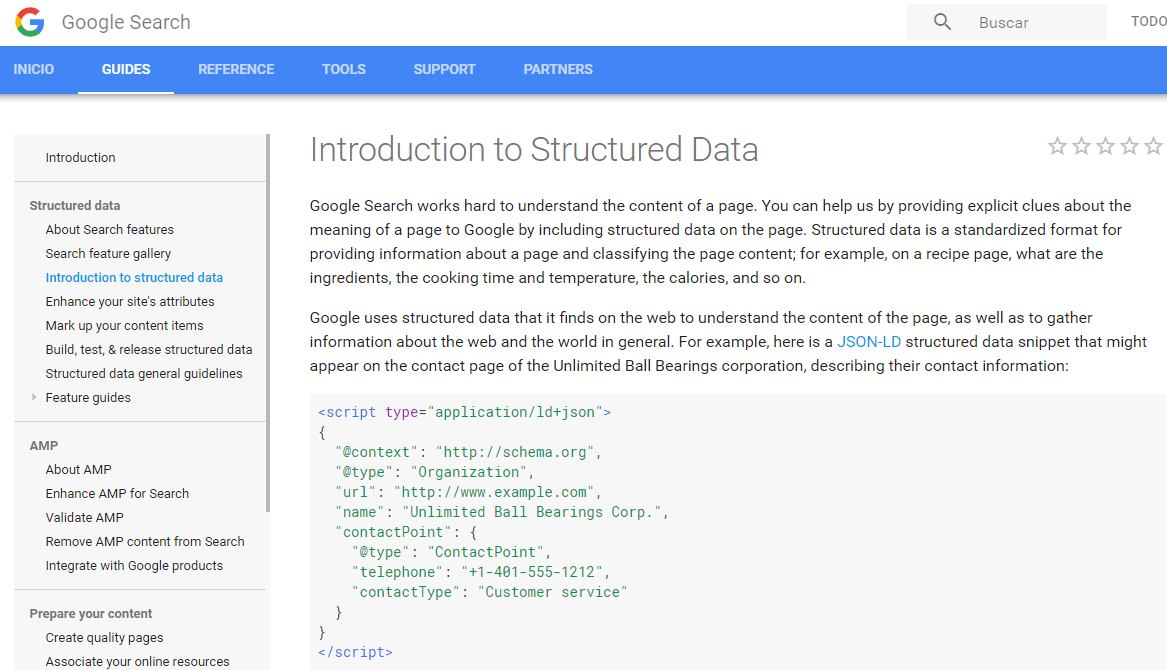
Anotación de contenido Web (euskadi.eus) mediante schema.org y JSON-LD
[2016 - 2018]
Proyecto: Servicios para la creación de un sistema Repositorio de datos de Directorio
Financiación: Contrato con EJIE, Gobierno Vasco, tras concurso
Publicación de datos de Directorio en Linked Open Data, previo procesamiento con herramienta NER
[2018 - 2019]
Proyecto: Servicios para la apertura de Datos Meteorológicos
Financiación: Contrato con EJIE, Gobierno Vasco, tras concurso
Servicios para la apertura de Datos Meteorológicos y Renovación de los Canales de Difusión de Información (Web / Móvil / etc)
[2018 - 2019]
Proyecto: SOLDAGE (Semantic Open Linked DAta Generator)
Financiación: programa HAZITEK del Gobierno Vasco (150.000€)
Asistente para generación de datos FAIR (Findable, Accesible, Interoperable, Reproducible)
[2017 - 2018]
Proyecto: Linking Open Domains, Plataforma para la generación de datos enlazados (LODGen)
Financiación: Ministerio de Industria, Energia y Turismo (40.000€), Acción Estratégica Economía y Sociedad Digital (AEESD) 1/2015
Asistente para generación de Linked Open Data y aplicaciones asociadas en el dominio del turismo
[2015 - 2018]
Proyecto: ENGIMU (Enlazando Gipuzkoa con el Mundo)
Financiación: Gipuzkoako Foru Aldundia (40.000€), Gipuzkoa IKT: Innovación digital Empresas
Asistente para generación de Linked Open Data a partir de datos provenientes de portales Open Data de administraciones públicas
[2015]

Libros
Role and application of Ontology Design Patterns in bio-ontologies. Mikel Egaña Aranguren. LAP Lambert Academic Publishing. ISBN-13: 978-3843376617, 2010
Artículos en revistas JCR
Aranguren, M., Bechhofer, S., Lord, P., Sattler, U., and Stevens, R. (2007). Understanding and using the meaning of statements in a bio-ontology: recasting the Gene Ontology in OWL. BMC bioinformatics, 8(1):57
[Factor Impacto: 2.58]

Stevens, R., Egaña Aranguren, M., Wolstencroft, K., Sattler, U., Drummond, N., Horridge, M., and Rector, A. (2007). Using OWL to model biological knowledge. International Journal of Human-Computer Studies, 65(7):583–594
[Factor Impacto: 1.293]
Egaña Aranguren, M., Wroe, C., Goble, C., and Stevens, R. (2008). In situ migration of handcrafted ontologies to reasonable forms. Data & Knowledge Engineering, 66(1):147–162
[Factor Impacto: 1.115]
Antezana, E., Egaña, M., De Baets, B., Kuiper, M., and Mironov, V. (2008b). ONTO-PERL: an API for supporting the development and analysis of bio-ontologies. Bioinformatics, 24(6):885
[Factor Impacto: 4.981]
Aranguren, M., Antezana, E., Kuiper, M., and Stevens, R. (2008a). Ontology Design Patterns for bio-ontologies: a case study on the Cell Cycle Ontology. BMC bioinformatics, 9(Suppl 5):S1
[Factor Impacto: 2.58]

Antezana, E., Blondé, W., Egaña, M., Rutherford, A., Stevens, R., De Baets, B., Mironov, V., and Kuiper, M. (2009a). BioGateway: a semantic systems biology tool for the life sciences. BMC bioinformatics, 10(Suppl 10):S11
[Factor Impacto: 2.58]
Antezana, E., Egaña, M., Blondé, W., Illarramendi, A., Bilbao, I., De Baets, B., Stevens, R., Mironov, V., and Kuiper, M. (2009b). The Cell Cycle Ontology: an application ontology for the representation and integrated analysis of the cell cycle process. Genome Biology, 10(5):R58
[Factor Impacto: 10.8]

Minarro-Gimenez, J., Egana-Aranguren, M., Villazon-Terrazas, B., and FernandezBreis, J. (2012). Publishing Orthology and Diseases Information in the Linked Open Data Cloud. Current Bioinformatics, 7(3):255–266
[Factor Impacto: 0.971]
Mironov, V., Antezana, E., Egaña, M., Blondé, W., De Baets, B., Kuiper, M., and Stevens, R. (2011). Flexibility and utility of the Cell Cycle Ontology. Applied Ontology, 6(3):247–261
[Factor Impacto: 0.615]
Miñarro-Gimenez, J., Aranguren, M., Béjar, R., Fernández-Breis, J., and Madrid, M. (2011). Semantic integration of information about orthologs and diseases: The OGO system. Journal of biomedical informatics, 44:1020–1031
[Factor Impacto: 2.126]
Egaña Aranguren, M., Fernández-Breis, J. T., Antezana, E., Mungall, C., Rodríguez González, A., and Wilkinson, M. D. (2013). OPPL-Galaxy, a Galaxy tool for enhancing ontology exploitation as part of bioinformatics workflows. Journal of biomedical semantics, 4(1):2
[Factor Impacto: 2.24]
Duque-Ramos, A., Fernández-Breis, J. T., Iniesta, M., Dumontier, M., Egaña Aranguren, M., Schulz, S., Aussenac-Gilles, N., and Stevens, R. (2013). Evaluation of the OQuaRE framework for ontology quality. Expert Systems with Applications, 40(7):2696–2703.
[Factor Impacto: 2.26]
Aranguren, M. E., González, A. R., and Wilkinson, M. D. (2014). Executing SADI services in Galaxy. Journal of Biomedical Semantics, 5(1):42+.
[Factor Impacto: 2.26]
José Antonio Miñarro Giménez, Mikel Egaña Aranguren, Boris Villazón Terrazas, and Jesualdo Tomás Fernández Breis (2014). Translational research combining orthologous genes and human diseases with the OGOLOD dataset. Semantic Web Journal, 5(2):145–149
[Factor Impacto: 1.786]
González, A. R., Callahan, A., Toledo, J. C., García, A., Aranguren, M. E., Dumontier, M., and Wilkinson, M. D. (2014a). Automatically exposing OpenLifeData via SADI semantic Web Services. Journal of Biomedical Semantics, 5(1):46+
[Factor Impacto: 2.26]
Pawluczyk, M., Weiss, J., Links, M. G., Aranguren, M. E., Wilkinson, M. D., and Egea-Cortines, M. (2015). Quantitative evaluation of bias in PCR amplification and Next Generation Sequencing derived from metabarcoding samples. Analytical and Bioanalytical Chemistry, 407(7):1841–1848
[Factor Impacto: 3.436]

Mikel Egaña Aranguren, Mark D. Wilkinson (2015). Enhanced reproducibility of SADI Web service workflows with Galaxy and Docker. GigaScience, 4(59)
[Factor Impacto: 7.46]







3.- Semantics: what the data means
2.- Functional: what I did with the data
1.- Computational: how I did it
Capítulos en libros
Aranguren, M., Stevens, R., Antezana, E., Fernández-Breis, J.T., Kuiper, M., and Mironov, V. (2010). Technologies and Best Practices for Building Bio-Ontologies. In Knowledge-Based Bioinformatics, volume Gil Alterovitz and Marco Ramoni (Eds.), pages 67–86. Wiley Online Library
Ponencias en congresos
González, A. R., Romero, M. M., Aranguren, M. E., and Wilkinson, M. D. (2014). Nanopublishing clinical diagnoses: tracking diagnostic knowledge base content and utilization. In 27th International Symposium on Computer-Based Medical Systems (CBMS), pages 335–340
Iglesias, A. R., Aranguren, M. E., González, A. R., and Wilkinson, M. D. (2013). Plant Pathogen Interactions Ontology (PPIO). In Rojas, I. and Guzman, F. M. O., editors, IWBBIO, pages 695–702. Copicentro Editorial
Aranguren, M., Fernández-Breis, J., and Antezana, E. (2011). OPPL-Galaxy: enhancing ontology exploitation in galaxy with OPPL. In Proceedings of the 4th International Workshop on Semantic Web Applications and Tools for the Life Sciences, pages 12–19. ACM
Miñarro-Giménez, J., Aranguren, M., García-Sánchez, F., and Fernández-Breis, J. (2010). A semantic query interface for the OGO platform. In Information Technology in Bio-and Medical Informatics, ITBAM 2010, pages 128–142. Springer
Egaña, M., Rector, A., Stevens, R., and Antezana, E. (2008). Applying ontology design patterns in bio-ontologies. In Gangemi, A. and Euzenat, J., editors, Knowledge Engineering: Practice and Patterns, volume 5268 of Lecture Notes in Computer Science, pages 7–16. Springer
Dirección de trabajos de fin de máster
[2014-2015]
Salvador Alonso Martínez, "Imagen Docker para pipelines de Metagenómica", Máster en Bioinformática de la Universidad de Murcia. 12 Créditos ECTS. Calificación: Notable
Charlas invitadas
[2016]
Los Datos Enlazados y la Web Semántica. Tikitalka, VE Interactive, Spain
[2014]
[2011]
Building reasonable biomedical ontologies for a Life Sciences Semantic Web. 3S (Systems, Synthetic, and Semantic) Biology summer school. CIBIO (Centre for Integrative Biology), University of Trento, Italy
Linked Data for Functional Genomics. NTNU, Trondheim, Norway
Aplicación de la Web Semántica en Biología Molecular. Universidad de Deusto, Facultad de Ingenieria, Spain
Aplicación de la Web Semántica en Bioinformática. UM, Facultad de Informática, Spain
Métodos y resultados actuales en Bioinformática: know-how y know-what de las redes tecnocientíficas en Bioinformática. EHU, Facultad de Filosofia, Spain
[2010]
[2008]
[2004]
Tribunales doctorado
Alejandro Rodríguez Iglesias, "FAIR approaches applied to unraveling plantpathogen interactions data and RNA processing evolution", UPM, Spain
Meifania Monica Chen, "Lipoprotein Ontology: A Formal Representation of Lipoproteins", Curtin University, Australia
Jose Antonio Miñarro-Gimenez, "Entorno para la gestion semantica de informacion biomedica en investigacion traslacional". UM, Spain
Doris Meja Avila, "Estrategia de interoperabilidad semantica en el contexto de integracion de conocimiento geografico y ambiental. Caso de aplicacion: Biodiversity Ontology". UPM, Spain
[2014]
[2013]
[2012]
[2011]
Revisor
2017 Chapter review "Integrating Biological Data using Semantic Web Technology" in Evolutionary Genomics. computational and statistical methods, 3rd edition, Springer
2015 BMC Medical Informatics and Decision Making
2013 PeerJ
2013 Data and Knowledge Engineering (DKE)
2012 BMC Bioinformatics
2012 Journal of Biomedical Informatics (JBI)
2012 Computational and Mathematical Methods in Medicine (CMMM)
2012 Journal of Medical Systems (JOMS)
2012 Journal of Biomedical Semantics (JBS)
2011 Semantic Web Journal (SWJ)
2011 Journal of Research and Practice in Information Technology (JRPIT)
Comité científico
2017 Semantic Web Solutions for Large-Scale Biomedical Data Analytics (SeWeBMeDA)
2015 CAEPIA
2012 Managing Interoperability and compleXity in Health Systems, in conjunction with the ACM International Conference on Information and Knowledge Management
2012 Joint Workshop on Semantic Technologies Applied to Biomedical Informatics and
Individualized Medicine (SATBI + SWIM 2012), in conjunction with International Semantic Web Conference (ISWC)
2011 Managing Interoperability and compleXity in Health Systems, in conjunction with the ACM International Conference on Information and Knowledge Management
2011 Knowledge Capture (K-CAP)
2011 Semantic Applied Technologies on Biomedical Informatics (SATBI 2011), in
conjunction with the ACM International Conference on Bioinformatics and Computational Biology
2008 ONTORACT
Métricas de producción científica
H-index (Google Scholar): 14
H-index (Scopus): 9
Citas totales (Google Scholar): 739
[Más información en https://mikel-egana-aranguren.github.io]
Meritos docentes y de gestión
Divulgación
Software libre (GNU/linux) para biólogos. BioGaia 3, 2003 (Colegio Oficial de Biólogos de Euskadi)
¿Qué puede hacer la web semántica por la biología? BioGaia 7, 2009 (Colegio Oficial de Biólogos de Euskadi)
Docencia en másteres universitarios oficiales
10 horas (1 crédito ECTS) asignadas para impartir clases en español sobre Life Sciences Semantic Web en asignatura Tecnologías Informáticas Avanzadas del Máster en Bioinformática de la Universidad de Murcia
[2017-2018]
10 horas (1 crédito ECTS) impartidas en español sobre Life Sciences Semantic Web en asignatura Tecnologías Informáticas Avanzadas del Máster en Bioinformática de la Universidad de Murcia
[2016-2017]
10 horas (1 crédito ECTS) impartidas en español sobre Life Sciences Semantic Web en asignatura Tecnologías Informáticas Avanzadas del Máster en Bioinformática de la Universidad de Murcia
[2015-2016]
10 horas (1 crédito ECTS) impartidas en español sobre Life Sciences Semantic Web en asignatura Tecnologías Informáticas Avanzadas del Máster en Bioinformática de la Universidad de Murcia
[2014-2015]
0,3 créditos ECTS impartidos en inglés sobre sistema Bioinformático Galaxy en el Máster Erasmus Mundus en Medio Ambiente y Recursos Marinos, UPV/EHU
[2013-2014]
Docencia universitaria impartida en otro idioma
8 horas impartidas en inglés en curso ATHENS “Semantic technologies: Ontology Development and Linked Data”, UPM
[17/11/2012 -24/11/2012]
8 horas impartidas en inglés en curso ATHENS “Semantic technologies: Ontology Development and Linked Data”, UPM
8 horas impartidas en inglés en curso ATHENS “Semantic technologies: Ontology Development and Linked Data”, UPM
[17/04/2011 - 24/04/2011]
[14/11/2011 - 18/11/2011]
Otros méritos docentes
[2016 - 2017] 924 horas dedicadas a tutorización de prácticas externas extracurriculares del Grado en Ingeniería Informática de Gestión y Sistemas de Información (UPV/EHU), en Eurohelp Consulting S.L.
[2016] 8 horas impartidas sobre Linked Open Data en Español en EJIE (Eusko Jaurlaritzaren Informatika Elkartea, Gobierno Vasco)
[2015] 8 horas impartidas sobre Linked Open Data en Español en IZFE (Informatika Zerbitzuen Foru Elkartea, Diputación Foral de Gipuzkoa)
[08/09/2014 - 11/09/2014] Tutorial "Semantic biology: Use of Semantic Web resources for knowledge discovery". 3S (Systems, Synthetic, and Semantic) Biology summer school. CIBIO (Centre for Integrative Biology), Universidad de Trento, Italia. Inglés, 8 horas
[2013 - 2014] 8 horas impartidas en Español sobre Life Sciences Semantic Web en Máster propio en Bioinformática de la Universidad de Murcia
[2013] Charlas introductorias para estudiantes de bachiller, con motivo de la semana de las ciencia del CBGP, UPM-INIA, Madrid. Español, 2 horas
[2013] Tutoriales internos sobre sistema Galaxy para análisis bioinformáticos a investigadores del CBGP, UPM-INIA, Madrid. Español e inglés
[2012 - 2013] 8 horas impartidas en Español sobre Life Sciences Semantic Web en Máster propio en Bioinformática de la Universidad de Murcia
[2010 - 2011] 12 horas impartidas en la asignatura Inteligencia Artificial, Título Oficial de Ingeniería Informática, UPM
[2011] Tutorial sobre herramienta Populous en SWAT4LS, Londres, inglés
[2005 - 2008] Tutoriales sobre OWL para biólogos, inglés
Meritos académicos
Estudios de postgrado
[01/01/2005 - 24/07/2009] Doctorado en informática, Universidad de Manchester, Reino Unido (Título homologado). Nota final: minor corrections
[22/09/2003 - 19/09/2004] Máster bioinformática, Universidad de Manchester, Reino Unido (Título homologado). Nota final: distinction
Estudios de grado
[22/09/1997 - 25/09/2003] Licenciatura biología, UPV/EHU. Nota media: 6,6
[23/09/2001 - 23/02/2002] Estancia Erasmus en Canterbury Christ Church University College, Reino Unido
Idiomas
Inglés
Nivel alto, hablado y escrito. Uso continuo durante estancia Erasmus, máster bioinformática, doctorado en informática, postgrado Marie Curie, congresos, docencia, etc
[10/09/2009] Certificado IELTS 7,5 (Equivalente a C2)
Euskera
Nivel alto, hablado y escrito
[18/06/2010] EGA
[19/07/2010] HABE 3 (C1)
Otros méritos académicos
[23/06/2017] Curso sobre modelización predictiva, 20 horas. Tecnalia
[16/04/2003] Curso sobre Microsoft Visual Basic, 25 horas. UPV/EHU
[01/04/2003] Curso sobre prevención de riesgos laborales, 30 horas. UPV/EHU
[11/03/2003] Curso sobre Administración UNIX, 24 horas. UPV/EHU
[16/12/2002] Curso sobre norma ISO 9001:2000, calidad y auditorías internas, 30 horas. UPV/EHU
[19/07/2002] Curso sobre XML, 16 horas. Universidad de Deusto
[21/03/1998] Curso técnico medioambiente Ekoeskola, 65 horas. Plataforma ecologista Erreka
Programa docente
Ingeniería del Software
Introducción
Producir software de excelencia técnica
Aplicar buenas prácticas actuales con rigor
Individuo/grupos
Contexto realista
Prerequisitos
Conocimientos básicos de:
- Programación orientada a objetos con Java
Asignaturas aprobadas:
- Metodología de la Programación
- Programación Básica
- Programación Modular y Orientación a Objetos
- Estructuras de Datos y Algoritmos
| Tipo docencia | Magistral | Prácticas ordenador |
|---|---|---|
| Horas lectivas | 30 | 30 |
| Horas de trabajo | 45 | 45 |
Docencia
Programa teórico
TEMA 1. Introducción
- Información general sobre la asignatura: temario, métodos de evaluación y de docencia
- Nociones generales sobre la Ingeniería del Software: historia, contexto actual, objetivos, métodos, y herramientas actuales
TEMA 2. UML
- Presentación de UML
- UML estructural
- UML de comportamiento
TEMA 3. Scrum
- Introducción teórica a metodologías ágiles y Scrum
- Roles: product owner, scrum master
- Funcionamiento mediante sprints
TEMA 4. Control de versiones distribuido
- Introducción al control de versiones distribuido: objetivos y justificación, características, comparación con control de versiones centralizado
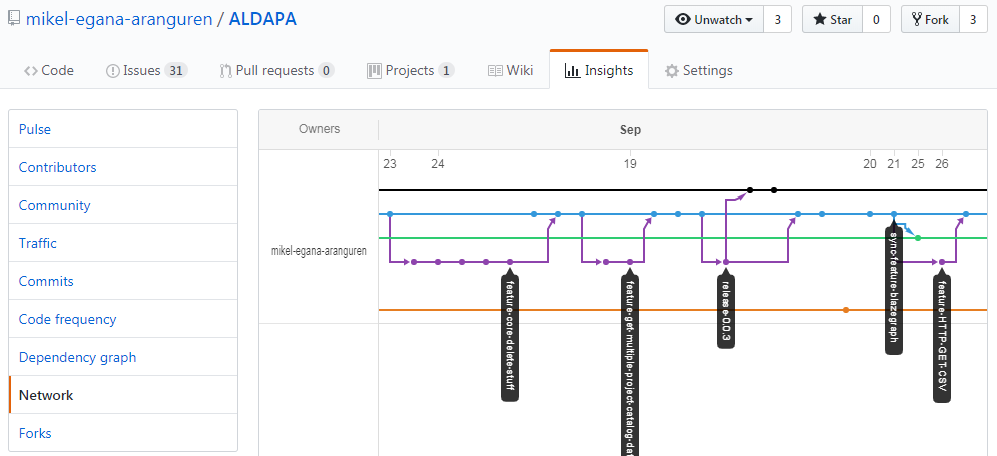
- Control de versiones distribuido con GitHub y método GitFlow
- Gestión de proyectos Scrum en GitHub: Issues, Milestones, Projects (Kanban)
TEMA 5. Buenas prácticas de diseño y arquitectura de software
- Patrones GRASP: controller, creator, indirection, information expert, high cohesion, low coupling, polymorphism, protected variations, pure fabrication
- Introspección y metaprogramación
- Patrones de diseño: patrones de creación, patrones estructurales, patrones de comportamiento, patrones de concurrencia
- Antipatrones y "bad smells"
TEMA 6. Refactorización
- Definición e introducción téorica
- Ejemplos y técnicas más usadas
TEMA 7. Tests
- Definición e introducción téorica
- Tests unitarios
- Tests de integración y de sistema
- Regresión
Programa práctico
PRÁCTICA 1. GitHub
El alumno aprenderá a:
- Participar en un proyecto de desarrollo de software en equipo mediante la plataforma GitHub
- Crear un repositorio en GitHub y configurar el plugin EGit de Eclipse para conectarse
- Distinguir entre el repositorio local y remoto, y cómo realizar commit, push y pull
- Crear ramas y fusionarlas mediante merge
- Crear forks, hacer pull requests y configurar remotos upstream
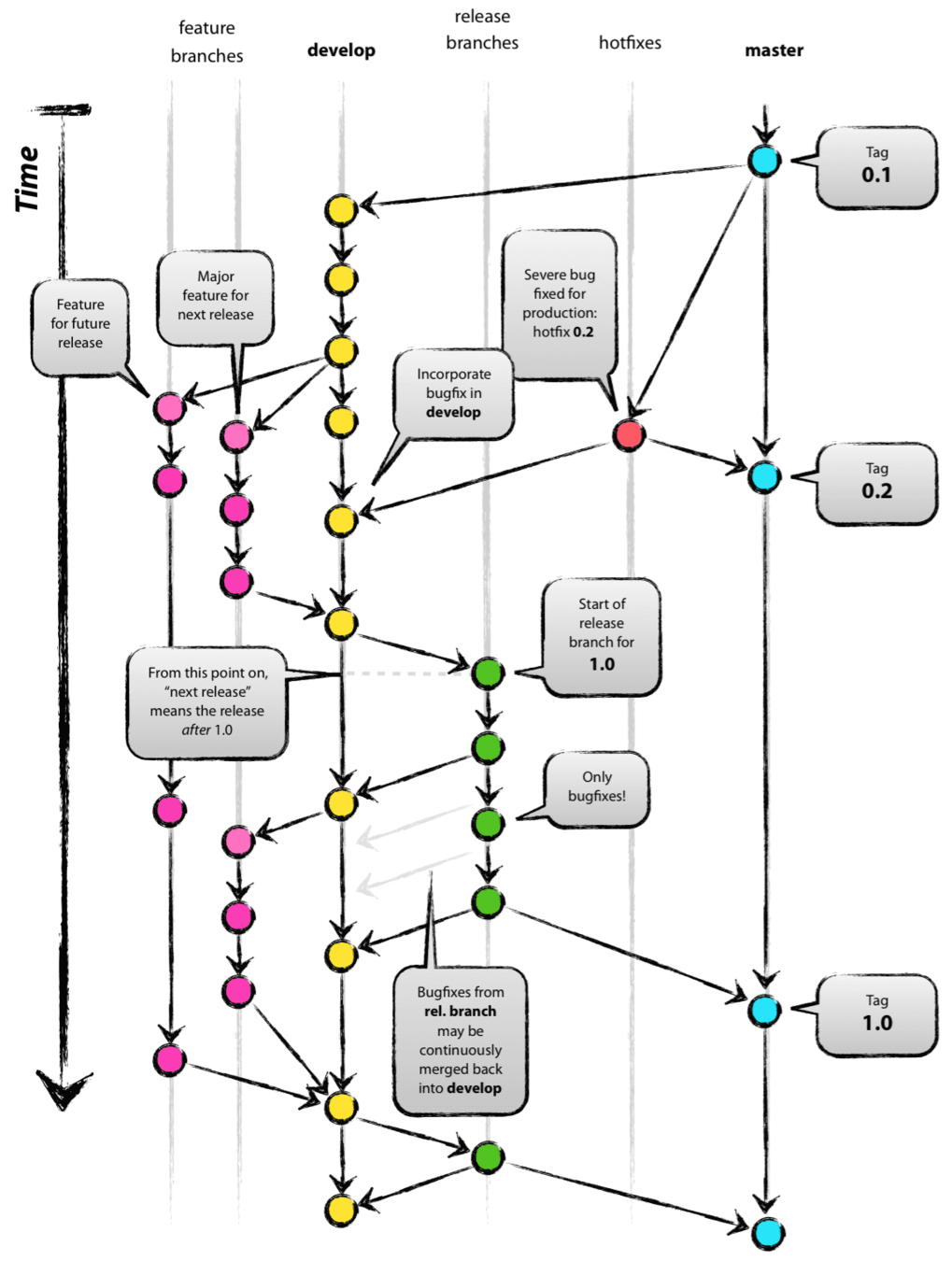
- Trabajar mediante el modelo GitFlow: rama master, rama develop, ramas feature-*, ramas release-*, ramas hotfix-*, y relaciones entre ramas
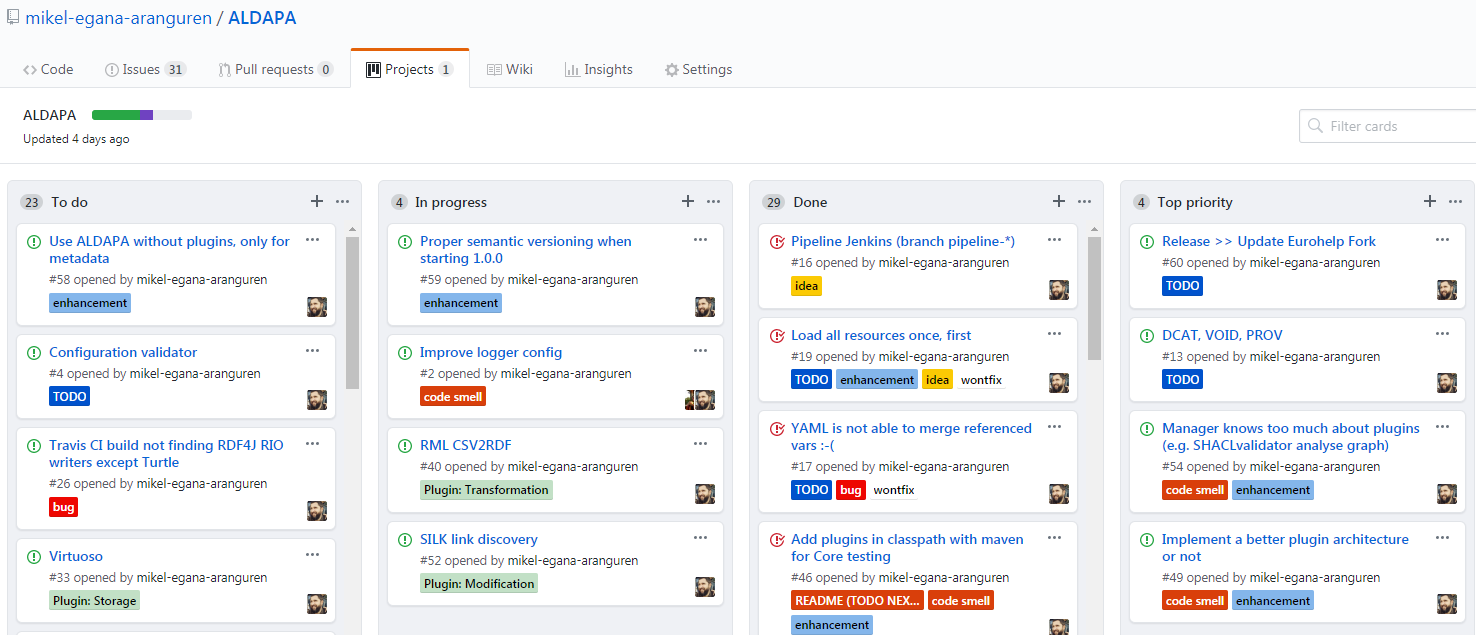
- Trabajar con GitHub Projects: Tags, Releases, Issues, Milestones, Kanban
Los alumnos trabajarán en grupos sobre un proyecto común, con un código fuente muy simple
PRÁCTICA 2. Maven y Eclipse
El alumno aprenderá a:
- Entender el funcionamiento básico de Maven: ciclo de vida, objetivos, repositorios
- Crear y configurar un proyecto con Maven
- Gestionar dependencias con Maven
- Construir un proyecto con Maven
- Integración continua con Travis y GitHub
Los alumnos trabajarán individualmente en un proyecto
PRÁCTICA 3. Tests y logging en Eclipse
El alumno aprenderá a:
- Crear y ejecutar tests unitarios en Eclipse/Maven con JUnit
- Configurar un logger adecuadamente, con LOG4J2, en Eclipse/Maven
- Análisis de cobertura de tests con CodeCov y GitHub
Los alumnos trabajarán individualmente en un proyecto
PRÁCTICA 4. Buenas prácticas de desarrollo de software
El alumno aprenderá a:
- Aplicar los patrones de diseño aprendidos en la parte teórica
- Aplicar el análisis estático de código con Codacy y GitHub, para detectar antipatrones y "code smells"
Los alumnos trabajarán individualmente en un proyecto
Metodología
Clases teóricas:
- Breve exposición
- Discusión, ejercicios, etc.
Clases prácticas:
- Aplicación de teoría (individual/grupos)
Proyectos:
- Grupos de 4
- Lista inicial de proyectos disponibles, con requisitos y descripción
- Scrum, GitHub, Gitflow
Evaluación
60% Exámen
40% Proyecto en grupos
Uso correcto de GitHub y modelo GitFlow (ramas, merges, pull requests)
Uso de Tags, Releases, Issues, Milestones, y Kanban para la gestión del proyecto mediante Scrum en GitHub
Aplicación de buenas prácticas de diseño y arquitectura de software
Aplicación correcta de técnicas de desarrollo de software: integración continua, tests unitarios, Logger, Maven, etc.
Calidad del código fuente
Calidad de la documentación
Aportaciones de cada alumno mediante analisis estadístico del código fuente con GitInspector
Defensa del proyecto
Proyecto de investigación
Estrategias para publicar y consumir Grandes Datos sobre Metagenómica de acuerdo a los principios FAIR
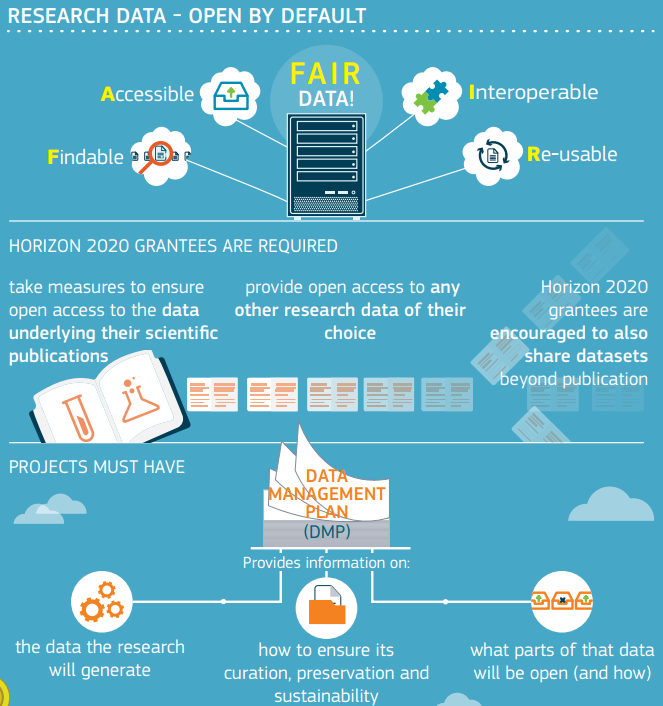
FAIR DATA
FINDABLE
→ Unambiguous identifiers supported by searchable metadata
ACCESSIBLE
→ Clearly-defined access protocol, preferably machine-actionable
INTEROPERABLE
→ Use shared vocabularies/ontologies in machine-accessible format
REUSABLE
→ Contextual information, allowing proper interpretation
→ Rich provenance information facilitating accurate citation
“Make adequate data stewardship mandatory
for all research proposals.
...Horizon 2020, should only support projects that properly address Data Stewardship... data infrastructures, that do not specify FAIR conditions...
should not be eligible for funding.”
Background

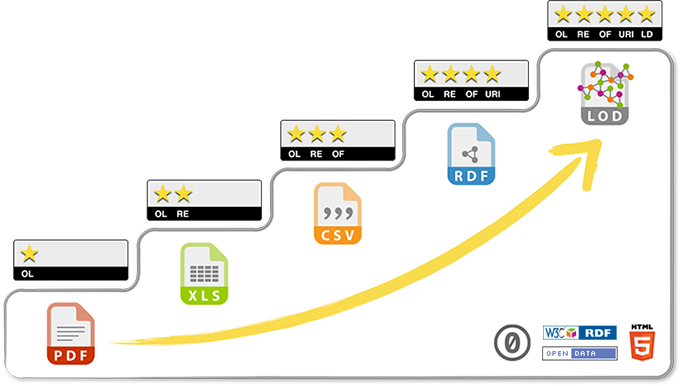
Linked Data
1.- Use URIs to identify entities and their relationships.
2.- Use HTTP URIs in order for agents to resolve (locate on the Web) such entities.
3.- When an entity is resolved via HTTP, information about the entity should be provided, in open standards. The format in which information is provided should be defined through content negotiation with the requesting agent (http://tools.ietf.org/html/rfc7231#section-5.3).
4.- The provided information should have HTTP resolvable typed links to other Linked Data resources, in order for the agent to discover more information by simply browsing through the links, as in "normal" Web browsing.
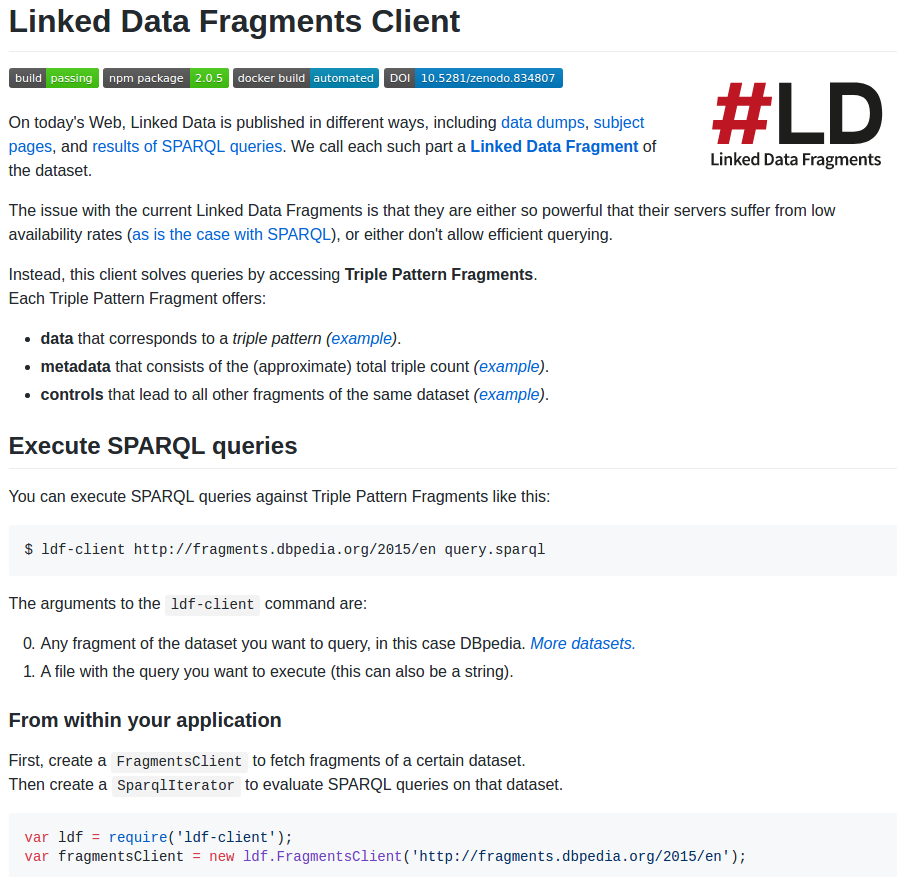
SADI

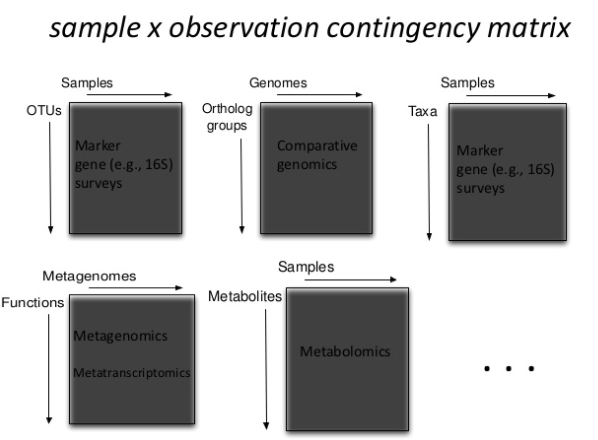
Big Data as Linked Data
1.- "Pure Linked Data" strategy
2.- "SADI Linked Data" strategy
3.- "Linked Data Fragments" strategy
1.- "Pure Linked Data" strategy

2.- "SADI Linked Data" strategy
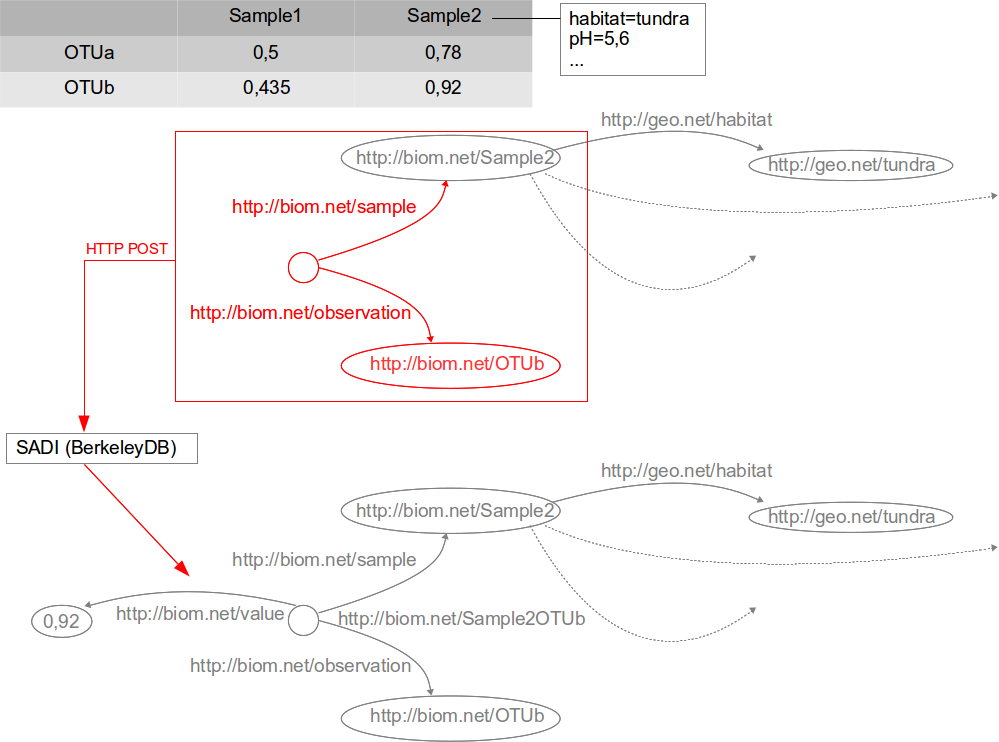
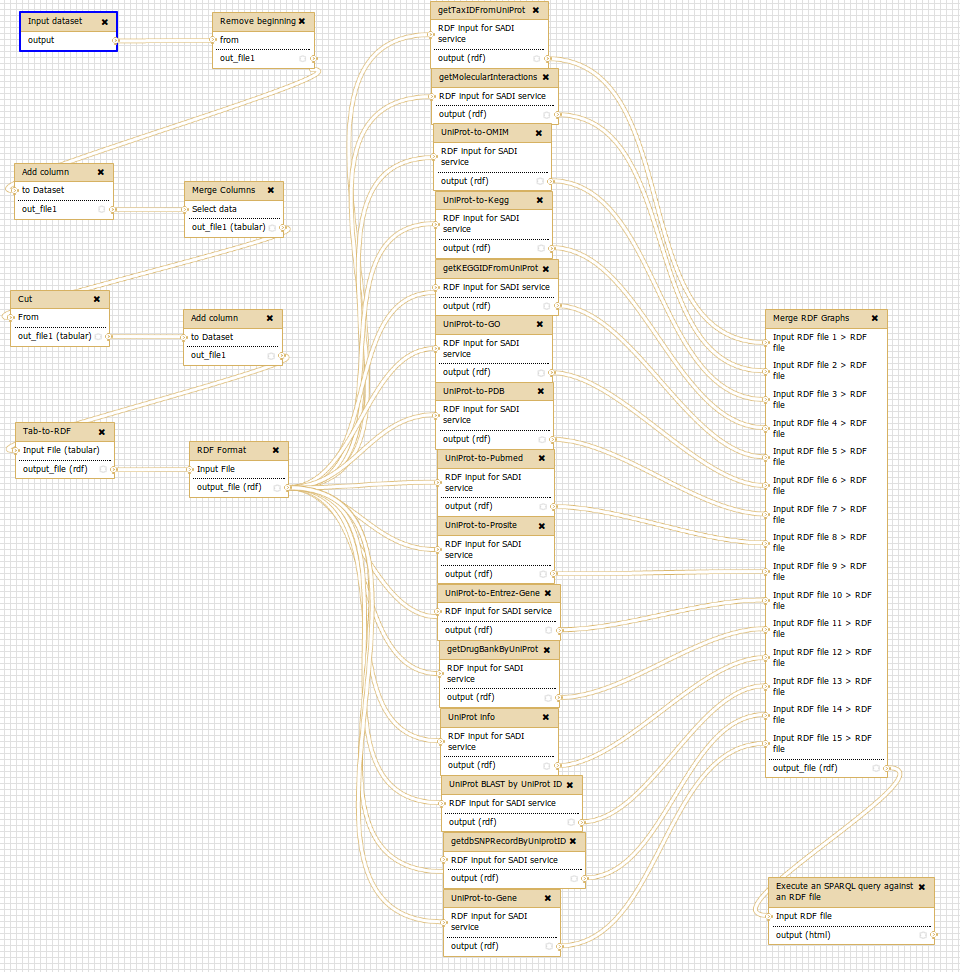

3.- "Linked Data Fragments" strategy
Big Data as Linked Data: specific targets
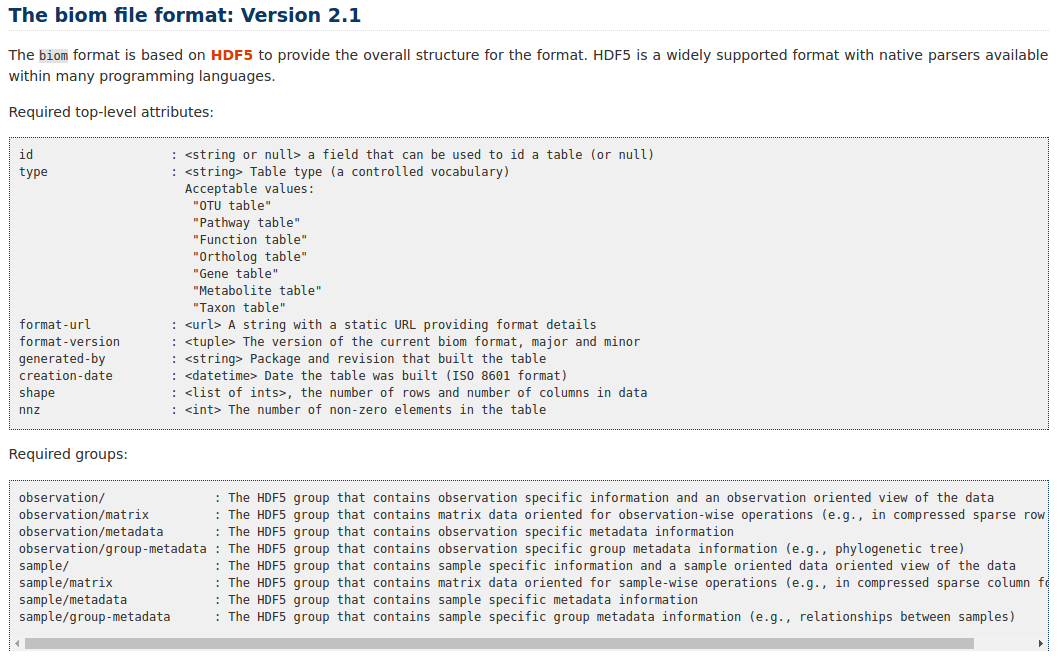
Develop HDF5 (BIOM) to RDF converter
Values (Key-Value Store / RDF )
Metadata (RDF)
Metadata links to LOD cloud (RDF)
Provenance
Convert BIOM to RDF
Publish RDF
Pure Linked Data
SADI Linked Data
Linked Data Fragments
Key-Value store
Triple Store
Link discovery
Evaluation
New queries due to integration
Performance
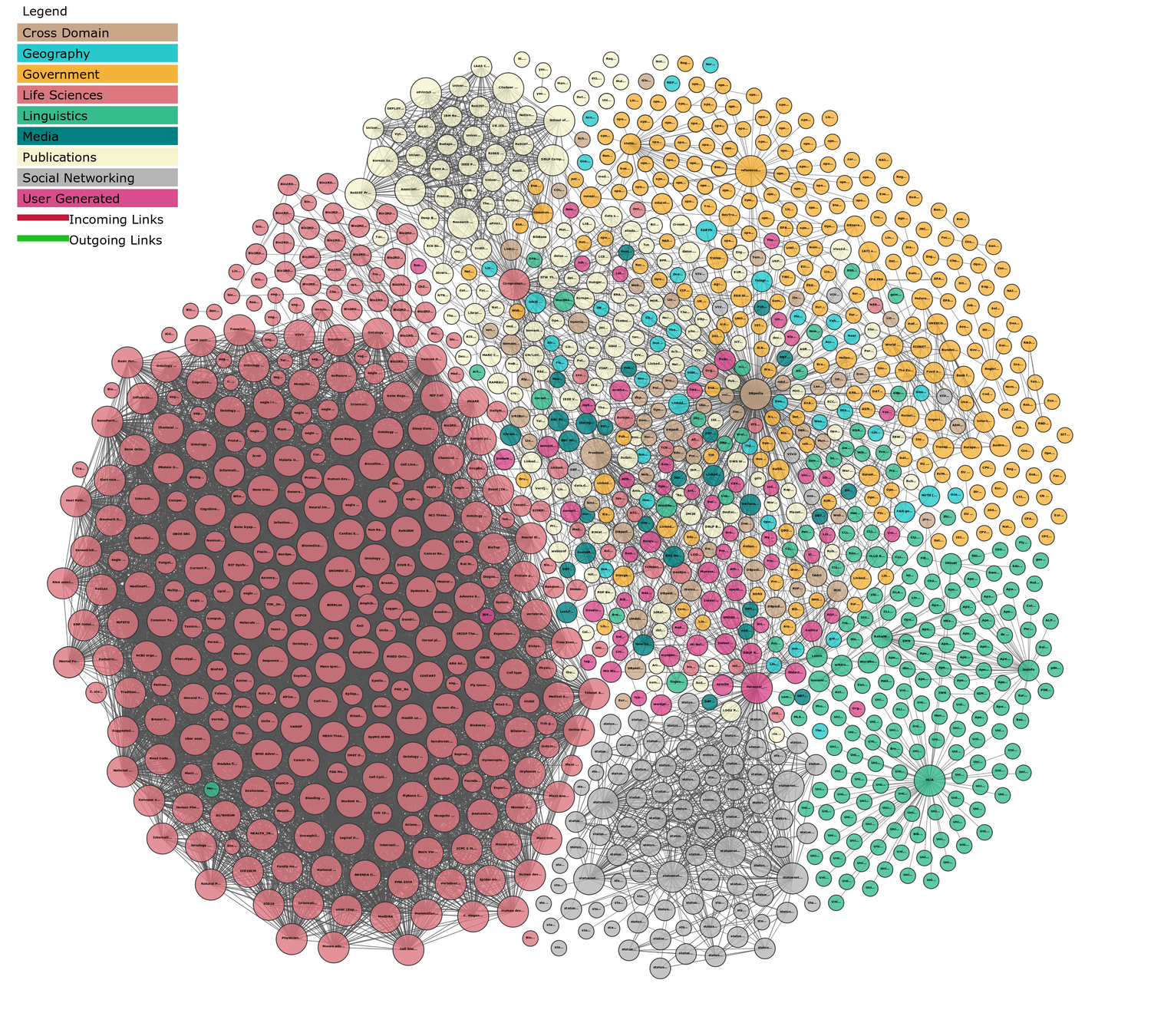
???
2020 Mikel Egaña Aranguren EHU Ingenieria Software Ayudante Doctor
By mikel-egana-aranguren
2020 Mikel Egaña Aranguren EHU Ingenieria Software Ayudante Doctor
- 1,335