EXPLORATORIOS EN R
Javier Rodríguez Barrios
MSc. Multivariados
Taller 2.3 Explor. Plancton
Figuras de Burbujas
# Librerías requeridas
library(tidyverse)
library(dplyr)
library(kableExtra) # Para la edición de tablas
library(readxl) # Cargar bases de Excel
library(ggrepel) # insertar rótulos a los puntos
library(plotrix) # Figuras de cajas con múltiples variables
library(ggplot2) # Paquete gráfico
library(forcats) # Para manipulación de factores
library(viridis) # Opciones de paletas de colores
library(plotly)
library(hrbrthemes)
library(gapminder)1. BURBUJAS CON DATOS HIPOTÉTICOS
# Cargar datos desde Excel
biol <- read_xlsx("plancton.xlsx", sheet = "Riqueza")
# Explorar estructura
# glimpse(biol)
# summary(biol)
# tabla con los datos
head(biol) %>%
kbl() %>%
kable_classic(full_width = F)# Datos de ejemplo
x <- c(2, 5, 7, 3, 6, 1, 9, 2)
y <- c(2.2, 2, 1, 2, 1, 4, 1, 6)
size <- c(100, 30, 50, 250, 120, 140, 80, 36)
group <- c("A", "A", "A", "B", "C", "B", "D", "B")
group1 <- c("A1", "A2", "A3", "B1", "C1", "B2", "D1", "B2")
# Data frame
datos <- data.frame(x, y, size, group, group1)Base de datos en formato ancho
# Figura de burbujas
ggplot(datos, aes(x = x, y = y, size = size, color = group)) +
geom_point() +
scale_size(name = "Tamaño", range = c(1, 10)) +
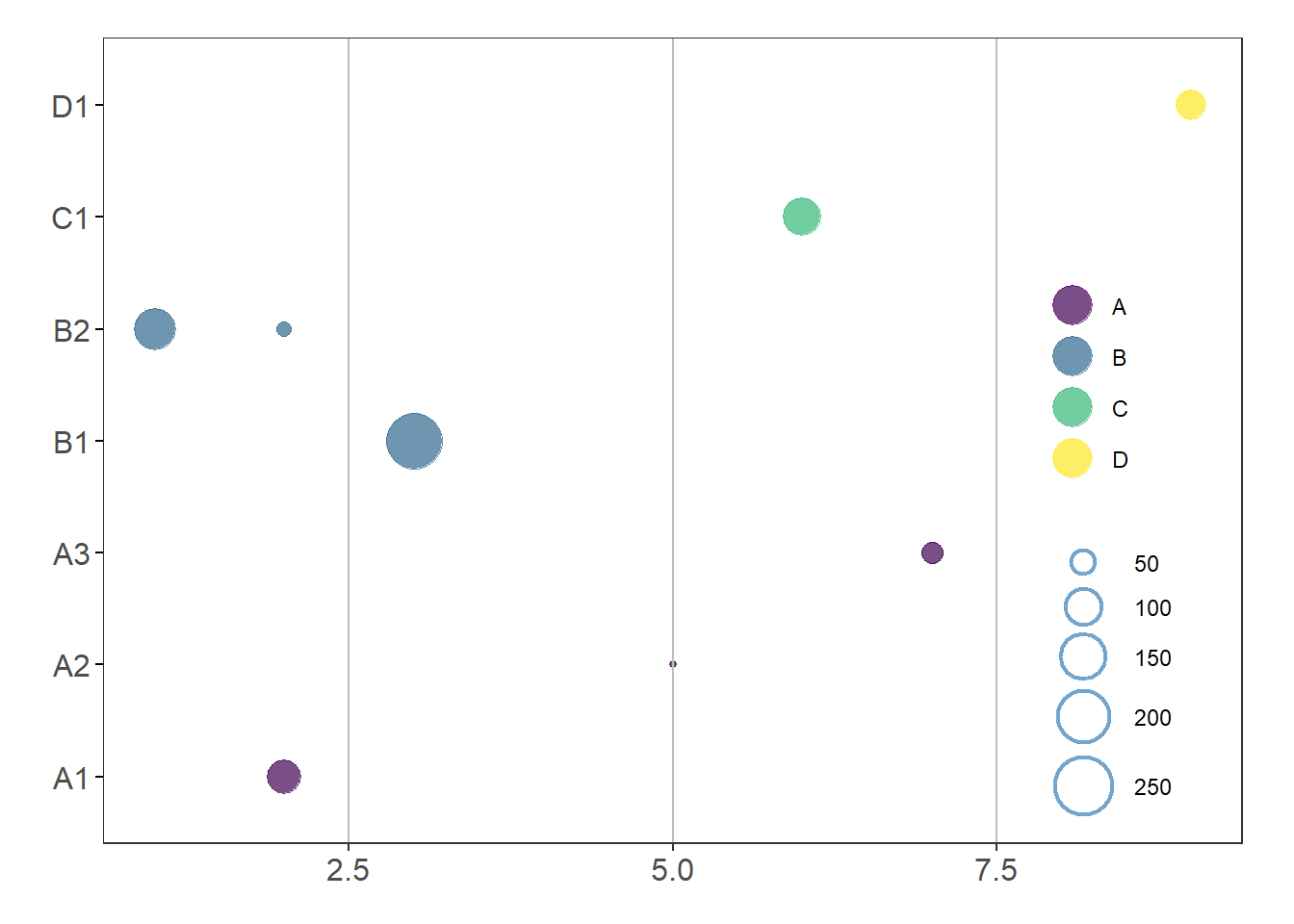
theme_bw()# Gráfico mejorado
ggplot(datos, aes(x = x, y = group1, size = size, color = group)) +
geom_point() +
scale_size(name = "Tamaño", range = c(1, 10)) +
theme_bw() +Base de datos en formato ancho
theme(
panel.grid = element_blank(), # Eliminar la cuadrícula del fondo
axis.ticks.y = element_blank(), # Eliminar las marcas del eje y
axis.text.x = element_text(size = 12), # Ajustar el tamaño del texto en el eje x
) + geom_vline(xintercept = c(2.5, 5, 7.5), color = "gray") + # Líneas verticales en los valores 2.5, 5 y 7.5
guides(
color = guide_legend(title = NULL,
override.aes = list(size = 3)), # Eliminar el título de la leyenda para "group"
size = guide_legend(title = NULL,
override.aes = list(fill = NA, color = "black")) # Modificar la leyenda del tamaño de los círculos
)# Gráfico mejorado
ggplot(datos, aes(x = x, y = group1, size = size, color = group)) +
geom_point() +
scale_size(name = "Tamaño", range = c(1, 10)) +
theme_bw() + theme(
panel.grid = element_blank(), # Eliminar la cuadrícula del fondo
axis.ticks.y = element_blank(), # Eliminar las marcas del eje y
axis.text.x = element_text(size = 12),# Ajustar el tamaño del texto en el eje x
) + geom_vline(xintercept = c(2.5, 5, 7.5), color = "gray") + # Líneas verticales en los valores 2.5, 5 y 7.5
guides(
color = guide_legend(title = NULL,
override.aes = list(size = 3)), # Eliminar el título de la leyenda para "group"
size = guide_legend(title = NULL,
override.aes = list(shape = 1, color = "black")) # Modificar la leyenda del tamaño de los círculos
)# Gráfico mejorado
ggplot(datos, aes(x = x, y = group1, size = size, color = group)) +
geom_point() +
scale_size(name = "Tamaño", range = c(1, 10)) +
theme_bw() +
theme(
panel.grid = element_blank(), # Eliminar la cuadrícula del fondo
axis.ticks.y = element_blank(), # Eliminar las marcas del eje y
axis.text.x = element_text(size = 12), # Ajustar el tamaño del texto en el eje x
axis.title.x = element_blank(), # Quitar el título del eje x
axis.title.y = element_blank(), # Quitar el título del eje y
legend.position = "right", # Colocar las leyendas a la derecha
legend.direction = "vertical" # Asegurarse que las leyendas estén en orientación vertical
) + geom_vline(xintercept = c(2.5, 5, 7.5), color = "gray") + # Líneas verticales en los valores 2.5, 5 y 7.5
guides(
color = guide_legend(title = NULL,
override.aes = list(size = 5)), # Eliminar el título de la leyenda para "group"
size = guide_legend(title = NULL,
override.aes = list(shape = 1, color = "black", stroke = 1.2)) # Aumentar el grosor del contorno de los círculos en la leyenda
)# Gráfico mejorado
ggplot(datos, aes(x = x, y = group1, size = size, color = group)) +
geom_point(alpha=0.7) + # Transparecia de los colores
scale_size(name = "Tamaño", range = c(1, 10)) +
scale_color_viridis(discrete=TRUE, guide=FALSE) + # Escala de colores
theme_bw() +
theme(
panel.grid = element_blank(), # Eliminar la cuadrícula del fondo
axis.ticks.y = element_line(color = "black"), # Añadir marcas de graduación en el eje y
axis.text.x = element_text(size = 12), # Ajustar el tamaño del texto en el eje x
axis.text.y = element_text(size = 12), # Ajustar el tamaño del texto en el eje y
axis.title.x = element_blank(), # Quitar el título del eje x
axis.title.y = element_blank(), # Quitar el título del eje y
legend.position = c(0.95, 0.01), # Colocar las leyendas en la parte inferior derecha
legend.justification = c(1, 0), # Ajustar la justificación de las leyendas
legend.direction = "vertical", # Colocar las leyendas en orientación vertical
plot.margin = margin(15, 20, 15, 20), # Aumentar márgenes para evitar solapamiento
) +
geom_vline(xintercept = c(2.5, 5, 7.5), color = "gray") + # Líneas verticales en los valores 2.5, 5 y 7.5
guides(
size = guide_legend(title = NULL,
override.aes = list(shape = 1, color = "#377eb8", stroke = 1.2)), # Aumentar el grosor del contorno de los círculos en la leyenda
color = guide_legend(title = NULL,
override.aes = list(size = 7)) # Eliminar el título de la leyenda para "group"
)2. PLANCTON ESTUARINO
# Cargar datos desde Excel
biol <- read_xlsx("plancton.xlsx", sheet = "Riqueza")
# Explorar estructura
# glimpse(biol)
# summary(biol)
# tabla con los datos
head(biol) %>%
kbl() %>%
kable_classic(full_width = F)# Organización de la base de datos
biol <-
biol %>%
# Convertir variables a factores
mutate(across(c(Station, Size, Layers, Groups), as.factor)) %>%
# Abreviar nombres de los grupos taxonómicos
mutate(Abrev = abbreviate(Groups, minlength = 4)) %>%
# Reorganizar las columnas
select(Station, Size, Layers, Temperature, Salinity, Density, Groups, Abrev, Abundance)Burbujas con leyenda afuera (Salinidad)
# Organización de la base de datos
biol <-
biol %>%
# Convertir variables a factores
mutate(across(c(Station, Size, Layers, Groups), as.factor)) %>%
# Abreviar nombres de los grupos taxonómicos
mutate(Abrev = abbreviate(Groups, minlength = 4)) %>%
# Reorganizar las columnas
select(Station, Size, Layers, Temperature, Salinity, Density,
Groups, Abrev, Abundance)# Calcular la Abundancia total por grupo y ordenar taxones
orden_abundancia <-
biol %>%
group_by(Groups) %>%
summarise(Total_Abundance = sum(Abundance, na.rm = TRUE)) %>%
arrange(desc(Total_Abundance)) %>%
pull(Groups)Burbujas con leyenda afuera (Salinidad)
# Aplicar el orden a la variable categórica (invirtiendo el orden para que los más abundantes estén arriba)
biol <- biol %>%
mutate(Groups = factor(Groups, levels = rev(orden_abundancia)),
Abrev = factor(Abrev,
levels = rev(abbreviate(orden_abundancia, minlength = 4))))# Calcular la Abundancia total por grupo y ordenar taxones
orden_abundancia <-
biol %>%
group_by(Groups) %>%
summarise(Total_Abundance = sum(Abundance, na.rm = TRUE)) %>%
arrange(desc(Total_Abundance)) %>%
pull(Groups)Burbujas con leyenda afuera (Salinidad)
# Crear el gráfico de burbujas
ggplot(biol, aes(x = Salinity, y = Abrev, size = Abundance, color = Station)) +
geom_point(alpha = 0.7) + # Transparencia para mejor visualización
scale_size(name = "Abundancia", range = c(1, 8)) +
scale_x_continuous(limits = c(10, 35), breaks = seq(10, 35, by = 5)) +
theme_bw() +theme(
panel.grid = element_blank(),
axis.ticks.y = element_blank(),
axis.text.x = element_text(size = 12),
axis.title.x = element_blank(),
axis.title.y = element_blank(),
legend.position = "right",
legend.direction = "vertical"
) + # Líneas verticales grises
geom_vline(xintercept = seq(10, 35, by = 5), color = "gray") +
# Circulos de la abundancia en la leyenda
guides(
size = guide_legend(title = NULL,
override.aes = list(shape = 1,
color = "#377eb8",
stroke = 1.2)),
# Circulos de las Layers en la leyenda
color = guide_legend(title = NULL, # Eliminar el título de la leyenda para "group"
override.aes = list(size = 7))
)Burbujas con leyenda afuera (Salinidad)
# Crear el gráfico de burbujas
ggplot(biol, aes(x = Salinity, y = Abrev, size = Abundance, color = Station)) +
geom_point(alpha = 0.7) + # Transparencia para mejor visualización
scale_size(name = "Abundancia", range = c(1, 8)) +
scale_x_continuous(limits = c(10, 35), breaks = seq(10, 35, by = 5)) +
scale_color_viridis(discrete=TRUE, guide=FALSE) + # Escala de colores
theme_bw() + theme(
panel.grid = element_blank(), # Eliminar la cuadrícula del fondo
axis.ticks.y = element_line(color = "black"), # Añadir marcas de graduación en el eje y
axis.text.x = element_text(size = 8), # Ajustar el tamaño del texto en el eje x
axis.text.y = element_text(size = 8), # Ajustar el tamaño del texto en el eje y
axis.title.x = element_blank(), # Quitar el título del eje x
axis.title.y = element_blank(), # Quitar el título del eje y
legend.position = c(0.2, 0.01), # Colocar las leyendas en la parte inferior derecha
legend.justification = c(1, 0), # Ajustar la justificación de las leyendas
legend.direction = "vertical", # Colocar las leyendas en orientación vertical
plot.margin = margin(15, 20, 15, 20), # Aumentar márgenes para evitar solapamiento
) +Burbujas con leyenda adentro (Salinidad)
# Líneas verticales grises
geom_vline(xintercept = seq(10, 35, by = 5), color = "gray") +
# Circulos de la abundancia en la leyenda
guides(
size = guide_legend(title = NULL,
override.aes = list(shape = 1,
color = "#377eb8",
stroke = 1.2)),
# Circulos de las Layers en la leyenda
color = guide_legend(title = NULL, # Eliminar el título de la leyenda para "group"
override.aes = list(size = 7))
)Burbujas con leyenda adentro (Salinidad)
# Organización de la base de datos
biol <-
biol %>%
# Convertir variables a factores
mutate(across(c(Station, Size, Layers, Groups), as.factor)) %>%
# Abreviar nombres de los grupos taxonómicos
mutate(Abrev = abbreviate(Groups, minlength = 4)) %>%
# Reorganizar las columnas
select(Station, Size, Layers, Temperature, Salinity,
Density, Groups, Abrev, Abundance)
# Calcular la Abundancia total por grupo y ordenar taxones
orden_abundancia <-
biol %>%
group_by(Groups) %>%
summarise(Total_Abundance = sum(Abundance, na.rm = TRUE)) %>%
arrange(desc(Total_Abundance)) %>%
pull(Groups)
# Aplicar el orden a la variable categórica (invirtiendo el orden para que los más abundantes estén arriba)
biol <-
biol %>%
mutate(Groups = factor(Groups, levels = rev(orden_abundancia)),
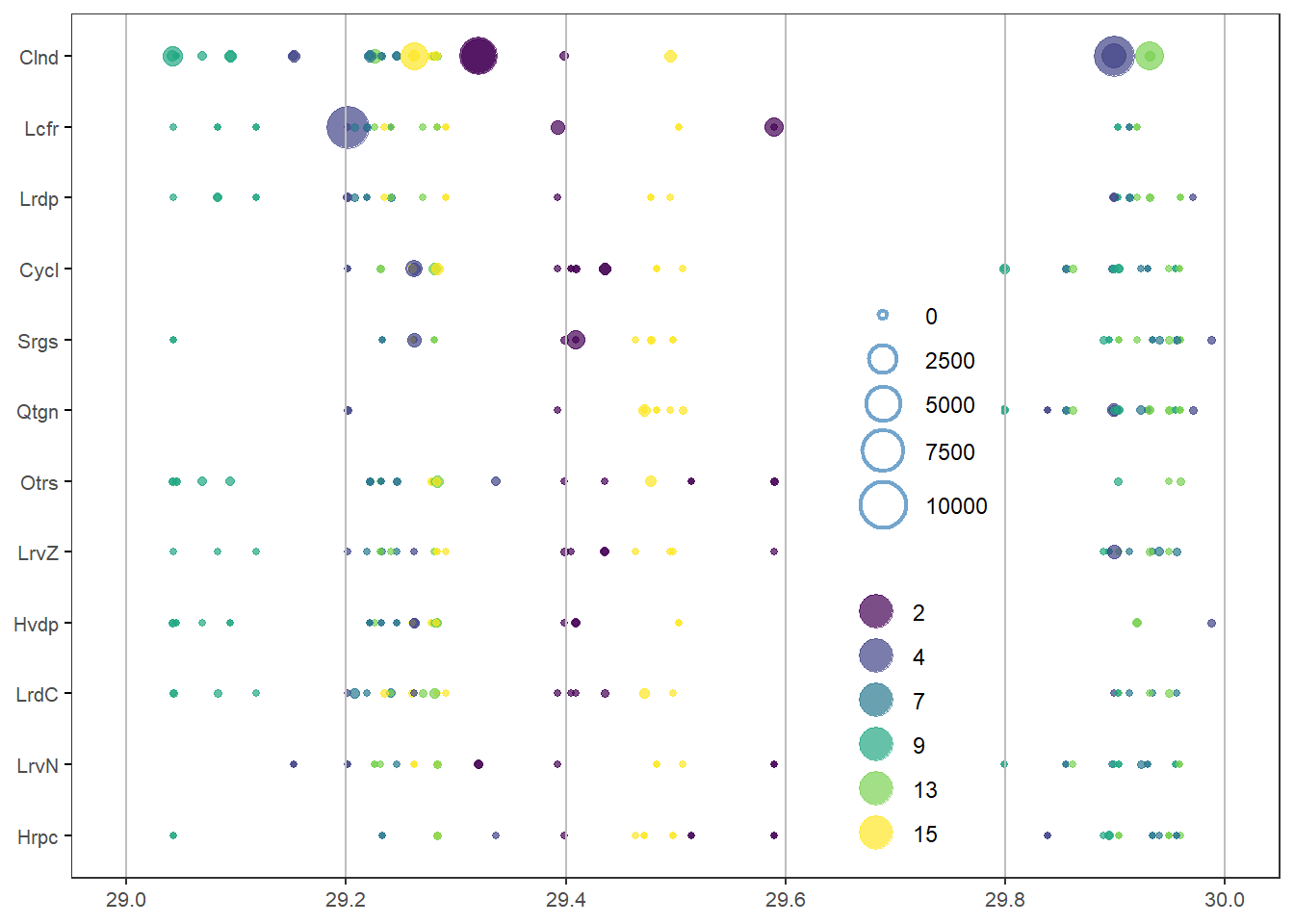
Abrev = factor(Abrev, levels = rev(abbreviate(orden_abundancia, minlength = 4))))Burbujas con leyenda adentro (Temperatura)
# Crear el gráfico de burbujas con Temperature en el eje X
ggplot(biol, aes(x = Temperature, y = Abrev, size = Abundance, color = Station)) +
geom_point(alpha = 0.7) + # Transparencia para mejor visualización
scale_size(name = "Abundancia", range = c(1, 8)) +
scale_x_continuous(limits = c(29, 30),
breaks = seq(29, 30, by = 0.2)) + # Límites y gradación del eje x
scale_color_viridis(discrete=TRUE, guide=FALSE) + # Escala de colores
theme_bw() +
theme(
panel.grid = element_blank(), # Eliminar la cuadrícula del fondo
axis.ticks.y = element_line(color = "black"), # Añadir marcas de graduación en el eje y
axis.text.x = element_text(size = 8), # Ajustar el tamaño del texto en el eje x
axis.text.y = element_text(size = 8), # Ajustar el tamaño del texto en el eje y
axis.title.x = element_blank(), # Quitar el título del eje x
axis.title.y = element_blank(), # Quitar el título del eje y
legend.position = c(0.77, 0.01), # Colocar las leyendas en la parte inferior derecha
legend.justification = c(1, 0), # Ajustar la justificación de las leyendas
legend.direction = "vertical" # Colocar las leyendas en orientación vertical
) +
# Líneas verticales grises
geom_vline(xintercept = seq(floor(min(biol$Temperature, na.rm = TRUE)),
ceiling(max(biol$Temperature, na.rm = TRUE)),
by = 0.2), color = "gray") + # Intervalos para las líneas verticales
# Círculos de la abundancia en la leyenda
guides(
size = guide_legend(title = NULL,
override.aes = list(shape = 1,
color = "#377eb8",
stroke = 1.2)),
# Círculos de las Layers en la leyenda
color = guide_legend(title = NULL, # Eliminar el título de la leyenda para "group"
override.aes = list(size = 6))
)Burbujas con leyenda adentro (Temperatura)
library(plotly)
# Crear el gráfico de burbujas con Temperature en el eje X
fig <-
ggplot(biol, aes(x = Temperature, y = Abrev, size = Abundance, color = Station)) +
geom_point(alpha = 0.7) + # Transparencia para mejor visualización
scale_size(name = "Abundancia", range = c(1, 8)) +
scale_x_continuous(limits = c(29, 30),
breaks = seq(29, 30, by = 0.2)) + # Límites y gradación del eje x
scale_color_viridis(discrete=TRUE, guide=FALSE) + # Escala de colores
theme_bw() +
theme(
panel.grid = element_blank(), # Eliminar la cuadrícula del fondo
axis.ticks.y = element_line(color = "black"), # Añadir marcas de graduación en el eje y
axis.text.x = element_text(size = 8), # Ajustar el tamaño del texto en el eje x
axis.text.y = element_text(size = 8), # Ajustar el tamaño del texto en el eje y
axis.title.x = element_blank(), # Quitar el título del eje x
axis.title.y = element_blank(), # Quitar el título del eje y
legend.position = c(0.77, 0.01), # Colocar las leyendas en la parte inferior derecha
legend.justification = c(1, 0), # Ajustar la justificación de las leyendas
legend.direction = "vertical" # Colocar las leyendas en orientación vertical
) +
# Líneas verticales grises
geom_vline(xintercept = seq(floor(min(biol$Temperature, na.rm = TRUE)),
ceiling(max(biol$Temperature, na.rm = TRUE)),
by = 0.2), color = "gray") + # Intervalos para las líneas verticales
# Círculos de la abundancia en la leyenda
guides(
size = guide_legend(title = NULL,
override.aes = list(shape = 1,
color = "#377eb8",
stroke = 1.2)),
# Círculos de las Layers en la leyenda
color = guide_legend(title = NULL, # Eliminar el título de la leyenda para "group"
override.aes = list(size = 6))
)
ggplotly(fig) Burbujas Animación
TALLER DE ENTRENAMIENTO
Objetivo 1: Poner en práctica los conceptos vistos en esta opción de exploración con burbujas, incorporando los siguientes requisitos en el estudio de caso:
-
Organización de los datos taxonómicos en formato largo, junto a las variables ambientales.
-
Figuras de burbujas con las principales variables ambientales identificadas.
TALLER DE ENTRENAMIENTO
Objetivo 2: Realizar el cuestioario.
2.3 Exploratorios burbujas
By Javier Rodriguez
2.3 Exploratorios burbujas
- 20



