EXPLORATORIOS EN R
Javier Rodríguez Barrios
MSc. Multivariados
Taller 2.2 Explor. Plancton
# Librerías requeridas
library(tidyverse)
library(dplyr)
library(kableExtra) # Para la edición de tablas
library(readxl) # Cargar bases de Excel
require(stats) # No se requiere instalar
library(lattice) # No se requiere instalar
library(ggrepel) # insertar rótulos a los puntos
require(SciViews) # Fig. dispersión con coef. de pearson
library(plotrix) # Figuras de cajas con múltiples variables
library(corrplot) # Figuras de elipses
library(psych) # Matrices de correlación para figuras de elipses
library(reshape) # Figuras de cajas con múltiples variables
library(reshape2) # Figuras de cajas con múltiples variables
library(gridExtra) # Para figuras estadísticas (varios factores)
library(grid) # Para figuras estadísticas (varios factores)
library(ggplot2) # Paquete gráfico
library(forcats) # Para manipulación de factores
library(viridis) # Opciones de paletas de colores# Cargar datos desde Excel
biol <- read_xlsx("plancton.xlsx", sheet = "Riqueza")
# Explorar estructura
# glimpse(biol)
# summary(biol)
# tabla con los datos
head(biol) %>%
kbl() %>%
kable_classic(full_width = F)biol1 <-
biol %>%
# Abreviaturas de los taxones
mutate(Abrev = abbreviate(Groups, minlength = 4)) %>%
# Variables a factores
mutate(across(c(Station, Size, Layers), as.factor)) %>%
# Agrupamiento para el formato ancho
group_by(Station, Size, Layers) %>%Base de datos en formato ancho
# Promedios de las variables ambientales
summarize(
across(c(Temperature, Salinity, Density), ~round(mean(.),2)),
# Totales de las abundancias por cada factor
Abundance = list(setNames(tapply(Abundance, Abrev,sum,
default = 0), unique(Abrev))),
# Corregir algunos errores del agrupamiento
.groups = "drop") %>% # Separar las abundancias en las columnas de cada taxon
unnest_wider(Abundance) %>%
# Crear columna Ref, tomando iniciales de tres factores
mutate(
Ref = paste0(substr(Station, 1, 2),
substr(Size, 1, 1),
substr(Layers, 1, 1))) %>% # Pasar la columna de referencia (consec) a la 1a columna
select(Ref, everything()) %>%
# Crear la columna Ab con la suma de las columnas de taxones
mutate(
# Suma de las columnas especificadas (Ab)
Ab = rowSums(across(Qtgn:Otrs), na.rm = TRUE)
) %>% Base de datos en formato ancho
# Mover la columna de abundancias (AB)
select(Ref,Station,Size,Layers,Temperature,
Salinity,Density,Ab, everything()) # tabla con los datos
head(biol1) %>%
kbl() %>%
kable_classic(full_width = F)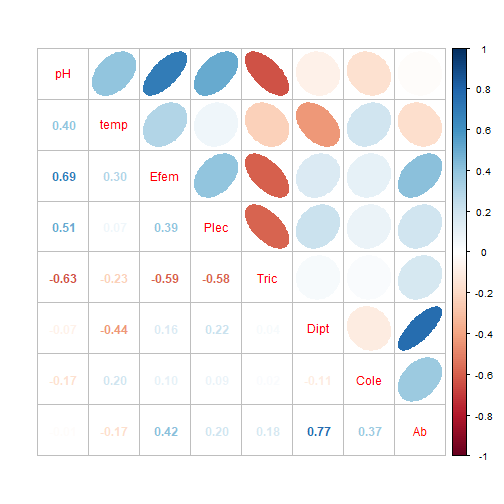
1. ELIPSES
# Elipses con colores
M <- cor(biol1[,...]) # Matriz de Correlación (M)# Elipses con colores
corrplot(M, method = "ellipse") # Figura de correlaciones con elipses# Elipses con colores
corrplot(M, method = "circle") # Figura de correlaciones con circulos# Elipses con colores
corrplot.mixed(M, upper="ellipse")# Elipses con colores
M1 <- cor(biol1[,5:7], biol1[,8:20]) # Matriz de Correlación (M)
# Elipses con colores
corrplot(M1, method = "ellipse", type="upper")OPCIONES DE ELIPSES
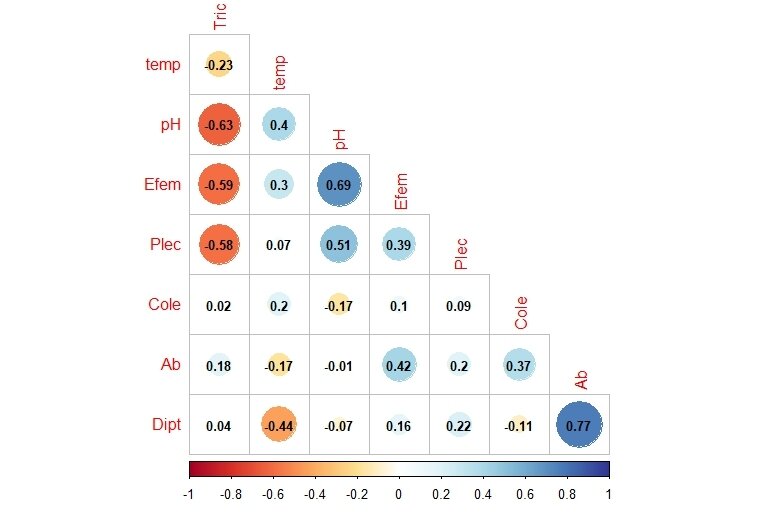
# Figura de elipses con coeficientes de correlación
corrplot(M, method = "circle", # Correlaciones con circulos
type = "lower", insig="blank", # Forma del panel
order = "AOE", diag = FALSE, # Ordenar por nivel de correlación
addCoef.col ="black", # Color de los coeficientes
number.cex = 0.8, # Tamaño del texto
col = COL2("RdYlBu", 200)) # Transparencia de los circulos2. DISPERSIÓN
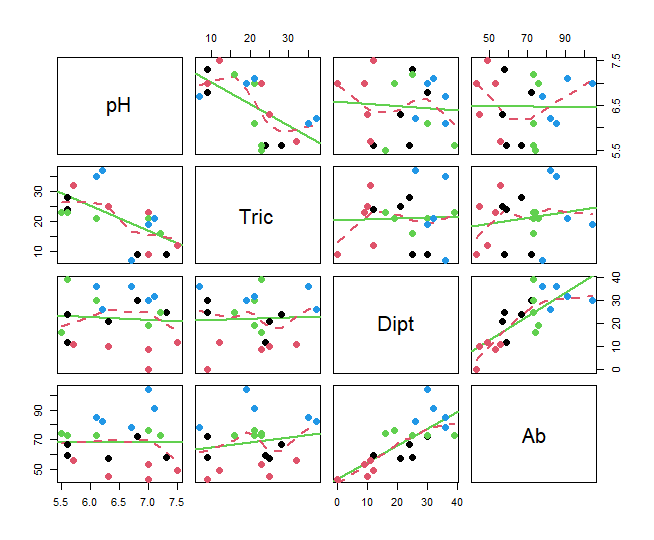
# Correlaciones de Pearson
library(SciViews)
pairs(biol1[,c(5:7)], diag.panel = panel.hist,
upper.panel = panel.cor, lower.panel = panel.smooth)3. HISTOGRAMAS
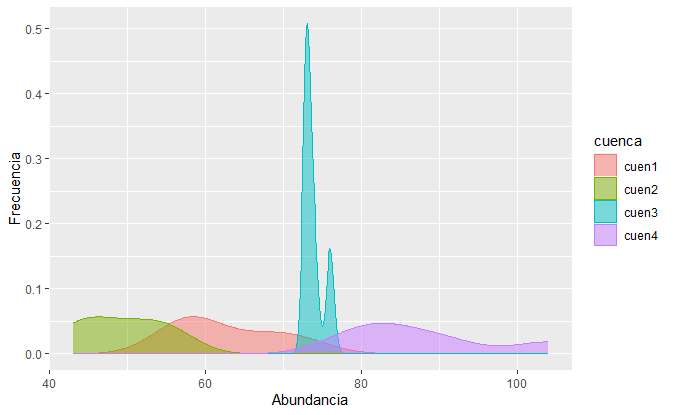
# Frecuencias de abundancias por capas
ggplot(data = biol1, aes(x = Ab, color = Layers)) +
geom_density(aes(fill = Layers), alpha = 0.5) +
labs( y="Frecuencia", x="Abundancia") +
theme_bw() +
theme(panel.grid = element_blank()
)# Otra opción
ggplot(data = biol1, aes(x = Ab, color = Layers)) +
geom_density(aes(fill = Layers)) +
facet_wrap(~ Size) +
theme_bw() +
theme(panel.grid = element_blank()
)
4. DISPERSIÓN X-Y
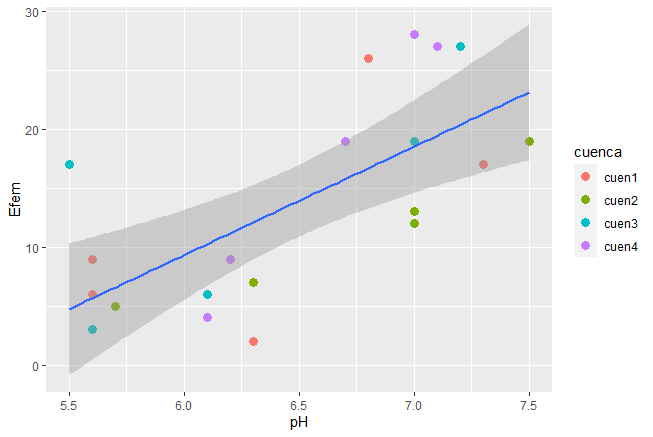
# Regresiones lineales (Esquema ggplot2)
# ***Ejercicio*** Graficar la relación entre el pH y los efemerópteros
ggplot(datos, ... ) +
geom_point(aes(color = Layers), size = 3) +
geom_smooth(method= "lm") +
theme_bw() +
theme(panel.grid = element_blank()
)# Regresiones suavizadas - Loess o Lowess (Esquema ggplot2)
ggplot(biol1,aes(x = Density, y = Ab)) +
geom_point(aes(color = Layers), size = 3) +
geom_smooth() +
theme_bw() +
theme(panel.grid = element_blank()
)# Regresiones suavizadas (Loess)
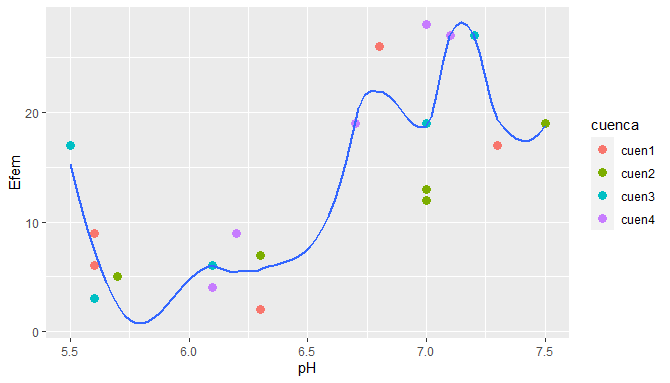
ggplot(biol1,aes(x = Density, y = Ab)) +
geom_point(aes(color = Layers), size = 3) +
geom_smooth(se = FALSE, span = 0.5) +
theme_bw() +
theme(panel.grid = element_blank()
)
CAJAS Y BIGOTES
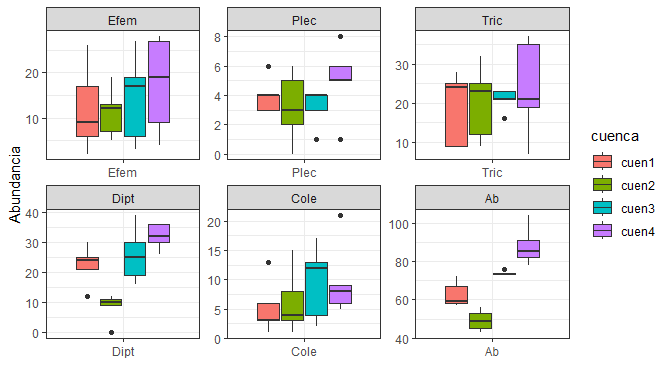
Cajas con la biblioteca "ggplot2"
# Convertir variables a factores en caso que se requiera
biol1 <-
biol1 %>%
mutate(across(c(Station, Size, Layers), as.factor))
Cajas con un factor: Estaciones.
# Gráfico de caja de la abundancia por estación
ggplot(biol, aes(x = factor(...), y = ...)) +
geom_boxplot(aes(fill = factor(Station))) +
# Aplicar la transformación logarítmica
scale_fill_manual(values = c('#fc8d59','#ffffbf','#99d594',
'#377eb8','#e78ac3','#7570b3')) +
scale_y_continuous(trans = "log10") +
labs(title = "Distribución de la Abundancia por Estación",
x = "Estaciones", fill = "Estaciones",
y = expression(log[10]~(Abundancia~indv.~m^-3))
) +
theme_bw() +
theme(
panel.gridr = element_blank()
)library(forcats) # Para manipulación de factores
# Cambiar etiquetas de Layers con recode_factor()
biol <-
biol %>%
mutate(Layers = recode_factor(Layers,
"Depth" = "Profunda",
"Surface" = "Superficial"))Cajas con dos factores: Estaciones y capas (base biol*).
# Gráfico de caja de la abundancia por estación
ggplot(biol, aes(x = factor(...), y = ...)) +
geom_boxplot(aes(fill = Layers)) +
scale_y_continuous(trans = "log10") + # Aplicar la transformación logarítmica
labs(title = "Distribución de la Abundancia por Estación",
x = "Estaciones", fill = "Capas",
y = expression(log[10]~(Abundancia~indv.~m^-3))
) +
theme_bw()+
theme(
panel.grid = element_blank()
)Cajas con dos factores: Estaciones y capas (base biol1*).
# Gráfico de caja de la abundancia por estación
ggplot(biol1, aes(x = factor(...), y = ...)) +
geom_boxplot(aes(fill = ...)) +
# Aplicar la transformación logarítmica
scale_y_continuous(trans = "log10") +
labs(title = "Distribución de la Abundancia por Estación",
x = "Estaciones", fill = "Capas",
y = expression(log[10]~(Abundancia~indv.~m^-3))
) +
theme_bw()+
theme(
panel.grid = element_blank()
)
# facet_wrap(~ Layers, scales = "free") # Paneles por variable# Figuras multivariadas de Cajas y bigotes
ggplot(melt(biol1[,c(..., ...)]), aes(x=Station, y=value)) + # Usar Station en el eje X
geom_boxplot(aes(fill=Station)) +
scale_y_continuous(trans = "log10") + # Aplicar la transformación logarítmica
scale_color_viridis(discrete = TRUE) +
labs(title = "Distribución de la Abundancia por Estación",
x = "Estaciones", fill = "Estaciones",
y = expression(log[10]~(Abundancia~indv.~m^-3))
) +
facet_wrap(~ variable, scales = "free") + # Paneles por variable
theme_bw() +
theme(
panel.grid = element_blank()
)Cajas con multiples variables (melt).
# Categorización de la temperatura
biol <-
... %>%
mutate(claseTemp = case_when(
... <= quantile..., 1/3, na.rm = TRUE) ~ "T.Baja",
... <= quantile(Temperature, 2/3, na.rm = TRUE) ~ "T.Media",
... <= quantile(Temperature, 3/3, na.rm = TRUE) ~ "T.Alta"
))
# Categorización de la salinidad
biol <-
... %>%
mutate(claseSal = case_when(
... <= quantile(..., 1/3, na.rm = TRUE) ~ "S.Baja",
... <= quantile(Salinity, 2/3, na.rm = TRUE) ~ "S.Media",
... <= quantile(Salinity, 3/3, na.rm = TRUE) ~ "S.Alta"
))Cajas con variables contínuas categorizadas
# Crear gráfico con etiquetas de valores atípicos redondeados
ggplot(biol, aes(x = factor(Station), y = Abundance)) +
geom_boxplot(aes(fill = claseTemp)) +
labs(title = "Abundancia por Estación, salinidad y Temperatura",
x = "Estaciones", fill = "Temperatura",
y = expression(log[10]~(Abundancia~indv.~m^-3))
) +
scale_y_continuous(trans = "log10") + # Aplicar la transformación logarítmica
scale_color_viridis(discrete = TRUE) +
facet_wrap(~ claseSal, nrow = 1, strip.position = "top") +
theme_bw() +
theme(
panel.grid = element_blank()
) Cajas con variables contínuas categorizadas
7. FIGURAS CON ESTADÍSTICOS
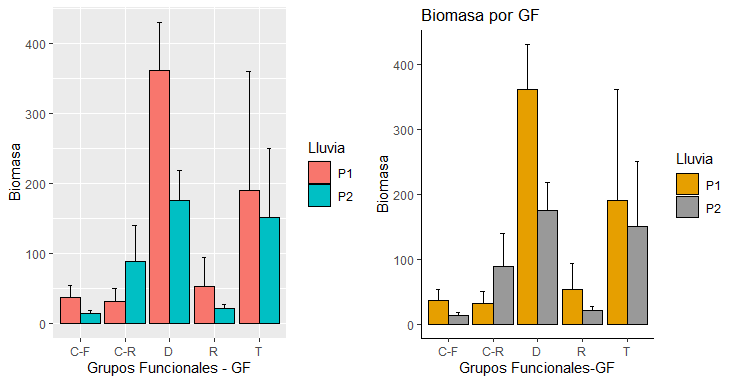
# Resumen estadístico "datos_resum"
datos_resum <-
biol1 %>% # Base de datos resumida
group_by(Size,Layers) %>% # Factor o variable agrupadora
summarise(datos.m = mean(Ab), # Media de cada grupo del factor
datos.de = sd(Ab), # Desviacioes estándar de cada grupo
datos.var = var(Ab), # Varianzas de cada grupo
n.Biom = n(), # Tamaño de cada grupo
datos.ee = sd(Ab)/sqrt(n())) # Error estándar de cada grupo
# tabla con los datos
head(datos_resum) %>%
kbl() %>%
kable_classic(full_width = F)
Figuras estadísticas de dos factores
# Figura 1 (f1)
f1 = ggplot(datos_resum, aes(x=Layers, y=datos.m, fill=Size)) +
geom_bar(stat="identity", color="black",
position=position_dodge()) +
geom_errorbar(aes(ymin=datos.m, ymax=datos.m+datos.de), width=.2,
position=position_dodge(.9)) # f2: Otro formato de figura bifactorial - theme_classic
f2 = f1+labs(title="Abundancia por Capas",
x="Capas de prof.",
y = "Abundancia")+
theme_classic() +
scale_fill_manual(values=c('#E69F00','#999999'))
# Impresión de un panel con las dos figuras de forma horizontal (f1 y f2)
grid.arrange (f1, f2, ncol=2)
# Impresión de un panel con las dos figuras de forma vertical (p1 y p2)
grid.arrange (f1, f2, nrow=2)TALLER DE ENTRENAMIENTO
Objetivo 1: Poner en práctica los conceptos vistos en el módulo de exploratorios multivariados, realizando las siguientes opciones gráficas en las bases de datos asignadas para los estdios de caso:
-
Figuras de elipses
-
Figuras de Dispersión por pares de variables (pairs)
-
Histogramas
-
Dispersión X-Y
-
Cajas y Bigotes
-
Coplot
-
Figuras con estadísticos (promedios, errores, …)
TALLER DE ENTRENAMIENTO
Objetivo 2: Poner en práctica lo anterior con la base de datos de lirios `"data(iris)"`
-
Figuras de elipses
-
Figuras de Dispersión por pares de variables (pairs)
-
Histogramas
-
Dispersión X-Y
-
Cajas y Bigotes
-
Coplot
-
Figuras con estadísticos (promedios, errores, …)
2.2 Exploratorios Resum
By Javier Rodriguez
2.2 Exploratorios Resum
- 19



