Wykorzystanie metod analizy brzegowej oraz obliczeń w modelu bezserwerowym na przykładzie procesu "basecallingu"
Piotr Grzesik
dr hab. inż. Dariusz Mrozek, prof. PŚ
Politechnika Śląska
Agenda
- Sekwencjonowanie nanoporowe
- MinION Nanopore
- Wyzwania związane z przetwarzaniem brzegowym
- Rozpatrywane zadanie
- Podejście brzegowe
- Przetwarzanie bezserwerowe
- Podejście bezserwerowe
- Podejście hybrydowe
- Podsumowanie
Sekwencjonowanie nanoporowe
Sekwencjonowanie nanoporowe - metoda sekwencjonowania DNA, opracowana przez Oxford Nanopore Technologies. Polega na monitorowaniu zmian natężenia prądu spowodowanego przez nić DNA/RNA przesuwającą się przez nanopory. Otrzymany sygnał jest następnie dekodowany do konkretnych sekwencji DNA/RNA. Proces ten nazywamy basecalling.

Sekwencjonowanie nanoporowe
MinION Nanopore
MinION Nanopore - przenośne urządzenie sekwencjonujące, udostępnione przez firmę Oxford Nanopore Technologies w 2014 roku. Jest to pierwsze z urządzeń pozwalające na prowadzenie eksperymentów sekwencjonowania w "przenośny" sposób w przystępnej cenie (~$1000). Jest zasilane przez USB, waży ok. 100g, co pozwala na wykorzystanie tego urządzenia w "terenie".
MinION Nanopore

Przetwarzanie brzegowe
Przetwarzanie brzegowe (ang. edge computing) - jest to podejście programistyczne, które polega na przeniesieniu procesów przetwarzania oraz przechowywania danych bliżej źródła danych. Pozwala to na zredukowanie wolumenu danych przesyłanych np. przez sieć Internet, pozwala na zmniejszenie czasu reakcji systemu na zmieniający się stan, a także poprawia odporność systemu w przypadku zawodnego połączenia z centrami danych.
Wyzwania związane z przetwarzaniem brzegowym
- Złożoność obliczeniowa
- Wolumen danych, które należy przetworzyć
- Ograniczony dostęp do sieci
- Ograniczony dostęp do źródeł energii
Rozpatrywane zadanie
Rozpatrywane zadanie składa się z trzech kroków - sekwencjonowania, basecallingu, oraz klasyfikacji. W pierwszym kroku, MinION Nanopore sekwencjonuje DNA, czego rezultatem są odczyty natężenia prądu w formacie FAST5. W drugim kroku, basecaller interpretuje dane z plików FAST, czego rezultatem są sekwencje DNA w postaci plików FASTA. W ostanim kroku, sekwencje te są klasyfikowane oraz identyfikowane.

Srodowisko testowe

Srodowisko testowe
-
Symulator MinION Nanopore, który generuje pliki w formacie fast5
-
Urządzenie brzegowe, które jest odpowiedzialne za proces basecallingu oraz klasyfikacji, wyposażone w dysk SSD
-
Serwis chmurowy, który służy jako magazyn dla sekwencjonowanych danych
-
Zasilacz z układem INA219 oraz Rasperry Pi 4 w celu pomiaru natężenia prądu
Specyfikacja urządzenia brzegowego
- Single board computer - Jetson Xavier NX
-
GPU - NVIDIA Volta™ architecture
with 384 NVIDIA® CUDA® cores and 48 Tensor cores -
6-core NVIDIA Carmel ARM®v8.2 64-bit CPU
6 MB L2 + 4 MB L3 - 8 GB 128-bit LPDDR4x 51.2GB/s
- Storage - SDHC card (32 GB, class 10) + SSD NVMe
- OS - Ubuntu 18.04.5 LTS with kernel version 4.19.140-tegra
Urządzenie brzegowe

Wykorzystane oprogramowanie
- Guppy - Zaawansowany, zamkniętoźródłowy basecalled, opracowany przez Oxford Nanopore Technologies. Oferuje wsparcie dla akceleracji CPU oraz GPU na wybranych urządzeniach. Wspiera architekturę x86_64 oraz AArch64. Może pracować w dwóch trybach, fast (szybkim) oraz hac (dokładnym).
- Kraken 2 - system klasyfikacji taksonomicznej wykorzystujący metodę "exact k-mer match" by osiągnąć wysoką dokładność oraz niski czas klasyfikacji. Ze względu na wysokie zużycie pamięci RAM przez standardową bazę danych, przygotowana została zminimalizowana baza danych, dedykowana identyfikacji wybranych bakterii oraz wirusów.
Eksperymenty
-
Wykorzystano dostępne dane z eksperymentów sekwencjonowania materiału zawierającego Escherichia coli oraz Klebsiella Pneumoniae
-
Zarejestrowano liczbę próbek przeprocesowanych na sekundę dla różnych trybów mocy w Jetson Xavier NX
-
Przetestowano basecallery takie jak Guppy, Bonito, Causalcall, Deepnano, Deepnano-blitz
-
Guppy oraz Bonito przetestowano z oraz bez akceleracji GPU
-
Klasyfikacja przy użyciu Kraken2 również była przetestowana dla różnych trybów mocy w Jetson Xavier NX
-
Dostępne tryby mocy w Jetson Xavier NX: 15W 6 CORE, 15W 4 CORE, 15W 2 CORE, 10W 2 CORE, 10W 4 CORE
Obserwacje
-
Jetson Xavier NX jest urządzeniem, które pozwala na przeprowadzanie tego typu eksperymentów sekwencjonowania i klasyfikacji przy ograniczonym dostępie do sieci i ograniczonych źródłach zasilania
-
Guppy z modelem "fast" pozwala na wsparcie dla basecallingu w czasie rzeczywistym nawet z 3 urządzeń MinION w tym samym czasie
-
Wykorzystanie trybu 10W w Jetson Xavier NX jest bardziej wydajne energetycznie niż wykorzystanie trybu 15W
-
Proces basecallingu bez akceleracji GPU jest niewykonalny na Jetson Xavier NX i podobnych urządzeniach
-
Na obecny moment, jedynym basecallerem, który oferuje wystarczającą wydajność na urządzeniach brzegowych jest Guppy z akceleracją GPU
-
Istnieje konieczność optymalizacji albo ograniczenia zbiorów danych w celu przetwarzania ich na brzegu (baza danych Kraken2)
-
Wiele istniejących narzędzi nie zakłada wsparcia dla architektury AArch64
Przetwarzanie bezserwerowe
Przetwarzanie bezserwerowe (ang. serverless computing) polega na wykorzystaniu prostych, bezstanowych funkcji (stąd alternatywna nazwa Function-as-a-service), które nie wymagają zarządzania, oferują tolerancję awarii, wspierają przetwarzanie równoległe, alokują zasoby tylko gdy zachodzi taka potrzeba, oraz mogą być szybko skalowane. Dodatkową zaletą takiego podejścia jest to, że użytkownicy płacą tylko za faktyczny czas wykonania funkcji.
Przetwarzanie bezserwerowe
Przetwarzanie bezserwerowe stało się w ostatnim czasie popularne w kontekście wykorzystania w bioinformatyce:
- sVEP (Serverless Variant Effect Predictor) - Cloud Native Variant Annotation Pipeline - speedup vs traditional VM-based solutions
- GT-Scan2 - Serverless Target Finder for genome editing - more cost-effective than traditional VM-based solutions
- Serverless all-against-all Pairwise comparison using Smith-Waterman algorithm - both faster and more cost-effective than traditional VM-based solutions
AWS Lambda
- Platforma przetwarzania bezserwerowego, wprowadzona w 2014 przez Amazon Web Services
- Oferuje wsparcie dla JVM, .NET Core, Go, Ruby, Python, Node.js. Dzięki "custom runtimes" jest w stanie wspierać dowolne środowisko uruchomieniowe
- Oferuje łatwą integrację z innymi usługami AWS, takimi jak S3, Kinesis, CloudWatch, API Gateway
- Limit czasu wykonywania - 15 minut
- Alternatywy - Google Cloud Functions, Microsoft Azure Functions, Apache OpenWhisk
Dlaczego AWS Lambda?
- Wsparcie dla maksymalnie 10,240 MB pamięci
- Wsparcie dla 6 wirtualnych rdzeni CPU
- Wsparcie dla kontenerów Docker
- Wsparcie dla funkcji do 10 GB
- Wsparcie dla zestawu instrukcji AVX2
Rozpatrywane zadanie

Rozpatrywane zadanie
W ramach zaproponowanego rozwiązania, pierwszy krok to przesłanie plików FAST5 z MinION Nanopore do usługi S3. W następnym kroku, pierwsza z funkcji Lambda dzieli dostępne pliki FAST5 na równe sekcje. W następnym kroku uruchamia przetwarzanie każdej z sekcji przez oddzielną funkcję Lambda. W tym kroku, każda z funkcji prowadzi proces basecallingu. Po przetworzeniu, rezultat jest ponownie zapisywany w usłudze S3.
Wykorzystane oprogramowanie
- Guppy - Zaawansowany, zamkniętoźródłowy basecalled, opracowany przez Oxford Nanopore Technologies. Oferuje wsparcie dla akceleracji CPU oraz GPU na wybranych urządzeniach. Wspiera architekturę x86_64 oraz AArch64. Może pracować w dwóch trybach, fast (szybkim) oraz hac (dokładnym).
Eksperymenty
-
Wykorzystano dostępne dane z eksperymentów sekwencjonowania materiału zawierającego Escherichia coli oraz Klebsiella Pneumoniae
-
Przetestowano basecallery takie jak Guppy, Bonito, Causalcall, Deepnano, Deepnano-blitz
-
Eksperymenty zostały przeprowadzone dla konfiguracji z 256, 512, 1024, 2048, 4096, 6144, 8192, oraz 10240 MB RAM dla pojedynczej funkcji
-
Zarejestrowane liczbę próbek przeprocesowanych na sekundę oraz na 1 MB pamięci dla modeli fast oraz hac (Guppy)
Obserwacje
- Guppy CPU basecaller oferuje najlepsze wyniki (szybkość)
- Inne basecallery są bardzo wolne (Bonito, Causalcall) lub wymagają głębszych zmian w implementacji (Deepnano-blitz)
- Wsparcie dla kontenerów Docker pozwala na implementację basecallingu przy użyciu AWS Lambda
- Dla testowanych przypadków, osiągnięto skalę 100 funkcji, każda z 6 vCPU w mniej niż 30 sekund
- Dla najwyższej konfiguracji, jedna funkcja jest w stanie przetworzyć ok. 600000 próbek/s, co przy teoretycznym maksimum dla MinION (2,300,000 próbek/s) pozwala przetwarzać dane z jednego MinION z wykorzystaniem 3-4 funkcji
Podejscie hybrydowe
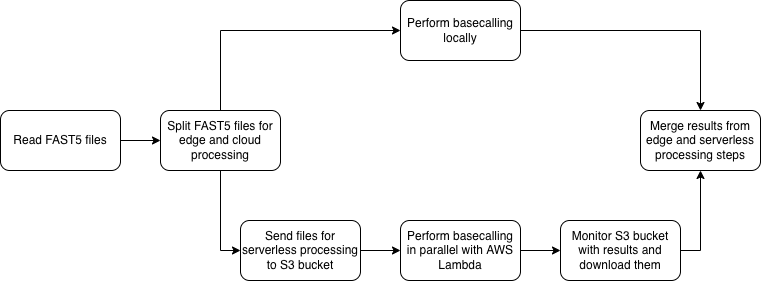
Podejscie hybrydowe
W zaproponowanym podejściu, pierwszy krok służy ocenie, czy przy panujących warunkach istnieje możliwość wykorzystania "cloud offloadingu" do przyspieszenia przetwarzania brzegowego. W następnym kroku, pliki są dzielone na serie, które mają zostać przetworzone na brzegu oraz w chmurze. Podział ten oparty jest na wyznaczonych eksperymentalnie średnich szybkościach przetwarzania dla wybranych prędkości wysyłania. W ostatnim kroku, po przetworzeniu plików, urządzenie brzegowe pobiera rezultaty.
Eksperymenty
-
Wykorzystano dostępne dane z eksperymentów sekwencjonowania materiału zawierającego Escherichia coli oraz Klebsiella Pneumoniae
-
Wykorzystano Jetson Xavier NX jako urządzenie brzegowe, testy prowadzone były dla trybów mocy: 15W 6 CORE oraz 10W 2 CORE
-
W eksperymentach wykorzystano basecaller Guppy (CPU w modelu bezserwerowym, GPU na brzegu)
-
Testy prowadzone były dla prędkości wysyłania 128, 256, oraz 512 kB/s
-
Podczas eksperymentów rejestrowany był całkowity czas przetwarzania dla wybranego zestawu danych, a także całkowita energia zużyta podczas tego przetwarzania
Rezultaty
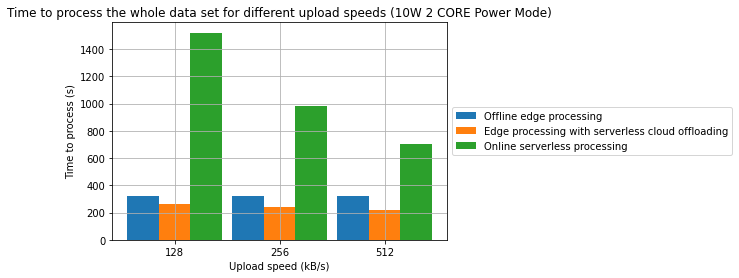
Rezultaty
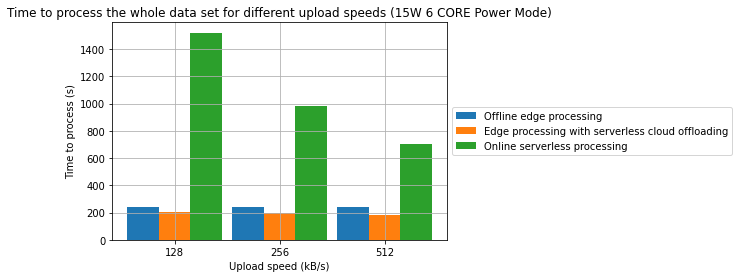
Rezultaty
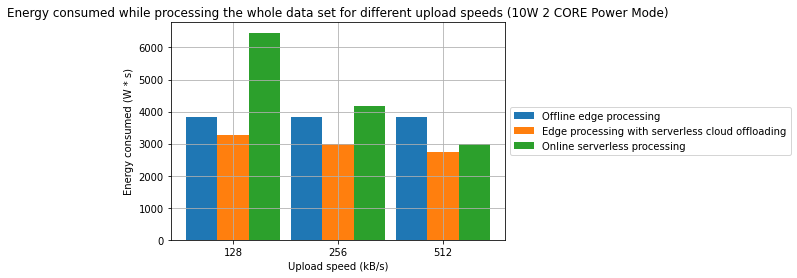
Rezultaty
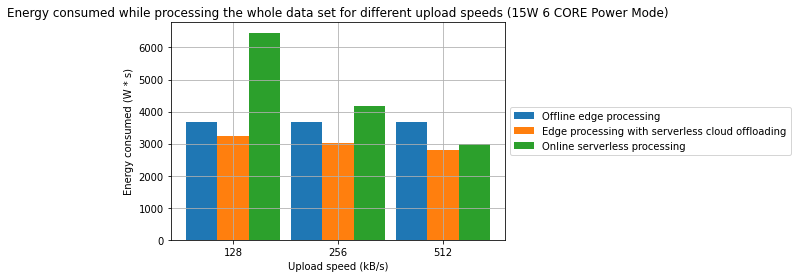
Obserwacje
- Połączenie przetwarzania brzegowego oraz przetwarzania bezserwerowego w środowisku chmurowym może być efektywną strategią pozwalającą na obniżenie konsumpcji energii oraz czasu przetwarzania
- W jednym z badanych przypadków, osiągnięto redukcję czasu przetwarzania o 31.2%. W innym z badanych przypadków osiągnięto redukcję energii o 28.7%. W obu przypadkach punktem odniesienia jest przetwarzanie wyłącznie w modelu brzegowym.
- Zaobserowano również, że w wybranych przypadkach, przetwarzanie w pełni w chmurze może być bardziej wydajne energetycznie niż jakakolwiek forma przetwarzania brzegowego
Copy of [CONF VERSION] Accelerating Edge Metagenomic Analysis with Serverless-based Cloud Offloading
By progressive
Copy of [CONF VERSION] Accelerating Edge Metagenomic Analysis with Serverless-based Cloud Offloading
- 317



