EXPLORATORIOS EN R
Javier Rodríguez Barrios
Visualización de datos en R
# Librerías requeridas
library(tidyverse)
library(xtable) # Importar y exportar
library(openxlsx) # exportar "*.xlsx"
library(readxl) # Importar y exportar
library(gtable) # Importar y exportar
require(stats) # No se requiere instalar
library(lattice) # No se requiere instalar
library(ggrepel) # insertar rótulos a los puntos
require(SciViews) # Fig. dispersión con coef. de pearson
library(plotrix) # Figuras de cajas con múltiples variables
library(corrplot) # Figuras de elipses
library(psych) # Matrices de correlación para figuras de elipses
library(GGally) # Requerido para figura de pares con ggpairs
library(gganimate) # Para figuras animadas de dispersión
library(reshape) # Figuras de cajas con múltiples variables
library(gridExtra) # Para figuras estadísticas (varios factores)
library(grid) # Para figuras estadísticas (varios factores)#------------
datos <- read_excel("Insectos.xlsx") # paquete "readxl"
head(datos)Lectura de la Base de datos
#------------
datos <- read_csv2("Insectos.csv") # paquete "readxl"
head(datos)datos <- read.csv2("Insectos.csv") # paquete "utils"
head(datos)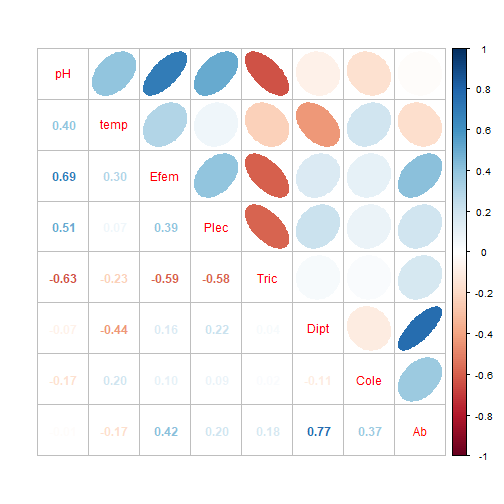
1. ELIPSES
# Elipses con colores
M <- cor(datos[,3:10]) # Matriz de Correlación (M)
# Elipses con colores
X11()
corrplot(M, method = "ellipse") # Figura de correlaciones con elipses# Elipses con colores
X11()
corrplot(M, method = "circle") # Figura de correlaciones con circulos# Elipses con colores
X11()
corrplot.mixed(M, upper="ellipse")# Elipses con colores
x11()
par(mfrow = c(1,2), mar = c(3, 3, 2, 1))
corrplot(M, method = "ellipse") # Figura de correlaciones con elipses
# Elipses con colores
corrplot(M, method = "circle") # Figura de correlaciones con circulos
par(mfrow = c(1,1))OPCIONES DE ELIPSES
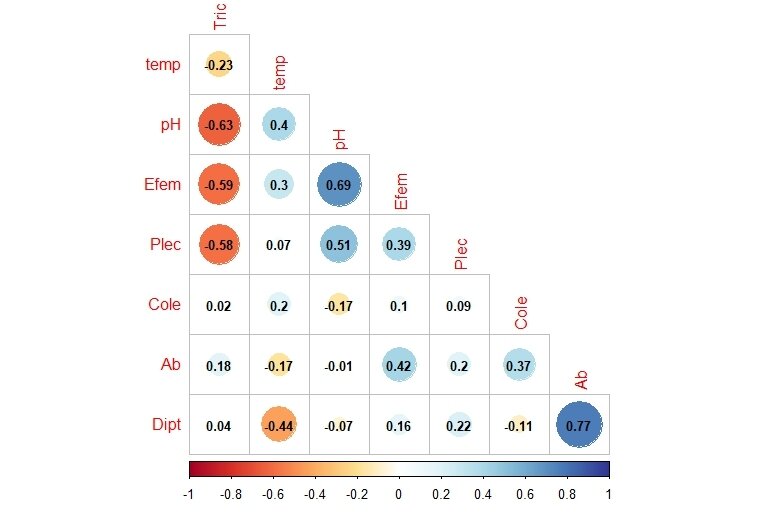
# Figura de elipses con coeficientes de correlación
corrplot(M, method = "circle", # Correlaciones con circulos
type = "lower", insig="blank", # Forma del panel
order = "AOE", diag = FALSE, # Ordenar por nivel de correlación
addCoef.col ="black", # Color de los coeficientes
number.cex = 0.8, # Tamaño del texto
col = COL2("RdYlBu", 200)) # Transparencia de los circulos# Elipses con colores
M1 <- cor(datos[,3:4], datos[,5:10]) # Matriz de Correlación (M)
# Elipses con colores
X11(6,8)
corrplot(M1, method = "ellipse", type="upper")2. DISPERSIÓN
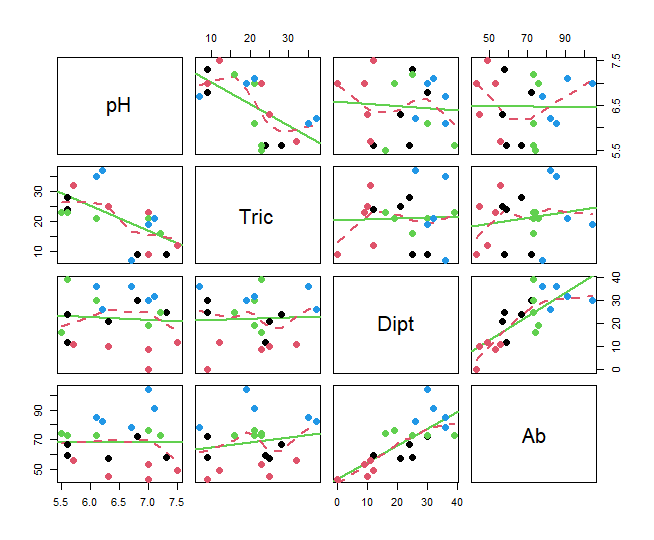
# 4. Figuras de pares
# **Ejercicio**: incluir las variables 2 a la 8
pairs (( ... ),panel=function(x,y)
{abline(lsfit(x,y)$coef,lwd=2,col=3) # lwd = Ancho de la línea
lines(lowess(x,y),lty=1,lwd=3,col=2) # col= Color de la línea
points(x,y,cex=1)}) # Incluir el factor (cuenca)
# ***Ejercicio***: Incluir las variables 2,6,7,9. Graficar la **cuenca**
pairs (( ... ),panel=function(x,y)
{abline(lsfit(x,y)$coef,lwd=2,col=3)
lines(lowess(x,y),lty=2,lwd=2,col=2)
points(x,y,col= ... , cex=1.4,pch=19)})
# Correlaciones de Pearson
x11()
pairs(datos[,c(3,5,7,8,10)], diag.panel = panel.hist,
upper.panel = panel.cor, lower.panel = panel.smooth)# Figura de pares con coeficientes de correlación y regresiones
ggpairs(datos, columns = c(3,7,8,10),
aes(color=cuenca, alpha = 0.5),
lower=list(continuous="smooth"))3. HISTOGRAMAS
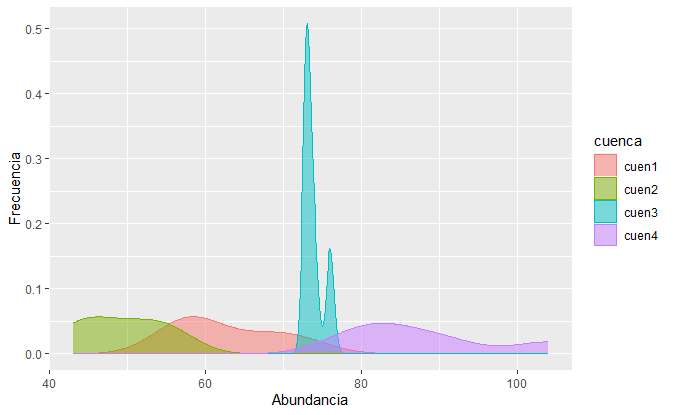
# Frecuencias de abundancias
# ***Ejercicio***= Rotular el eje Y con "Porcentaje del Total",
# El eje X con "Abundancia de insectos"
histogram (~Ab,data=datos, ...)# Frecuencias de abundancias por cuencas
histogram (~Ab|cuenca,data=datos, ylab="Porcentaje del Total",
xlab="Abundancia de insectos")# Frecuencias por densidad
densityplot(~Ab,data=datos, ylab="Porcentaje del Total",
xlab="Abundancia de insectos")
densityplot(~Ab|cuenca,data=datos, ylab="Porcentaje del Total",
xlab="Abundancia de insectos")# Frecuencias de abundancias por densidad
ggplot(data = datos, aes(x = Ab, color = cuenca)) +
geom_density(aes(fill = cuenca), alpha = 0.5) +
labs( y="Frecuencia", x="Abundancia")# Otra opción
ggplot(data = datos, aes(x = Ab, color = cuenca)) +
geom_density(aes(fill = cuenca)) +
facet_wrap(~ cuenca)4. DISPERSIÓN X-Y
# Regresión lineal (esquema básico)
plot(Efem~Plec,col=as.integer(cuenca),data=datos)
legend(0,25,legend=levels(datos$cuenca),pch=19,col=1:4,cex=0.8)
lines(abline(lm(datos$Efem~datos$Plec),lwd=2,col=2, lty=2))# Regresiones lineales (Esquema ggplot2)
# ***Ejercicio*** Graficar la relación entre el pH y los efemerópteros
ggplot(datos, ... ) +
geom_point(aes(color = cuenca), size = 3) +
geom_smooth(method= "lm")# Regresiones suavizadas - Loess o Lowess (Esquema ggplot2)
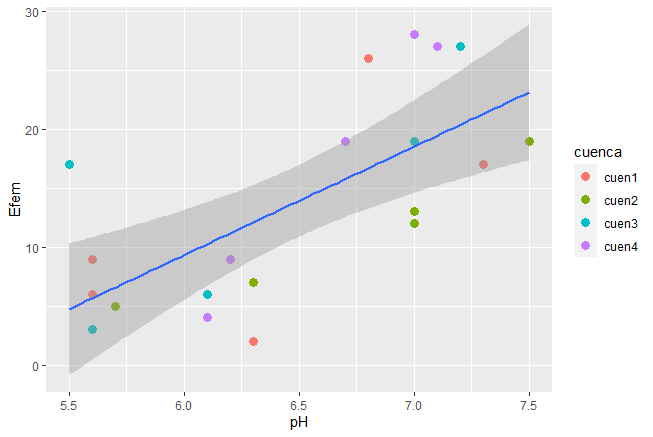
ggplot(datos,aes(x = pH, y = Efem)) +
geom_point(aes(color = cuenca), size = 3) +
geom_smooth()# Regresiones suavizadas (Loess)
ggplot(datos,aes(x = pH, y = Efem)) +
geom_point(aes(color = cuenca), size = 3) +
geom_smooth(se = FALSE, span = 0.4)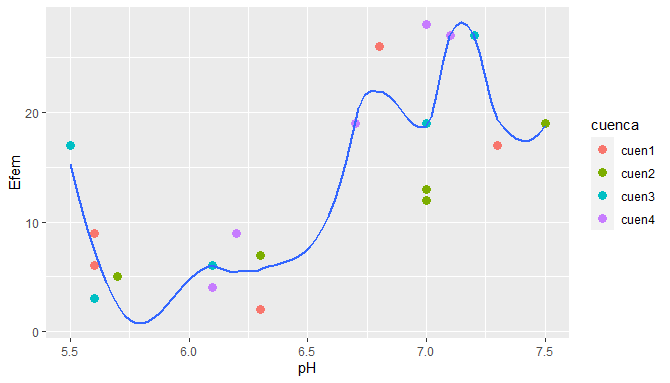
CAJAS Y BIGOTES
# Figuras de Cajas y bigotes
datos$cuenca<-factor(datos$cuenca,
levels=c("cuen1","cuen2","cuen3","cuen4"))# Cajas y Bigotes con muescas
# *** Ejercicio*** Graficar La abundancia (Ab) por tipos de cuencas.
boxplot( ... ~ ..., ...,
xlab="Cuencas",ylab="Abundancia",
col="lightgray", cex.lab=1.3)Cajas con la biblioteca "lattice"
# Buscar en google: colorbrewer2
# Resultado: ColorBrewer: Color Advice for Maps
... +
geom_boxplot(aes(fill = cuenca)) +
scale_fill_manual(values = c('#fc8d59','#ffffbf',
'#99d594','#99d594'))Cajas con la biblioteca "ggplot2"
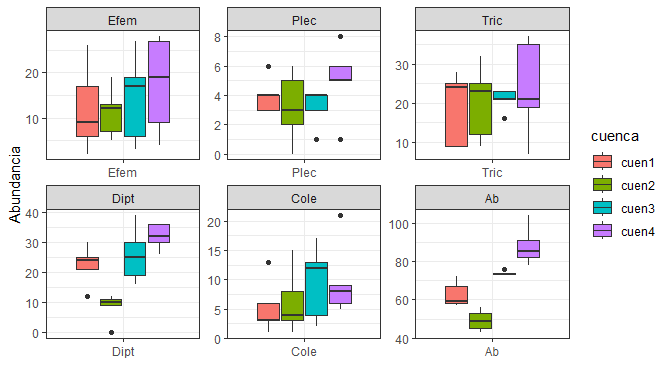
# Figuras de Cajas y bigotes (ggplot2)
ggplot(melt(datos[,c(2,5:10)]), aes(x=variable, y=value)) +
geom_boxplot(aes(fill=cuenca)) +
labs(x="",y="Abundancia") +
facet_wrap(~ variable,scales="free") +
theme_bw()Cajas con la biblioteca "ggplot2"
# violin: como histograma acostado
ggplot(iris, aes(x = Species, y = Sepal.Length)) +
geom_violin(aes(fill = Species)) +
geom_jitter() +
scale_fill_manual(values = c('#fc8d59','#ffffbf','#99d594')) +
theme_bw()7. FIGURAS CON ESTADÍSTICOS
# Resumen estadístico "datos_resum"
datos_resum <- datos %>% # Base de datos resumida
group_by(cuenca) %>% # Factor o variable agrupadora
summarise(datos.m = mean(Ab), # Media de cada grupo del factor
datos.de = sd(Ab), # Desviacioes est?ndar de cada grupo
datos.var = var(Ab), # Varianzas de cada grupo
n.Ab = n(), # Tamaño de cada grupo
datos.ee = sd(Ab)/sqrt(n())) # Error est?ndar de cada grupo
datos_resum# Figura de promedios y errores estándar
DatosPlot<-
ggplot(datos_resum, aes(cuenca, datos.m, dev.off())) +
geom_bar(stat="identity", col="black", fill="#377eb8") +
geom_errorbar(aes(ymin=datos.m-datos.ee,
ymax=datos.m+datos.ee),width=0.2) # Imprimir la figura
print(DatosPlot + labs(y="Abundancia ± e.e.", x = "cuenca") +
theme_classic())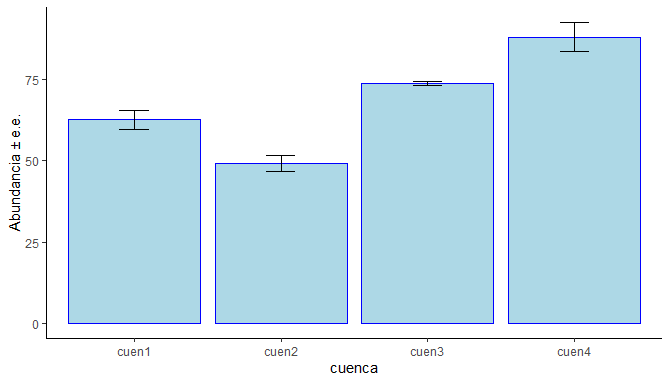
# Base de datos multifactorial (insectos1)
datos1<-read_csv2("Insectos2.csv") # Formato *Tidyr
head(datos1) # Encabezado de la base "datos1"
str(datos1) # estructura de la base "datos1"# Resumen estadístico "datos_resum"
datos_resum <- datos1 %>% # Base de datos resumida
group_by(Lluvia,GF) %>% # Factor o variable agrupadora
summarise(datos.m = mean(Biom), # Media de cada grupo del factor
datos.de = sd(Biom), # Desviacioes estándar de cada grupo
datos.var = var(Biom), # Varianzas de cada grupo
n.Biom = n(), # Tamaño de cada grupo
datos.ee = sd(Biom)/sqrt(n())) # Error estándar de cada grupo
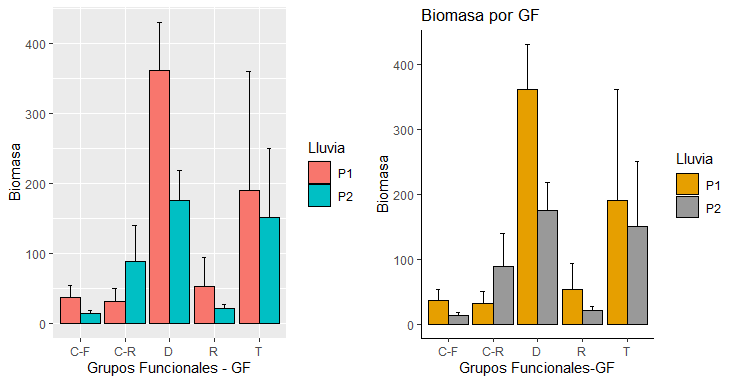
datos_resum# Figura 1 (f1)
f1 = ggplot(datos_resum, aes(x=GF, y=datos.m, fill=Lluvia)) +
geom_bar(stat="identity", color="black",
position=position_dodge()) +
geom_errorbar(aes(ymin=datos.m, ymax=datos.m+datos.de), width=.2,
position=position_dodge(.9))# Impresión de un panel con las dos figuras (f1 y f2)
grid.arrange (p1, p2, ncol=2)# f2: Otro formato de figura bifactorial - theme_classic
f2 = f1+labs(title="Biomasa por GF",
x="Grupos Funcionales - GF",
y = "Biomasa")+
theme_classic() +
scale_fill_manual(values=c('#E69F00','#999999'))# Impresión de un panel con las dos figuras (f1 y f2)
grid.arrange (p1, p2, nrow=2)OTRAS OPCIONES
Para la casa
6. COPLOT
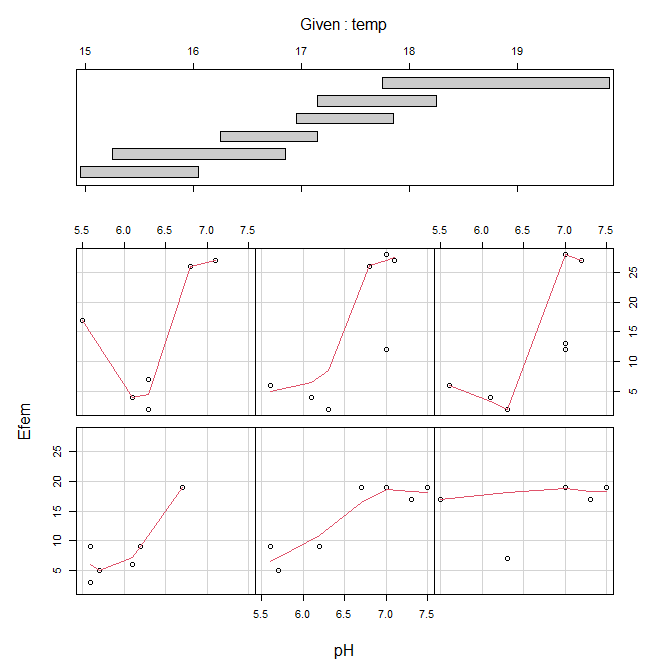
# Coplot con líneas de ajuste suavizado (loess)
with(datos, {
coplot(Efem~pH|temp,
panel = panel.smooth)})# Función para el coplot
panel.lm = function(x, y, ...) {
tmp<-lm(y~x,na.action=na.omit)
abline(tmp, lwd = 1.5, col= 2)
points(x,y, ...)}
# Cotplot con variables categorizadas
summary(datos[,3:9])
clasetemp <- cut(datos$temp,seq(15,20,1.2),include.lowest=T,
labels = c("t.baja", "t.media1","t.media2", "t.alta"))
clasepH <- cut(datos$pH,seq(5,8,1),include.lowest=T,
labels = c("pH.bajo", "pH.medio","pH.alto"))# Relación trivariada - Lineal
coplot(Efem~pH | clasetemp, pch=19,
panel = panel.lm, data=datos)# Relación trivariada - Suavizada
with(datos, {
coplot(Efem~pH|clasetemp,
panel = panel.smooth)})# Función para el coplot
panel.lm = function(x, y, ...) {
tmp<-lm(y~x,na.action=na.omit)
abline(tmp, lwd = 1.5, col= 2)
points(x,y, ...)}4. FIGURAS ANIMADAS
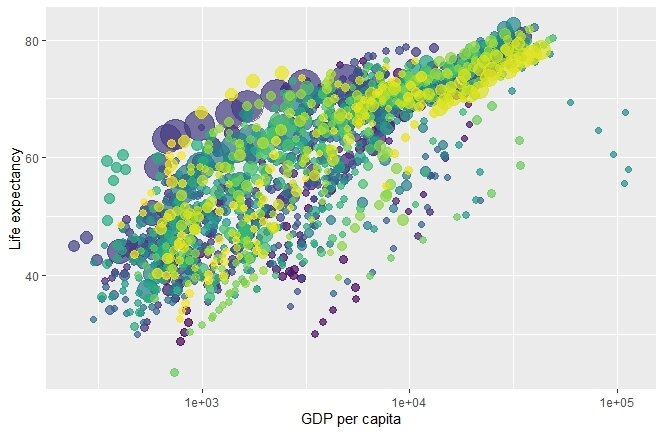
library(ggplot2)
library(gganimate)
theme_set(theme_bw()) # Tema o fondo de la figura por default
library(gapminder)# Edición de la figura
p <- ggplot(
gapminder,
aes(x = gdpPercap, y=lifeExp, size = pop, colour = country)) +
geom_point(show.legend = FALSE, alpha = 0.7) +
scale_color_viridis_d() +
scale_size(range = c(2, 12)) +
scale_x_log10() +
labs(x = "GDP per capita", y = "Life expectancy")# Figura estática
p
# Figura animada
p + transition_time(year) +
labs(title = "Year: {frame_time}")TALLER DE ENTRENAMIENTO
Objetivo 1: Poner en práctica los conceptos vistos en el módulo de exploratorios multivariados, realizando las siguientes opciones gráficas en las bases de datos asignadas para los estdios de caso:
-
Figuras de elipses
-
Figuras de Dispersión por pares de variables (pairs)
-
Histogramas
-
Dispersión X-Y
-
Cajas y Bigotes
-
Coplot
-
Figuras con estadísticos (promedios, errores, …)
TALLER DE ENTRENAMIENTO
Objetivo 2: Poner en práctica lo anterior con la base de datos de lirios `"data(iris)"`
-
Figuras de elipses
-
Figuras de Dispersión por pares de variables (pairs)
-
Histogramas
-
Dispersión X-Y
-
Cajas y Bigotes
-
Coplot
-
Figuras con estadísticos (promedios, errores, …)
Exploratorios Visualización (1)
By Javier Rodriguez
Exploratorios Visualización (1)
- 27



